+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6tko | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Phosphorylated turkey beta1 adrenoceptor with bound agonist formoterol coupled to arrestin-2 in lipid nanodisc. | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | SIGNALING PROTEIN / GPCR / Arrestin / Complex / Nanodisc | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報beta1-adrenergic receptor activity / positive regulation of heart contraction / angiotensin receptor binding / regulation of circadian sleep/wake cycle, sleep / TGFBR3 regulates TGF-beta signaling / Activation of SMO / negative regulation of interleukin-8 production / G protein-coupled receptor internalization / arrestin family protein binding / norepinephrine-epinephrine-mediated vasodilation involved in regulation of systemic arterial blood pressure ...beta1-adrenergic receptor activity / positive regulation of heart contraction / angiotensin receptor binding / regulation of circadian sleep/wake cycle, sleep / TGFBR3 regulates TGF-beta signaling / Activation of SMO / negative regulation of interleukin-8 production / G protein-coupled receptor internalization / arrestin family protein binding / norepinephrine-epinephrine-mediated vasodilation involved in regulation of systemic arterial blood pressure / Lysosome Vesicle Biogenesis / negative regulation of NF-kappaB transcription factor activity / positive regulation of cardiac muscle hypertrophy / Golgi Associated Vesicle Biogenesis / stress fiber assembly / positive regulation of Rho protein signal transduction / pseudopodium / negative regulation of interleukin-6 production / positive regulation of receptor internalization / negative regulation of Notch signaling pathway / enzyme inhibitor activity / adenylate cyclase-activating adrenergic receptor signaling pathway / insulin-like growth factor receptor binding / clathrin-coated pit / negative regulation of protein ubiquitination / GTPase activator activity / Activated NOTCH1 Transmits Signal to the Nucleus / cytoplasmic vesicle membrane / G protein-coupled receptor binding / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / positive regulation of protein phosphorylation / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / endocytic vesicle membrane / Signaling by BRAF and RAF1 fusions / Cargo recognition for clathrin-mediated endocytosis / protein transport / Thrombin signalling through proteinase activated receptors (PARs) / Clathrin-mediated endocytosis / cytoplasmic vesicle / ubiquitin-dependent protein catabolic process / G alpha (s) signalling events / molecular adaptor activity / proteasome-mediated ubiquitin-dependent protein catabolic process / transcription coactivator activity / early endosome / positive regulation of ERK1 and ERK2 cascade / positive regulation of MAPK cascade / Ub-specific processing proteases / protein ubiquitination / nuclear body / Golgi membrane / lysosomal membrane / ubiquitin protein ligase binding / regulation of transcription by RNA polymerase II / chromatin / signal transduction / positive regulation of transcription by RNA polymerase II / nucleoplasm / identical protein binding / nucleus / membrane / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)Phage display vector pTDisp (その他) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Lee, Y. / Tate, C.G. | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Molecular basis of β-arrestin coupling to formoterol-bound β-adrenoceptor. 著者: Yang Lee / Tony Warne / Rony Nehmé / Shubhi Pandey / Hemlata Dwivedi-Agnihotri / Madhu Chaturvedi / Patricia C Edwards / Javier García-Nafría / Andrew G W Leslie / Arun K Shukla / Christopher G Tate /     要旨: The β-adrenoceptor (βAR) is a G-protein-coupled receptor (GPCR) that couples to the heterotrimeric G protein G. G-protein-mediated signalling is terminated by phosphorylation of the C terminus of ...The β-adrenoceptor (βAR) is a G-protein-coupled receptor (GPCR) that couples to the heterotrimeric G protein G. G-protein-mediated signalling is terminated by phosphorylation of the C terminus of the receptor by GPCR kinases (GRKs) and by coupling of β-arrestin 1 (βarr1, also known as arrestin 2), which displaces G and induces signalling through the MAP kinase pathway. The ability of synthetic agonists to induce signalling preferentially through either G proteins or arrestins-known as biased agonism-is important in drug development, because the therapeutic effect may arise from only one signalling cascade, whereas the other pathway may mediate undesirable side effects. To understand the molecular basis for arrestin coupling, here we determined the cryo-electron microscopy structure of the βAR-βarr1 complex in lipid nanodiscs bound to the biased agonist formoterol, and the crystal structure of formoterol-bound βAR coupled to the G-protein-mimetic nanobody Nb80. βarr1 couples to βAR in a manner distinct to that of G coupling to βAR-the finger loop of βarr1 occupies a narrower cleft on the intracellular surface, and is closer to transmembrane helix H7 of the receptor when compared with the C-terminal α5 helix of G. The conformation of the finger loop in βarr1 is different from that adopted by the finger loop of visual arrestin when it couples to rhodopsin. βAR coupled to βarr1 shows considerable differences in structure compared with βAR coupled to Nb80, including an inward movement of extracellular loop 3 and the cytoplasmic ends of H5 and H6. We observe weakened interactions between formoterol and two serine residues in H5 at the orthosteric binding site of βAR, and find that formoterol has a lower affinity for the βAR-βarr1 complex than for the βAR-G complex. The structural differences between these complexes of βAR provide a foundation for the design of small molecules that could bias signalling in the β-adrenoceptors. #1:  ジャーナル: Biorxiv / 年: 2020 ジャーナル: Biorxiv / 年: 2020タイトル: Molecular determinants of beta-arrestin coupling to formoterol-bound beta1-adrenoceptor. 著者: Lee, Y. / Warne, T. / Nehme, R. / Pandey, S. / Dwivedi-Agnihotri, H. / Edwards, P.C. / Garcia-Nafria, J. / Leslie, A.G.W. / Shukla, A.K. / Tate, C.G. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6tko.cif.gz 6tko.cif.gz | 205.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6tko.ent.gz pdb6tko.ent.gz | 152.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6tko.json.gz 6tko.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6tko_validation.pdf.gz 6tko_validation.pdf.gz | 1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6tko_full_validation.pdf.gz 6tko_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  6tko_validation.xml.gz 6tko_validation.xml.gz | 40.2 KB | 表示 | |
| CIF形式データ |  6tko_validation.cif.gz 6tko_validation.cif.gz | 62.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/tk/6tko https://data.pdbj.org/pub/pdb/validation_reports/tk/6tko ftp://data.pdbj.org/pub/pdb/validation_reports/tk/6tko ftp://data.pdbj.org/pub/pdb/validation_reports/tk/6tko | HTTPS FTP |
-関連構造データ
| 関連構造データ |  10515MC  6iblC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10342 (タイトル: Cryo-EM structure of the formoterol-bound turkey beta1 adrenoceptor coupled to beta-arrestin-1 with bound Fab30 in lipid nanodisc EMPIAR-10342 (タイトル: Cryo-EM structure of the formoterol-bound turkey beta1 adrenoceptor coupled to beta-arrestin-1 with bound Fab30 in lipid nanodiscData size: 4.0 TB Data #1: Unaligned multi-frame micrographs [micrographs - multiframe] Data #2: Unaligned multi-frame micrographs [micrographs - multiframe] Data #3: Unaligned multi-frame micrographs [micrographs - multiframe] Data #4: Nanodisc subtracted particles [picked particles - multiframe - processed] Data #5: Nanodisc-reinstated particles [picked particles - multiframe - unprocessed]) |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 37275.395 Da / 分子数: 1 変異: S32G M44C M90V V103C C116L E130W D322K F327A F338M C358A 由来タイプ: 組換発現 詳細: Relative to wild-type sequence: construct is truncated at the N-terminus; and in ICL3; its C-terminus is truncated at C358A and fused with a linker to a series of phosphorylated residues ...詳細: Relative to wild-type sequence: construct is truncated at the N-terminus; and in ICL3; its C-terminus is truncated at C358A and fused with a linker to a series of phosphorylated residues (ARGRPLPETGGGDE[pS]A[pT][pT]A[pS][pS][pS]LAKDTSS). 由来: (組換発現)  遺伝子: ADRB1 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P07700 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P07700 |
|---|---|
| #2: タンパク質 | 分子量: 47021.332 Da / 分子数: 1 / 変異: M1G L68C R169E / 由来タイプ: 組換発現 詳細: Additional N-terminal Gly residue, in place of starting Met, remaining from proteolytic cleavage. 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: ARRB1, ARR1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: ARRB1, ARR1 / 発現宿主:  |
| #3: 抗体 | 分子量: 25512.354 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現) Phage display vector pTDisp (その他) 発現宿主:  |
| #4: 抗体 | 分子量: 23435.064 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現) Phage display vector pTDisp (その他) 発現宿主:  |
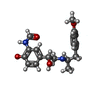
| #5: 化合物 | ChemComp-H98 / ~{ |
| 研究の焦点であるリガンドがあるか | Y |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.133 MDa / 実験値: NO | ||||||||||||||||||||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 | ||||||||||||||||||||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||||||||||||||||||||
| 試料 | 濃度: 1 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES / 詳細: This sample was monodisperse. | ||||||||||||||||||||||||||||||||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 | ||||||||||||||||||||||||||||||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277.15 K 詳細: Blotted for 2-3 seconds before plunging. Liquid ethane maintained at 92.15 K. |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EM imaging | 加速電圧: 300 kV / アライメント法: COMA FREE / C2レンズ絞り径: 100 µm / 凍結剤: NITROGEN / 電子線源:
| |||||||||||||||
| 撮影 |
| |||||||||||||||
| 電子光学装置 |
|
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 10161197 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 403991 詳細: Half maps were locally filtered between refinement iterations using SIDESPLITTER, an adaptation of the LAFTER algorithm that maintains gold-standard separation between the two half maps. 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 80.6 / プロトコル: OTHER / 空間: REAL / Target criteria: Correlation coefficient 詳細: Initial placement was done manually in Coot, followed by rigid-body refinement in PHENIX and jelly-body refinement in REFMAC5. Manual remodeling was performed in Coot and iterated with real ...詳細: Initial placement was done manually in Coot, followed by rigid-body refinement in PHENIX and jelly-body refinement in REFMAC5. Manual remodeling was performed in Coot and iterated with real space refinement in PHENIX. Chemical restraints for formoterol were calculated in eLBOW using AM1 optimisation. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | 3D fitting-ID: 1 / Source name: PDB / タイプ: experimental model
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 80.62 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera






 PDBj
PDBj