| 登録情報 | データベース: PDB / ID: 6acr
|
|---|
| タイトル | Crystal structure of human ALK2 kinase domain with R206H mutation in complex with RK-59638 |
|---|
 要素 要素 | Activin receptor type-1 |
|---|
 キーワード キーワード | SIGNALING PROTEIN / Inhibitor / Complex / Kinase |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
endocardial cushion cell fate commitment / mitral valve morphogenesis / BMP receptor complex / cardiac muscle cell fate commitment / BMP receptor activity / atrial septum primum morphogenesis / endocardial cushion fusion / positive regulation of cardiac epithelial to mesenchymal transition / acute inflammatory response / positive regulation of determination of dorsal identity ...endocardial cushion cell fate commitment / mitral valve morphogenesis / BMP receptor complex / cardiac muscle cell fate commitment / BMP receptor activity / atrial septum primum morphogenesis / endocardial cushion fusion / positive regulation of cardiac epithelial to mesenchymal transition / acute inflammatory response / positive regulation of determination of dorsal identity / transforming growth factor beta receptor activity, type I / smooth muscle cell differentiation / activin receptor complex / activin receptor activity, type I / endocardial cushion formation / pharyngeal system development / transmembrane receptor protein serine/threonine kinase activity / receptor protein serine/threonine kinase / activin binding / cellular response to BMP stimulus / activin receptor signaling pathway / negative regulation of activin receptor signaling pathway / embryonic heart tube morphogenesis / gastrulation with mouth forming second / dorsal/ventral pattern formation / transforming growth factor beta binding / determination of left/right symmetry / atrioventricular valve morphogenesis / neural crest cell migration / branching involved in blood vessel morphogenesis / ventricular septum morphogenesis / negative regulation of G1/S transition of mitotic cell cycle / SMAD binding / germ cell development / peptide hormone binding / positive regulation of intracellular signal transduction / mesoderm formation / positive regulation of SMAD protein signal transduction / regulation of ossification / positive regulation of osteoblast differentiation / positive regulation of bone mineralization / BMP signaling pathway / negative regulation of signal transduction / transforming growth factor beta receptor signaling pathway / protein tyrosine kinase binding / negative regulation of extrinsic apoptotic signaling pathway / cellular response to growth factor stimulus / apical part of cell / osteoblast differentiation / heart development / in utero embryonic development / cell differentiation / protein kinase activity / positive regulation of cell migration / cadherin binding / protein serine/threonine kinase activity / positive regulation of DNA-templated transcription / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / ATP binding / metal ion binding / plasma membrane類似検索 - 分子機能 GS domain / Transforming growth factor beta type I GS-motif / GS domain profile. / GS motif / Activin types I and II receptor domain / Activin types I and II receptor domain / Ser/Thr protein kinase, TGFB receptor / Snake toxin-like superfamily / Protein tyrosine and serine/threonine kinase / Phosphorylase Kinase; domain 1 ...GS domain / Transforming growth factor beta type I GS-motif / GS domain profile. / GS motif / Activin types I and II receptor domain / Activin types I and II receptor domain / Ser/Thr protein kinase, TGFB receptor / Snake toxin-like superfamily / Protein tyrosine and serine/threonine kinase / Phosphorylase Kinase; domain 1 / Phosphorylase Kinase; domain 1 / Transferase(Phosphotransferase) domain 1 / Transferase(Phosphotransferase); domain 1 / Serine/threonine-protein kinase, active site / Serine/Threonine protein kinases active-site signature. / Protein kinase domain / Serine/Threonine protein kinases, catalytic domain / Protein kinase, ATP binding site / Protein kinases ATP-binding region signature. / Protein kinase domain profile. / Protein kinase domain / Protein kinase-like domain superfamily / 2-Layer Sandwich / Orthogonal Bundle / Mainly Alpha / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.01 Å 分子置換 / 解像度: 2.01 Å |
|---|
 データ登録者 データ登録者 | Sakai, N. / Mishima-Tsumagari, C. / Matsumoto, T. / Shirouzu, M. |
|---|
 引用 引用 |  ジャーナル: Chem. Pharm. Bull. / 年: 2019 ジャーナル: Chem. Pharm. Bull. / 年: 2019
タイトル: Bis-Heteroaryl Pyrazoles: Identification of Orally Bioavailable Inhibitors of Activin Receptor-Like Kinase-2 (R206H).
著者: Sekimata, K. / Sato, T. / Sakai, N. / Watanabe, H. / Mishima-Tsumagari, C. / Taguri, T. / Matsumoto, T. / Fujii, Y. / Handa, N. / Honma, T. / Tanaka, A. / Shirouzu, M. / Yokoyama, S. / ...著者: Sekimata, K. / Sato, T. / Sakai, N. / Watanabe, H. / Mishima-Tsumagari, C. / Taguri, T. / Matsumoto, T. / Fujii, Y. / Handa, N. / Honma, T. / Tanaka, A. / Shirouzu, M. / Yokoyama, S. / Miyazono, K. / Hashizume, Y. / Koyama, H. |
|---|
| 履歴 | | 登録 | 2018年7月27日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2019年3月20日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年11月22日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.01 Å
分子置換 / 解像度: 2.01 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Chem. Pharm. Bull. / 年: 2019
ジャーナル: Chem. Pharm. Bull. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6acr.cif.gz
6acr.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6acr.ent.gz
pdb6acr.ent.gz PDB形式
PDB形式 6acr.json.gz
6acr.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6acr_validation.pdf.gz
6acr_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6acr_full_validation.pdf.gz
6acr_full_validation.pdf.gz 6acr_validation.xml.gz
6acr_validation.xml.gz 6acr_validation.cif.gz
6acr_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ac/6acr
https://data.pdbj.org/pub/pdb/validation_reports/ac/6acr ftp://data.pdbj.org/pub/pdb/validation_reports/ac/6acr
ftp://data.pdbj.org/pub/pdb/validation_reports/ac/6acr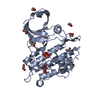
 リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: ACVR1, ACVRLK2 / 細胞株 (発現宿主): SF9
Homo sapiens (ヒト) / 遺伝子: ACVR1, ACVRLK2 / 細胞株 (発現宿主): SF9
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Photon Factory
Photon Factory  / ビームライン: BL-17A / 波長: 0.98 Å
/ ビームライン: BL-17A / 波長: 0.98 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj