ソフトウェア 名称 バージョン 分類 dev_2650精密化 データスケーリング 3.22 データ抽出 データ削減 位相決定
精密化 構造決定の手法 開始モデル 解像度 / 交差検証法 / σ(F) / 位相誤差 / 立体化学のターゲット値 Rfactor 反射数 %反射 Rfree 0.2231 494 5.33 % Rwork 0.1721 8741 - obs 0.1761 9273 93.62 %
溶媒の処理 減衰半径 / VDWプローブ半径 / 溶媒モデル 原子変位パラメータ Biso max 2 / Biso mean 2 / Biso min 2 精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 1357 0 0 59 1416 Biso mean - - - 39.79 - 残基数 - - - - 176
拘束条件 Refine-ID タイプ Dev ideal 数 X-RAY DIFFRACTION f_bond_d0.002 1393 X-RAY DIFFRACTION f_angle_d0.469 1892 X-RAY DIFFRACTION f_chiral_restr0.042 202 X-RAY DIFFRACTION f_plane_restr0.004 251 X-RAY DIFFRACTION f_dihedral_angle_d6.694 810
LS精密化 シェル Refine-ID / Rfactor Rfree error / Total num. of bins used
解像度 (Å)Rfactor Rfree Num. reflection Rfree Rfactor Rwork Num. reflection Rwork Num. reflection all % reflection obs (%)2.2053-2.4268 0.2544 98 0.2004 1749 1847 72 2.4268-2.7767 0.3176 118 0.1837 2344 2462 95 2.7767-3.494 0.2322 123 0.178 2348 2471 95 3.494-17.7069 0.1885 151 0.1586 2300 2451 94
精密化 TLS 手法 / Refine-ID
大きな表を表示 (25 x 12) 大きな表を隠す ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)S33 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 0.006 -0.0014 -0.0061 0.0034 0.0037 0.011 -0.0081 -0.0021 0.0015 -0.0076 -0.0113 0.0129 -0.0098 0.0013 -0.007 0.0301 -0.0755 -0.0024 0.1311 -0.0346 0.1068 -2.8887 12.5443 4.044 2 0.0093 0.0027 -0.0003 0.0046 0.0024 0.0018 -0.0024 0.0031 -0.0016 -0.0029 -0.0037 0.001 -0.0004 -0.0021 0 0.312 -0.035 0.0221 0.3938 0.0033 0.3644 -20.9271 9.7786 -4.5617 3 0.0007 0.0007 0.0007 0.0014 0.0013 0.001 0.0043 0.0051 -0.0079 -0.0112 0.0051 0.0094 0.0154 -0.0204 0 0.1534 -0.1237 0.0031 0.2699 -0.0115 0.1813 -9.9982 3.0523 -7.9754 4 0.001 -0.0001 -0.0007 -0.0001 0.0006 0.0008 0.0118 0.0144 -0.01 -0.008 0.0167 -0.01 0.0352 0.0184 -0 0.0669 -0.0116 0.0163 0.1942 -0.031 0.1633 0.9586 2.9885 -0.939 5 0.0026 -0.001 0.0002 0.0011 0.0008 0.0013 -0.0057 -0.0054 0.0029 -0.0028 0.0038 0.0004 -0.0026 -0.0137 0 0.1597 -0.0309 -0.0259 0.2423 0.0201 0.2418 -18.4662 8.3594 6.9712 6 0.0109 0.0125 0.0093 0.0198 0.0174 0.0268 0.0332 0.0003 0.017 -0.0154 0.0011 -0.0021 -0.0089 0.0251 0.0458 0.0422 -0.078 0.0509 0.1858 0.004 0.1749 1.3813 11.412 -1.9925 7 0.0027 0.002 0.0003 0.0037 0.004 0.0098 0.0059 0.0074 0.01 0.005 0.0073 0.0091 -0.0026 0.0103 0.0155 0.0548 -0.1187 -0.0338 0.087 0.0349 0.1995 0.4004 11.3894 10.5522 8 0.0041 0.0008 -0.0026 0.0047 -0.005 0.0057 -0.0046 0.0126 -0.0016 0.0016 0.0049 0.0013 -0.0001 -0.0122 0.0001 0.1615 -0.164 -0.0153 0.0913 0.0088 0.1382 -9.1018 8.5067 15.0826 9 0.0005 0.0001 0.0017 0.0018 -0.0018 0.0033 -0.006 -0.0186 -0.0014 0.0031 0.0097 0.0092 0.0241 0.0216 -0 0.2152 -0.0961 -0.0478 0.1547 0.0415 0.1164 -0.142 7.6869 25.3526 10 -0.0004 0.0003 0.0007 0.0014 -0.0003 0.0011 -0.0017 -0.0104 -0.0279 -0.0072 -0.0078 0.0033 0.023 -0.0026 0 0.1038 -0.0328 0.0273 0.0488 -0.0167 0.0991 -3.4779 0.1194 17.346 11 0.0007 0.0013 -0.0021 0.0003 0.0012 0.0054 0.0019 0.0058 0.0098 0.0072 -0.0036 -0.002 0.0125 -0.0198 -0.013 0.1009 -0.1689 0.029 0.0963 0.0545 0.1483 -9.2775 5.8762 17.7117 12 0.0015 0.0005 0.0019 0.0018 0.0004 0.0023 -0.011 0.0035 -0.0179 0.0043 0.0349 -0.0042 0.0031 -0.0274 0 0.1177 -0.0764 0.0321 0.1692 -0.0345 0.218 -10.5784 3.72 7.5905
精密化 TLSグループ ID Refine-ID Refine TLS-ID Selection details Auth asym-ID Auth seq-ID 1 X-RAY DIFFRACTION 1 chain 'A' and (resid 1 through 10 )A1 - 10 2 X-RAY DIFFRACTION 2 chain 'A' and (resid 11 through 15 )A11 - 15 3 X-RAY DIFFRACTION 3 chain 'A' and (resid 16 through 28 )A16 - 28 4 X-RAY DIFFRACTION 4 chain 'A' and (resid 29 through 42 )A29 - 42 5 X-RAY DIFFRACTION 5 chain 'A' and (resid 43 through 49 )A43 - 49 6 X-RAY DIFFRACTION 6 chain 'A' and (resid 50 through 87 )A50 - 87 7 X-RAY DIFFRACTION 7 chain 'A' and (resid 88 through 99 )A88 - 99 8 X-RAY DIFFRACTION 8 chain 'B' and (resid -1 through 15 ) B-1 - 15 9 X-RAY DIFFRACTION 9 chain 'B' and (resid 16 through 31 )B16 - 31 10 X-RAY DIFFRACTION 10 chain 'B' and (resid 32 through 42 )B32 - 42 11 X-RAY DIFFRACTION 11 chain 'B' and (resid 43 through 86 )B43 - 86 12 X-RAY DIFFRACTION 12 chain 'B' and (resid 87 through 100 )B87 - 100
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.201 Å
分子置換 / 解像度: 2.201 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5uq4.cif.gz
5uq4.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5uq4.ent.gz
pdb5uq4.ent.gz PDB形式
PDB形式 5uq4.json.gz
5uq4.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5uq4_validation.pdf.gz
5uq4_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5uq4_full_validation.pdf.gz
5uq4_full_validation.pdf.gz 5uq4_validation.xml.gz
5uq4_validation.xml.gz 5uq4_validation.cif.gz
5uq4_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/uq/5uq4
https://data.pdbj.org/pub/pdb/validation_reports/uq/5uq4 ftp://data.pdbj.org/pub/pdb/validation_reports/uq/5uq4
ftp://data.pdbj.org/pub/pdb/validation_reports/uq/5uq4
 リンク
リンク 集合体
集合体
 要素
要素 Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌)
Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 19-ID / 波長: 0.97924 Å
/ ビームライン: 19-ID / 波長: 0.97924 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー










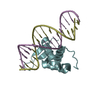
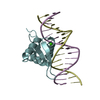

 PDBj
PDBj
