| 登録情報 | データベース: PDB / ID: 5tdc
|
|---|
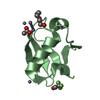
| タイトル | Crystal structure of the human UBR-box domain from UBR1 in complex with monomethylated arginine peptide. |
|---|
 要素 要素 | - E3 ubiquitin-protein ligase UBR1
- NMM-ILE-PHE-SER peptide
|
|---|
 キーワード キーワード | LIGASE / UBR-box / N-end rule / N-degron / monomethylated arginine |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
L-leucine binding / ubiquitin-dependent protein catabolic process via the N-end rule pathway / cellular response to L-leucine / negative regulation of TOR signaling / ubiquitin ligase complex / proteasome complex / RING-type E3 ubiquitin transferase / ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / proteasome-mediated ubiquitin-dependent protein catabolic process ...L-leucine binding / ubiquitin-dependent protein catabolic process via the N-end rule pathway / cellular response to L-leucine / negative regulation of TOR signaling / ubiquitin ligase complex / proteasome complex / RING-type E3 ubiquitin transferase / ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / proteasome-mediated ubiquitin-dependent protein catabolic process / protein ubiquitination / zinc ion binding / cytoplasm / cytosol類似検索 - 分子機能 : / E3 ubiquitin-protein ligase ELL-like / E3 ubiquitin-protein ligase UBR-like, C-terminal / : / Proteolysis_6 C-terminal / E3 ubiquitin-protein ligase UBR1-like, winged-helix domain / E3 ubiquitin-protein ligase UBR1-like / Adaptor protein ClpS, core / ATP-dependent Clp protease adaptor protein ClpS / Putative zinc finger in N-recognin (UBR box) ...: / E3 ubiquitin-protein ligase ELL-like / E3 ubiquitin-protein ligase UBR-like, C-terminal / : / Proteolysis_6 C-terminal / E3 ubiquitin-protein ligase UBR1-like, winged-helix domain / E3 ubiquitin-protein ligase UBR1-like / Adaptor protein ClpS, core / ATP-dependent Clp protease adaptor protein ClpS / Putative zinc finger in N-recognin (UBR box) / Zinc finger, UBR-type / Zinc finger UBR-type profile. / Putative zinc finger in N-recognin, a recognition component of the N-end rule pathway / Ribosomal protein L7/L12, C-terminal/adaptor protein ClpS-like / Winged helix DNA-binding domain superfamily類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.607 Å 分子置換 / 解像度: 1.607 Å |
|---|
 データ登録者 データ登録者 | Kozlov, G. / Munoz-Escobar, J. / Matta-Camacho, E. / Gehring, K. |
|---|
| 資金援助 |  カナダ, 1件 カナダ, 1件 | 組織 | 認可番号 | 国 |
|---|
| Canadian Institutes of Health Research (CIHR) | |  カナダ カナダ |
|
|---|
 引用 引用 |  ジャーナル: Structure / 年: 2017 ジャーナル: Structure / 年: 2017
タイトル: Bound Waters Mediate Binding of Diverse Substrates to a Ubiquitin Ligase.
著者: Munoz-Escobar, J. / Matta-Camacho, E. / Cho, C. / Kozlov, G. / Gehring, K. |
|---|
| 履歴 | | 登録 | 2016年9月19日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年3月22日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年4月26日 | Group: Database references |
|---|
| 改定 1.2 | 2017年5月10日 | Group: Database references |
|---|
| 改定 1.3 | 2017年9月20日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.4 | 2020年1月8日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.5 | 2023年10月4日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn / struct_conn_type
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_conn_type.id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.607 Å
分子置換 / 解像度: 1.607 Å  データ登録者
データ登録者 カナダ, 1件
カナダ, 1件  引用
引用 ジャーナル: Structure / 年: 2017
ジャーナル: Structure / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5tdc.cif.gz
5tdc.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5tdc.ent.gz
pdb5tdc.ent.gz PDB形式
PDB形式 5tdc.json.gz
5tdc.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5tdc_validation.pdf.gz
5tdc_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5tdc_full_validation.pdf.gz
5tdc_full_validation.pdf.gz 5tdc_validation.xml.gz
5tdc_validation.xml.gz 5tdc_validation.cif.gz
5tdc_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/td/5tdc
https://data.pdbj.org/pub/pdb/validation_reports/td/5tdc ftp://data.pdbj.org/pub/pdb/validation_reports/td/5tdc
ftp://data.pdbj.org/pub/pdb/validation_reports/td/5tdc リンク
リンク 集合体
集合体
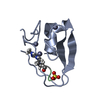

 要素
要素 Homo sapiens (ヒト) / 遺伝子: UBR1 / プラスミド: pGEX-6P-1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: UBR1 / プラスミド: pGEX-6P-1 / 発現宿主: 
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  CHESS
CHESS  / ビームライン: A1 / 波長: 0.9769 Å
/ ビームライン: A1 / 波長: 0.9769 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



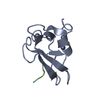

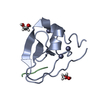




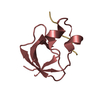






 PDBj
PDBj






