[English] 日本語
 Yorodumi
Yorodumi- PDB-5oni: LOW-SALT STRUCTURE OF PROTEIN KINASE CK2 CATALYTIC SUBUNIT (ISOFO... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5oni | ||||||
|---|---|---|---|---|---|---|---|
| Title | LOW-SALT STRUCTURE OF PROTEIN KINASE CK2 CATALYTIC SUBUNIT (ISOFORM CK2ALPHA) IN COMPLEX WITH THE INDENOINDOLE-TYPE INHIBITOR 4P | ||||||
 Components Components | Casein kinase II subunit alpha | ||||||
 Keywords Keywords | TRANSFERASE / protein kinase CK2 / casein kinase 2 / indenoindole-type inhibitors | ||||||
| Function / homology |  Function and homology information Function and homology informationPhosphorylation and nuclear translocation of the CRY:PER:kinase complex / Phosphorylation and nuclear translocation of BMAL1 (ARNTL) and CLOCK / Regulation of CDH1 posttranslational processing and trafficking to plasma membrane / positive regulation of aggrephagy / regulation of chromosome separation / WNT mediated activation of DVL / Condensation of Prometaphase Chromosomes / protein kinase CK2 complex / symbiont-mediated disruption of host cell PML body / Receptor Mediated Mitophagy ...Phosphorylation and nuclear translocation of the CRY:PER:kinase complex / Phosphorylation and nuclear translocation of BMAL1 (ARNTL) and CLOCK / Regulation of CDH1 posttranslational processing and trafficking to plasma membrane / positive regulation of aggrephagy / regulation of chromosome separation / WNT mediated activation of DVL / Condensation of Prometaphase Chromosomes / protein kinase CK2 complex / symbiont-mediated disruption of host cell PML body / Receptor Mediated Mitophagy / Sin3-type complex / Synthesis of PC / negative regulation of signal transduction by p53 class mediator / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / Maturation of hRSV A proteins / negative regulation of apoptotic signaling pathway / negative regulation of double-strand break repair via homologous recombination / positive regulation of Wnt signaling pathway / negative regulation of proteasomal ubiquitin-dependent protein catabolic process / Signal transduction by L1 / Hsp90 protein binding / PML body / Wnt signaling pathway / Regulation of PTEN stability and activity / kinase activity / positive regulation of protein catabolic process / KEAP1-NFE2L2 pathway / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / rhythmic process / double-strand break repair / protein folding / positive regulation of cell growth / Regulation of TP53 Activity through Phosphorylation / non-specific serine/threonine protein kinase / regulation of cell cycle / negative regulation of translation / protein stabilization / protein serine kinase activity / protein serine/threonine kinase activity / apoptotic process / positive regulation of cell population proliferation / DNA damage response / signal transduction / nucleoplasm / ATP binding / identical protein binding / nucleus / plasma membrane / cytosol Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2 Å MOLECULAR REPLACEMENT / Resolution: 2 Å | ||||||
 Authors Authors | Hochscherf, J. / Lindenblatt, D. / Witulski, B. / Birus, R. / Aichele, D. / Marminon, C. / Bouaziz, Z. / Le Borgne, M. / Jose, J. / Niefind, K. | ||||||
 Citation Citation |  Journal: Pharmaceuticals (Basel) / Year: 2017 Journal: Pharmaceuticals (Basel) / Year: 2017Title: Unexpected Binding Mode of a Potent Indeno[1,2-b]indole-Type Inhibitor of Protein Kinase CK2 Revealed by Complex Structures with the Catalytic Subunit CK2 alpha and Its Paralog CK2 alpha '. Authors: Hochscherf, J. / Lindenblatt, D. / Witulski, B. / Birus, R. / Aichele, D. / Marminon, C. / Bouaziz, Z. / Le Borgne, M. / Jose, J. / Niefind, K. #1: Journal: Pharmaceuticals (Basel) / Year: 2017 Title: Development of Pharmacophore Model for Indeno[1,2-b]indoles as Human Protein Kinase CK2 Inhibitors and Database Mining. Authors: Haidar, S. / Bouaziz, Z. / Marminon, C. / Laitinen, T. / Poso, A. / Le Borgne, M. / Jose, J. #2:  Journal: J. Mol. Biol. / Year: 2003 Journal: J. Mol. Biol. / Year: 2003Title: Crystal structure of a C-terminal deletion mutant of human protein kinase CK2 catalytic subunit. Authors: Ermakova, I. / Boldyreff, B. / Issinger, O.G. / Niefind, K. #3: Journal: Bioorg. Med. Chem. / Year: 2012 Title: Indeno[1,2-b]indole derivatives as a novel class of potent human protein kinase CK2 inhibitors. Authors: Hundsdoerfer, C. / Hemmerling, H.J. / Goetz, C. / Totzke, F. / Bednarski, P. / Le Borgne, M. / Jose, J. #4: Journal: Biochem. Biophys. Res. Commun. / Year: 2012 Title: Novel indeno[1,2-b]indoloquinones as inhibitors of the human protein kinase CK2 with antiproliferative activity towards a broad panel of cancer cell lines. Authors: Hundsdoerfer, C. / Hemmerling, H.J. / Hamberger, J. / Le Borgne, M. / Bednarski, P. / Goetz, C. / Totzke, F. / Jose, J. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5oni.cif.gz 5oni.cif.gz | 304.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5oni.ent.gz pdb5oni.ent.gz | 248.9 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5oni.json.gz 5oni.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/on/5oni https://data.pdbj.org/pub/pdb/validation_reports/on/5oni ftp://data.pdbj.org/pub/pdb/validation_reports/on/5oni ftp://data.pdbj.org/pub/pdb/validation_reports/on/5oni | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5omyC  5ooiC  2pvrS C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 2 molecules AB
| #1: Protein | Mass: 45208.559 Da / Num. of mol.: 2 / Mutation: C-terminal deletion from Ser336 to Gln391 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: CSNK2A1, CK2A1 / Production host: Homo sapiens (human) / Gene: CSNK2A1, CK2A1 / Production host:  References: UniProt: P68400, non-specific serine/threonine protein kinase |
|---|
-Non-polymers , 6 types, 313 molecules 



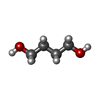






| #2: Chemical | | #3: Chemical | ChemComp-SO4 / #4: Chemical | ChemComp-NA / | #5: Chemical | #6: Chemical | ChemComp-BU1 / | #7: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.2 Å3/Da / Density % sol: 61.62 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, sitting drop Details: 90 MICROLITER ENZYME STOCK SOLUTION (6 MG/ML IN 500 MM NACL, 25 MM TRIS/HCL, PH 8.5) WAS MIXED WITH 10 MIKROLITER 4P STOCK SOLUTION (10 MM 4P IN DMSO). THIS MIXTURE WAS INCUBATED FOR 30 MIN ...Details: 90 MICROLITER ENZYME STOCK SOLUTION (6 MG/ML IN 500 MM NACL, 25 MM TRIS/HCL, PH 8.5) WAS MIXED WITH 10 MIKROLITER 4P STOCK SOLUTION (10 MM 4P IN DMSO). THIS MIXTURE WAS INCUBATED FOR 30 MIN AT ROOM TEMPERATURE. THE RESERVOIR SOLUTION OF THE CRYSTALLIZATION EXPERIMENT WAS 25 % (W/V) PEG5000, 0.2 M AMMONIUM SULPHATE, 0.1 M MES BUFFER, PH 6.5. PRIOR TO EQUILIBRATION THE CRYSTALLIZATION DROP WAS COMPOSED OF 1 MICROLITER RESERVOIR SOLUTION PLUS 1 MICROLITER ENZYME/4P MIXTURE., VAPOR DIFFUSION, SITTING DROP, TEMPERATURE 293K |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ESRF ESRF  / Beamline: MASSIF-1 / Wavelength: 0.966 Å / Beamline: MASSIF-1 / Wavelength: 0.966 Å |
| Detector | Type: DECTRIS PILATUS 2M / Detector: PIXEL / Date: Dec 15, 2016 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.966 Å / Relative weight: 1 |
| Reflection | Resolution: 2→57.041 Å / Num. obs: 70221 / % possible obs: 99.51 % / Redundancy: 6.2 % / Biso Wilson estimate: 44.4 Å2 / CC1/2: 0.999 / Rmerge(I) obs: 0.06473 / Rsym value: 0.06473 / Net I/σ(I): 14.2 |
| Reflection shell | Resolution: 2→2.072 Å / Redundancy: 6.3 % / Rmerge(I) obs: 1.909 / Mean I/σ(I) obs: 0.93 / Num. unique obs: 6881 / CC1/2: 0.437 / Rsym value: 1.909 / % possible all: 98.84 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 2PVR Resolution: 2→57.041 Å / SU ML: 0.22 / Cross valid method: FREE R-VALUE / σ(F): 1.97 / Phase error: 22.44
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2→57.041 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller






















 PDBj
PDBj












