+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4zyb | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | High resolution structure of M23 peptidase LytM with substrate analogue | ||||||
 要素 要素 | Glycyl-glycine endopeptidase LytM | ||||||
 キーワード キーワード | HYDROLASE / LYTM / LYSOSTAPHIN / PEPTIDOGLYCAN AMIDASE / PEPTIDASE / TETRAGLYCINE PHOSPHINATE / TRANSITION STATE ANALOGUE / COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lysostaphin / cobalt ion binding / peptide catabolic process / nickel cation binding / cell wall organization / metalloendopeptidase activity / manganese ion binding / proteolysis / extracellular region / zinc ion binding / metal ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  Staphylococcus aureus subsp. aureus NCTC 8325 (黄色ブドウ球菌) Staphylococcus aureus subsp. aureus NCTC 8325 (黄色ブドウ球菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.5 Å 分子置換 / 解像度: 1.5 Å | ||||||
 データ登録者 データ登録者 | Grabowska, M. / Jagielska, E. / Czapinska, H. / Bochtler, M. / Sabala, I. | ||||||
| 資金援助 |  ポーランド, 1件 ポーランド, 1件
| ||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2015 ジャーナル: Sci Rep / 年: 2015タイトル: High resolution structure of an M23 peptidase with a substrate analogue. 著者: Grabowska, M. / Jagielska, E. / Czapinska, H. / Bochtler, M. / Sabala, I. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4zyb.cif.gz 4zyb.cif.gz | 264.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4zyb.ent.gz pdb4zyb.ent.gz | 212.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4zyb.json.gz 4zyb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4zyb_validation.pdf.gz 4zyb_validation.pdf.gz | 1.7 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4zyb_full_validation.pdf.gz 4zyb_full_validation.pdf.gz | 1.7 MB | 表示 | |
| XML形式データ |  4zyb_validation.xml.gz 4zyb_validation.xml.gz | 33.3 KB | 表示 | |
| CIF形式データ |  4zyb_validation.cif.gz 4zyb_validation.cif.gz | 50 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/zy/4zyb https://data.pdbj.org/pub/pdb/validation_reports/zy/4zyb ftp://data.pdbj.org/pub/pdb/validation_reports/zy/4zyb ftp://data.pdbj.org/pub/pdb/validation_reports/zy/4zyb | HTTPS FTP |
-関連構造データ
| 関連構造データ |  2b0pS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 4分子 ABCD
| #1: タンパク質 | 分子量: 14564.811 Da / 分子数: 4 / 断片: unp residues 185-316 / 由来タイプ: 組換発現 由来: (組換発現)  Staphylococcus aureus subsp. aureus NCTC 8325 (黄色ブドウ球菌) Staphylococcus aureus subsp. aureus NCTC 8325 (黄色ブドウ球菌)遺伝子: lytM, SAS0252 / プラスミド: PLASMID / 詳細 (発現宿主): PET15B / 発現宿主:  |
|---|
-非ポリマー , 9種, 889分子 



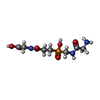










| #2: 化合物 | ChemComp-ZN / #3: 化合物 | ChemComp-CA / #4: 化合物 | ChemComp-CL / #5: 化合物 | ChemComp-UNL / 分子量: 103.120 Da / 分子数: 4 / 由来タイプ: 合成 #6: 化合物 | #7: 化合物 | ChemComp-4SQ / #8: 化合物 | #9: 化合物 | ChemComp-PEG / | #10: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.19 Å3/Da / 溶媒含有率: 43.76 % |
|---|---|
| 結晶化 | 温度: 290 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 詳細: 0.05 M HEPES-CsOH pH 7.5, 0.2 M calcium chloride and 25% polyethylene glycol 4000 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  BESSY BESSY  / ビームライン: 14.1 / 波長: 0.917 Å / ビームライン: 14.1 / 波長: 0.917 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2013年11月24日 |
| 放射 | モノクロメーター: KMC-1 / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.917 Å / 相対比: 1 |
| 反射 | 解像度: 1.45→40 Å / Num. obs: 87629 / % possible obs: 99.7 % / 冗長度: 6.8 % / CC1/2: 0.998 / Rmerge(I) obs: 0.121 / Rsym value: 0.112 / Net I/σ(I): 12 |
| 反射 シェル | 解像度: 1.45→1.54 Å / 冗長度: 6.5 % / Rmerge(I) obs: 0.706 / Mean I/σ(I) obs: 2.5 / CC1/2: 0.813 / Rsym value: 0.649 / % possible all: 98.1 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 2B0P 解像度: 1.5→36.3 Å / SU ML: 0.12 / 交差検証法: FREE R-VALUE / σ(F): 1.36 / 位相誤差: 17.15 / 立体化学のターゲット値: ML 詳細: The N-terminus of the truncated protein (absent in the full length native LytM) is disordered and has been built in only tentatively. The ligand could be reliably modelled in the C-terminal ...詳細: The N-terminus of the truncated protein (absent in the full length native LytM) is disordered and has been built in only tentatively. The ligand could be reliably modelled in the C-terminal and phosphinate part, its N-terminus is most likely disordered, since none of its conformations was clearly prevailing in all four molecules in the asymmetric unit. There are numerous close contacts between solvent molecules in the structure which might either result from their static disorder, or poorly ordered PEG molecules, or low occupancy metal ions all of which have been modelled tentatively in a few cases. Alternatively they may result from crystal defects (crystallites of different symmetries forming the macroscopically observed crystal). The identity of a few ions is unsure - for the technical reasons the unsure Cl- ions were kept as Cl ions whereas the unsure Na ions have been submitted as UNL atoms. For a few serine residues we observe difference density close to the side chain oxygen atom.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.5→36.3 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj








