ソフトウェア 名称 バージョン 分類 NB 精密化 Show 3.006 データ抽出 Show
精密化 構造決定の手法 開始モデル 解像度 / Occupancy max / Occupancy min / FOM work R set / SU ML / 交差検証法 / σ(F) / 立体化学のターゲット値 Rfactor 反射数 %反射 Selection details Rfree 0.224 5040 5.01 % random Rwork 0.196 - - - all 0.2 106726 - - obs 0.197 100656 94.31 % -
溶媒の処理 減衰半径 / VDWプローブ半径 / 溶媒モデル / Bsol 2 / ksol 3 原子変位パラメータ Biso max 2 / Biso mean 2 / Biso min 2 Baniso -1 Baniso -2 Baniso -3 1- 0.28 Å2 0 Å2 -0 Å2 2- - 0.28 Å2 0 Å2 3- - - -0.559 Å2
精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 3869 0 35 498 4402
拘束条件 Refine-ID タイプ Dev ideal 数 X-RAY DIFFRACTION f_bond_d0.016 3982 X-RAY DIFFRACTION f_angle_d1.517 5420 X-RAY DIFFRACTION f_chiral_restr0.11 619 X-RAY DIFFRACTION f_plane_restr0.007 700 X-RAY DIFFRACTION f_dihedral_angle_d16.826 1328
LS精密化 シェル Refine-ID / Total num. of bins used
大きな表を表示 (7 x 30) 大きな表を隠す 解像度 (Å)Rfactor Rfree Num. reflection Rfree Rfactor Rwork Num. reflection Rwork Num. reflection all % reflection obs (%)1.639-1.658 0.424 87 0.377 1386 1473 41 1.658-1.677 0.4 112 0.382 1799 1911 54 1.677-1.698 0.411 111 0.365 2325 2436 69 1.698-1.719 0.364 140 0.356 2751 2891 82 1.719-1.742 0.336 158 0.332 3058 3216 92 1.742-1.766 0.351 158 0.324 3222 3380 96 1.766-1.791 0.319 153 0.314 3295 3448 97 1.791-1.817 0.32 177 0.317 3301 3478 99 1.817-1.846 0.341 182 0.302 3305 3487 99 1.846-1.876 0.303 173 0.288 3341 3514 99 1.876-1.908 0.294 172 0.276 3355 3527 100 1.908-1.943 0.279 203 0.255 3330 3533 100 1.943-1.981 0.281 180 0.228 3362 3542 100 1.981-2.021 0.252 174 0.211 3358 3532 100 2.021-2.065 0.233 179 0.206 3359 3538 100 2.065-2.113 0.255 182 0.195 3372 3554 100 2.113-2.166 0.198 150 0.184 3414 3564 100 2.166-2.224 0.207 190 0.177 3342 3532 100 2.224-2.29 0.192 172 0.18 3374 3546 100 2.29-2.364 0.197 202 0.175 3353 3555 100 2.364-2.448 0.22 185 0.174 3374 3559 100 2.448-2.546 0.199 196 0.172 3381 3577 100 2.546-2.662 0.221 180 0.176 3354 3534 100 2.662-2.803 0.227 155 0.18 3404 3559 100 2.803-2.978 0.24 175 0.178 3418 3593 100 2.978-3.208 0.197 184 0.173 3397 3581 100 3.208-3.531 0.189 163 0.154 3426 3589 100 3.531-4.041 0.168 176 0.139 3449 3625 100 4.041-5.09 0.139 187 0.129 3447 3634 100 5.09-44.119 0.199 184 0.19 3564 3748 100
精密化 TLS 手法 / Refine-ID
大きな表を表示 (25 x 2) 大きな表を隠す ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)S33 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 0.5067 0.1485 0.2538 0.5167 0.2055 1.0882 0.0443 -0.0368 -0.0012 -0.0164 -0.0386 -0.0023 0.0493 0.0056 -0.0044 0.07 -0.0164 0.0083 0.1016 -0.0163 0.0675 9.1883 -62.0671 -14.9709 2 3.8982 -0.6772 0.0445 0.7605 -0.0275 0.9456 0.1461 0.2426 1.0077 -0.0435 -0.171 -0.1734 -0.1204 -0.0344 0.0094 0.09 0.0029 0.0434 0.1316 0.0611 0.3874 35.7796 -34.5038 -10.6219
精密化 TLSグループ ID Refine-ID Refine TLS-ID Selection details Auth asym-ID Auth seq-ID 1 X-RAY DIFFRACTION 1 chain AA0 - 274 2 X-RAY DIFFRACTION 2 chain BB1 - 274
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.639 Å
分子置換 / 解像度: 1.639 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 2009
ジャーナル: J.Mol.Biol. / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3ggh.cif.gz
3ggh.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3ggh.ent.gz
pdb3ggh.ent.gz PDB形式
PDB形式 3ggh.json.gz
3ggh.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3ggh_validation.pdf.gz
3ggh_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3ggh_full_validation.pdf.gz
3ggh_full_validation.pdf.gz 3ggh_validation.xml.gz
3ggh_validation.xml.gz 3ggh_validation.cif.gz
3ggh_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/gg/3ggh
https://data.pdbj.org/pub/pdb/validation_reports/gg/3ggh ftp://data.pdbj.org/pub/pdb/validation_reports/gg/3ggh
ftp://data.pdbj.org/pub/pdb/validation_reports/gg/3ggh リンク
リンク 集合体
集合体
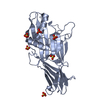

 要素
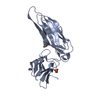
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID14-1 / 波長: 0.9834 Å
/ ビームライン: ID14-1 / 波長: 0.9834 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



















 PDBj
PDBj


