+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2n9b | ||||||
|---|---|---|---|---|---|---|---|
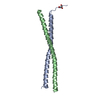
| タイトル | Solution NMR Structure of Antiparallel Myosin-10:GCN4 Tandem Coiled-Coil | ||||||
 要素 要素 | Unconventional myosin-X, General control protein GCN4 fusion | ||||||
 キーワード キーワード | MOTOR PROTEIN/TRANSCRIPTION / anti-parallel coiled-coil / coiled-coil / MOTOR PROTEIN-TRANSCRIPTION complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plus-end directed microfilament motor activity / cytoskeleton-dependent intracellular transport / filopodium tip / FCERI mediated MAPK activation / protein localization to nuclear periphery / Activation of the AP-1 family of transcription factors / regulation of filopodium assembly / response to amino acid starvation / negative regulation of ribosomal protein gene transcription by RNA polymerase II / positive regulation of cellular response to amino acid starvation ...plus-end directed microfilament motor activity / cytoskeleton-dependent intracellular transport / filopodium tip / FCERI mediated MAPK activation / protein localization to nuclear periphery / Activation of the AP-1 family of transcription factors / regulation of filopodium assembly / response to amino acid starvation / negative regulation of ribosomal protein gene transcription by RNA polymerase II / positive regulation of cellular response to amino acid starvation / mediator complex binding / Oxidative Stress Induced Senescence / filopodium membrane / myosin complex / microfilament motor activity / phosphatidylinositol-3,4,5-trisphosphate binding / TFIID-class transcription factor complex binding / amino acid biosynthetic process / positive regulation of RNA polymerase II transcription preinitiation complex assembly / positive regulation of transcription initiation by RNA polymerase II / cellular response to nutrient levels / ruffle / cellular response to amino acid starvation / filopodium / RNA polymerase II transcription regulator complex / actin filament binding / regulation of cell shape / lamellipodium / DNA-binding transcription activator activity, RNA polymerase II-specific / cell cortex / transcription regulator complex / sequence-specific DNA binding / RNA polymerase II-specific DNA-binding transcription factor binding / DNA-binding transcription factor activity, RNA polymerase II-specific / calmodulin binding / intracellular signal transduction / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / chromatin binding / negative regulation of transcription by RNA polymerase II / signal transduction / positive regulation of transcription by RNA polymerase II / ATP binding / identical protein binding / nucleus / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |   | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
| Model details | lowest energy, model1 | ||||||
 データ登録者 データ登録者 | Vavra, K.C. / Xia, Y. / Rock, R.S. | ||||||
 引用 引用 |  ジャーナル: Biophys.J. / 年: 2016 ジャーナル: Biophys.J. / 年: 2016タイトル: Competition between Coiled-Coil Structures and the Impact on Myosin-10 Bundle Selection 著者: Vavra, K.C. / Xia, Y. / Rock, R.S. #1:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2012 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2012タイトル: Antiparallel coiled-coil-mediated dimerization of myosin X. 著者: Lu, Q. / Ye, F. / Wei, Z. / Wen, Z. / Zhang, M. #2:  ジャーナル: Science / 年: 1991 ジャーナル: Science / 年: 1991タイトル: X-ray structure of the GCN4 leucine zipper, a two-stranded, parallel coiled coil 著者: Klemm, J.D. / Kim, P.S. / Alber, T. #3: ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2008 タイトル: A myosin motor that selects bundled actin for motility 著者: Nagy, S. / Ricca, B.L. / Norstrom, M.F. / Courson, D.F. / Brawley, C.M. / Smithback, P.A. / Rock, R.S. #4: ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1997 タイトル: Spare the rod, spoil the regulation: necessity for a myosin rod 著者: Trybus, K.M. / Freyzon, Y. / Faust, L.Z. / Sweeney, H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2n9b.cif.gz 2n9b.cif.gz | 456.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2n9b.ent.gz pdb2n9b.ent.gz | 386.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2n9b.json.gz 2n9b.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2n9b_validation.pdf.gz 2n9b_validation.pdf.gz | 536.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2n9b_full_validation.pdf.gz 2n9b_full_validation.pdf.gz | 724.6 KB | 表示 | |
| XML形式データ |  2n9b_validation.xml.gz 2n9b_validation.xml.gz | 36 KB | 表示 | |
| CIF形式データ |  2n9b_validation.cif.gz 2n9b_validation.cif.gz | 48.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/n9/2n9b https://data.pdbj.org/pub/pdb/validation_reports/n9/2n9b ftp://data.pdbj.org/pub/pdb/validation_reports/n9/2n9b ftp://data.pdbj.org/pub/pdb/validation_reports/n9/2n9b | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ | |
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 8200.290 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   遺伝子: MYO10, GCN4, AAS3, ARG9, YEL009C / 株: ATCC 204508 / S288c / 発現宿主:  |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR 詳細: Tandem antiparallel coiled-coil fusion derived from Bos taurus myosin-10 coiled-coil and GCN4-p1. Structure solved by solution NMR. Tertiary structure supported by Small-Angle X-ray scattering (see reference). | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | 内容: 1.1 mM [U-100% 13C; U-100% 15N] entity-1, 100 mM potassium phosphate-2, 1 mM EDTA-3, 0.03 % sodium azide-4, 90% H2O/10% D2O 溶媒系: 90% H2O/10% D2O | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||
| 試料状態 | イオン強度: 0.6 / pH: 6.5 / 圧: ambient / 温度: 308 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 | ||||||||||||||||||||||||||||
| NMR constraints | NOE constraints total: 1561 / NOE intraresidue total count: 332 / NOE long range total count: 149 / NOE medium range total count: 610 / NOE sequential total count: 470 / Hydrogen bond constraints total count: 294 / Protein phi angle constraints total count: 147 / Protein psi angle constraints total count: 147 | ||||||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 10 |
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj









 HSQC
HSQC