| 登録情報 | データベース: PDB / ID: 2n82
|
|---|
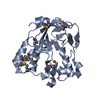
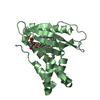
| タイトル | solution structure of the complex of microRNA 20b pre-element with Rbfox RRM |
|---|
 要素 要素 | - RNA (5'-R(*GP*GP*UP*AP*GP*UP*UP*UP*UP*GP*GP*CP*AP*UP*GP*AP*CP*UP*CP*UP*AP*CP*C)-3')
- RNA binding protein fox-1 homolog 1
|
|---|
 キーワード キーワード | RNA BINDING PROTEIN/RNA / microRNA / RRM / Rbfox / RNA BINDING PROTEIN-RNA complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
MECP2 regulates transcription factors / RNA transport / nuclear stress granule / regulation of alternative mRNA splicing, via spliceosome / RNA splicing / trans-Golgi network / cytoplasmic stress granule / mRNA processing / nervous system development / mRNA binding ...MECP2 regulates transcription factors / RNA transport / nuclear stress granule / regulation of alternative mRNA splicing, via spliceosome / RNA splicing / trans-Golgi network / cytoplasmic stress granule / mRNA processing / nervous system development / mRNA binding / RNA binding / nucleus / cytoplasm類似検索 - 分子機能 : / RNA binding protein Fox-1 / Fox-1 C-terminal domain / Calcitonin gene-related peptide regulator C terminal / FOX1, RNA recognition motif / RRM (RNA recognition motif) domain / RNA recognition motif / RNA recognition motif / Eukaryotic RNA Recognition Motif (RRM) profile. / RNA recognition motif domain ...: / RNA binding protein Fox-1 / Fox-1 C-terminal domain / Calcitonin gene-related peptide regulator C terminal / FOX1, RNA recognition motif / RRM (RNA recognition motif) domain / RNA recognition motif / RNA recognition motif / Eukaryotic RNA Recognition Motif (RRM) profile. / RNA recognition motif domain / RNA-binding domain superfamily / Nucleotide-binding alpha-beta plait domain superfamily / Alpha-Beta Plaits / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 RNA / RNA (> 10) / RNA binding protein fox-1 homolog 1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)
synthetic construct (人工物) |
|---|
| 手法 | 溶液NMR / restrained simulated annealing |
|---|
 データ登録者 データ登録者 | Yang, F. / Chen, Y. / Varani, G. |
|---|
 引用 引用 |  ジャーナル: Nucleic Acids Res. / 年: 2016 ジャーナル: Nucleic Acids Res. / 年: 2016
タイトル: Rbfox proteins regulate microRNA biogenesis by sequence-specific binding to their precursors and target downstream Dicer.
著者: Chen, Y. / Zubovic, L. / Yang, F. / Godin, K. / Pavelitz, T. / Castellanos, J. / Macchi, P. / Varani, G. |
|---|
| 履歴 | | 登録 | 2015年9月30日 | 登録サイト: BMRB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2016年4月13日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2016年6月1日 | Group: Database references |
|---|
| 改定 1.2 | 2023年6月14日 | Group: Data collection / Database references / Other
カテゴリ: database_2 / pdbx_database_status ...database_2 / pdbx_database_status / pdbx_nmr_software / pdbx_nmr_spectrometer / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _struct_ref_seq_dif.details |
|---|
| 改定 1.3 | 2024年5月15日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Nucleic Acids Res. / 年: 2016
ジャーナル: Nucleic Acids Res. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2n82.cif.gz
2n82.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2n82.ent.gz
pdb2n82.ent.gz PDB形式
PDB形式 2n82.json.gz
2n82.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2n82_validation.pdf.gz
2n82_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2n82_full_validation.pdf.gz
2n82_full_validation.pdf.gz 2n82_validation.xml.gz
2n82_validation.xml.gz 2n82_validation.cif.gz
2n82_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/n8/2n82
https://data.pdbj.org/pub/pdb/validation_reports/n8/2n82 ftp://data.pdbj.org/pub/pdb/validation_reports/n8/2n82
ftp://data.pdbj.org/pub/pdb/validation_reports/n8/2n82 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: RBFOX1, A2BP, A2BP1, FOX1, HRNBP1 / プラスミド: PET28 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: RBFOX1, A2BP, A2BP1, FOX1, HRNBP1 / プラスミド: PET28 / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj





























 HSQC
HSQC