| 登録情報 | データベース: PDB / ID: 2klc
|
|---|
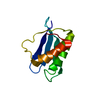
| タイトル | NMR solution structure of human ubiquitin-like domain of ubiquilin 1, Northeast Structural Genomics Consortium (NESG) target HT5A |
|---|
 要素 要素 | Ubiquilin-1 |
|---|
 キーワード キーワード | Structural Genomics / Unknown function / ubiquitin-like / PSI-2 / Protein Structure Initiative / Northeast Structural Genomics Consortium / NESG / Structural Genomics Consortium / SGC / protein binding / UBL / Ontario Centre for Structural Proteomics / OCSP / proteasome / ubiquitin ligase-associated / ubiquitination / protein degradation / Alzheimer's / Nucleus / Phosphoprotein / Ontario Centre for Structural Proteomics (OCSP) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
negative regulation of store-operated calcium channel activity / regulation of oxidative stress-induced intrinsic apoptotic signaling pathway / negative regulation of toll-like receptor 3 signaling pathway / positive regulation of ERAD pathway / aggrephagy / aggresome / autophagosome assembly / polyubiquitin modification-dependent protein binding / autophagosome maturation / regulation of protein ubiquitination ...negative regulation of store-operated calcium channel activity / regulation of oxidative stress-induced intrinsic apoptotic signaling pathway / negative regulation of toll-like receptor 3 signaling pathway / positive regulation of ERAD pathway / aggrephagy / aggresome / autophagosome assembly / polyubiquitin modification-dependent protein binding / autophagosome maturation / regulation of protein ubiquitination / regulation of macroautophagy / ERAD pathway / proteasome complex / autophagosome / response to endoplasmic reticulum stress / positive regulation of protein ubiquitination / macroautophagy / kinase binding / Cargo recognition for clathrin-mediated endocytosis / ubiquitin-dependent protein catabolic process / cytoplasmic vesicle / cellular response to hypoxia / perinuclear region of cytoplasm / endoplasmic reticulum / protein-containing complex / nucleoplasm / identical protein binding / plasma membrane / cytosol / cytoplasm類似検索 - 分子機能 Ubiquilin-1-like domain / Heat shock chaperonin-binding / Heat shock chaperonin-binding motif. / : / UBA/TS-N domain / Ubiquitin associated domain / Ubiquitin-associated domain / Ubiquitin-associated domain (UBA) profile. / UBA-like superfamily / Phosphatidylinositol 3-kinase Catalytic Subunit; Chain A, domain 1 ...Ubiquilin-1-like domain / Heat shock chaperonin-binding / Heat shock chaperonin-binding motif. / : / UBA/TS-N domain / Ubiquitin associated domain / Ubiquitin-associated domain / Ubiquitin-associated domain (UBA) profile. / UBA-like superfamily / Phosphatidylinositol 3-kinase Catalytic Subunit; Chain A, domain 1 / Ubiquitin-like (UB roll) / Ubiquitin family / Ubiquitin homologues / Ubiquitin domain profile. / Ubiquitin-like domain / Ubiquitin-like domain superfamily / Roll / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / restrained molecular dynamics |
|---|
| Model details | lowest energy, model 1 |
|---|
 データ登録者 データ登録者 | Doherty, R.S. / Dhe-Paganon, S. / Fares, C. / Lemak, S. / Gutmanas, A. / Garcia, M. / Yee, A. / Montelione, G.T. / Arrowsmith, C.H. / Northeast Structural Genomics Consortium (NESG) ...Doherty, R.S. / Dhe-Paganon, S. / Fares, C. / Lemak, S. / Gutmanas, A. / Garcia, M. / Yee, A. / Montelione, G.T. / Arrowsmith, C.H. / Northeast Structural Genomics Consortium (NESG) / Structural Genomics Consortium (SGC) / Ontario Centre for Structural Proteomics (OCSP) |
|---|
 引用 引用 | |
|---|
| 履歴 | | 登録 | 2009年6月30日 | 登録サイト: BMRB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2009年7月14日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2012年1月18日 | Group: Database references |
|---|
| 改定 1.3 | 2020年2月26日 | Group: Data collection / Database references / Other
カテゴリ: pdbx_database_status / pdbx_nmr_software ...pdbx_database_status / pdbx_nmr_software / pdbx_nmr_spectrometer / struct_ref_seq_dif
Item: _pdbx_database_status.status_code_cs / _pdbx_nmr_software.name ..._pdbx_database_status.status_code_cs / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _struct_ref_seq_dif.details |
|---|
| 改定 1.4 | 2024年5月1日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2klc.cif.gz
2klc.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2klc.ent.gz
pdb2klc.ent.gz PDB形式
PDB形式 2klc.json.gz
2klc.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/kl/2klc
https://data.pdbj.org/pub/pdb/validation_reports/kl/2klc ftp://data.pdbj.org/pub/pdb/validation_reports/kl/2klc
ftp://data.pdbj.org/pub/pdb/validation_reports/kl/2klc リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: DA41, PLIC1, UBQLN1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: DA41, PLIC1, UBQLN1 / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj


 HNCA
HNCA