+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1i9c | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | GLUTAMATE MUTASE FROM CLOSTRIDIUM COCHLEARIUM: COMPLEX WITH ADENOSYLCOBALAMIN AND SUBSTRATE | ||||||
 要素 要素 | (GLUTAMATE MUTASE) x 2 | ||||||
 キーワード キーワード | ISOMERASE / COENZYME B12 / RADICAL REACTION / RIBOSE PSEUDOROTATION / TIM-BARREL / ROSSMAN-FOLD | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報methylaspartate mutase / anaerobic L-glutamate catabolic process / methylaspartate mutase activity / L-glutamate catabolic process via L-citramalate / cobalamin binding / metal ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  Clostridium cochlearium (バクテリア) Clostridium cochlearium (バクテリア) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / RIGID BODY REFINEMENT / 解像度: 1.9 Å シンクロトロン / RIGID BODY REFINEMENT / 解像度: 1.9 Å | ||||||
 データ登録者 データ登録者 | Gruber, K. / Kratky, C. | ||||||
 引用 引用 |  ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2001 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2001タイトル: Radical Shuttling in a Protein: Ribose Pseudorotation Controls Alkyl-Radical Transfer in the Coenzyme B(12) Dependent Enzyme Glutamate Mutase 著者: Gruber, K. / Reitzer, R. / Kratky, C. #1:  ジャーナル: Structure / 年: 1999 ジャーナル: Structure / 年: 1999タイトル: Glutamate Mutase from Clostridum Cochlearium: The Structure of a Coenzyme B12-Dependent Enzyme Provides New Mechanistic Insights 著者: Reitzer, R. / Gruber, K. / Jogl, G. / Wagner, U.G. / Bothe, H. / Buckel, W. / Kratky, C. #2:  ジャーナル: Acta Crystallogr.,Sect.D / 年: 1998 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1998タイトル: Crystallization and Preliminary X-Ray Analysis of Recombinant Glutamate Mutase and of the Isolated Component S from Clostridium Cochlearium 著者: Reitzer, R. / Krasser, M. / Jogl, G. / Buckel, W. / Bothe, H. / Kratky, C. #3:  ジャーナル: Eur.J.Biochem. / 年: 1994 ジャーナル: Eur.J.Biochem. / 年: 1994タイトル: Characterization of the Coenzyme-B12-Dependent Glutamate Mutase from Clostridium Cochlearium Produced in Escherischia Coli 著者: Zelder, O. / Beatrix, B. / Leutbecher, U. / Buckel, W. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1i9c.cif.gz 1i9c.cif.gz | 293.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1i9c.ent.gz pdb1i9c.ent.gz | 230.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1i9c.json.gz 1i9c.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1i9c_validation.pdf.gz 1i9c_validation.pdf.gz | 2.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1i9c_full_validation.pdf.gz 1i9c_full_validation.pdf.gz | 2.3 MB | 表示 | |
| XML形式データ |  1i9c_validation.xml.gz 1i9c_validation.xml.gz | 61.3 KB | 表示 | |
| CIF形式データ |  1i9c_validation.cif.gz 1i9c_validation.cif.gz | 90.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/i9/1i9c https://data.pdbj.org/pub/pdb/validation_reports/i9/1i9c ftp://data.pdbj.org/pub/pdb/validation_reports/i9/1i9c ftp://data.pdbj.org/pub/pdb/validation_reports/i9/1i9c | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1ccwS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | The biological assembly is the heterotetramer present in the asymmetric unit |
- 要素
要素
-タンパク質 , 2種, 4分子 ACBD
| #1: タンパク質 | 分子量: 14830.991 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  Clostridium cochlearium (バクテリア) Clostridium cochlearium (バクテリア)遺伝子: GLMS / プラスミド: POZ3 / 発現宿主:  #2: タンパク質 | 分子量: 53623.172 Da / 分子数: 2 / 由来タイプ: 組換発現 詳細: COMPLEXED WITH ADENOSYLCOBALAMAIN AND A MIXTURE OF GLUTAMATE AND 2S,3S-3-METHYLASPARTIC ACID 由来: (組換発現)  Clostridium cochlearium (バクテリア) Clostridium cochlearium (バクテリア)遺伝子: GLME / プラスミド: POZ5 / 発現宿主:  |
|---|
-非ポリマー , 5種, 1266分子 


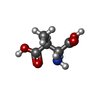





| #3: 化合物 | | #4: 化合物 | #5: 化合物 | #6: 化合物 | #7: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.4 Å3/Da / 溶媒含有率: 48.7 % | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 4.6 詳細: PEG-4000, cadmium chloride, coenzyme B12, sodium glutamate, pH 4.6, VAPOR DIFFUSION, SITTING DROP, temperature 293K | ||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS pH: 4.5 | ||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 103 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / ビームライン: BW7B / 波長: 0.8439 / ビームライン: BW7B / 波長: 0.8439 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1999年8月15日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.8439 Å / 相対比: 1 |
| 反射 | 解像度: 1.9→30 Å / Num. all: 121680 / Num. obs: 121680 / % possible obs: 99.9 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 4.6 % / Biso Wilson estimate: 15.5 Å2 / Rmerge(I) obs: 0.073 / Rsym value: 0.073 / Net I/σ(I): 18.4 |
| 反射 シェル | 解像度: 1.9→1.94 Å / 冗長度: 3.7 % / Rmerge(I) obs: 0.281 / Mean I/σ(I) obs: 5 / Num. unique all: 8102 / Rsym value: 0.281 / % possible all: 100 |
| 反射 | *PLUS Num. measured all: 555148 |
| 反射 シェル | *PLUS % possible obs: 100 % / Num. unique obs: 8102 / Num. measured obs: 30037 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: RIGID BODY REFINEMENT 開始モデル: 1CCW 解像度: 1.9→30 Å / Num. parameters: 45086 / Num. restraintsaints: 56174 / Isotropic thermal model: ISOTROPIC / 交差検証法: FREE R / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: ENGH AND HUBER 詳細: ANISOTROPIC SCALING APPLIED BY THE METHOD OF PARKIN, MOEZZI & HOPE, J.APPL.CRYST.28(1995)53-56
| |||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: MOEWS & KRETSINGER, J.MOL.BIOL.91(1973)201-228 | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 13.3 Å2 | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 39 / Occupancy sum hydrogen: 9804 / Occupancy sum non hydrogen: 11102 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.9→30 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL / バージョン: 97 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 精密化 | *PLUS σ(F): 0 / % reflection Rfree: 5 % / Rfactor obs: 0.168 | |||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | |||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj





