+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1g96 | ||||||
|---|---|---|---|---|---|---|---|
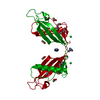
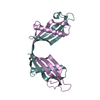
| タイトル | HUMAN CYSTATIN C; DIMERIC FORM WITH 3D DOMAIN SWAPPING | ||||||
 要素 要素 | CYSTATIN C | ||||||
 キーワード キーワード | hydrolase inhibitor / human cystatin C dimer / 3D domain swapping / amyloid formation / inhibitor of C1 and C13 cysteine proteases / AMYLOID ANGIOPATHY AND CEREBRAL HEMORRHAGE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of collagen catabolic process / negative regulation of elastin catabolic process / negative regulation of peptidase activity / negative regulation of blood vessel remodeling / peptidase inhibitor activity / negative regulation of extracellular matrix disassembly / regulation of tissue remodeling / endopeptidase inhibitor activity / cysteine-type endopeptidase inhibitor activity / supramolecular fiber organization ...negative regulation of collagen catabolic process / negative regulation of elastin catabolic process / negative regulation of peptidase activity / negative regulation of blood vessel remodeling / peptidase inhibitor activity / negative regulation of extracellular matrix disassembly / regulation of tissue remodeling / endopeptidase inhibitor activity / cysteine-type endopeptidase inhibitor activity / supramolecular fiber organization / negative regulation of proteolysis / Post-translational protein phosphorylation / defense response / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / tertiary granule lumen / amyloid-beta binding / protease binding / vesicle / ficolin-1-rich granule lumen / immune response / Amyloid fiber formation / endoplasmic reticulum lumen / Neutrophil degranulation / endoplasmic reticulum / Golgi apparatus / extracellular space / extracellular exosome / extracellular region / identical protein binding / plasma membrane / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.5 Å 分子置換 / 解像度: 2.5 Å | ||||||
 データ登録者 データ登録者 | Janowski, R. / Kozak, M. / Jankowska, E. / Grzonka, Z. / Grubb, A. / Abrahamson, M. / Jaskolski, M. | ||||||
 引用 引用 |  ジャーナル: Nat.Struct.Biol. / 年: 2001 ジャーナル: Nat.Struct.Biol. / 年: 2001タイトル: Human cystatin C, an amyloidogenic protein, dimerizes through three-dimensional domain swapping. 著者: Janowski, R. / Kozak, M. / Jankowska, E. / Grzonka, Z. / Grubb, A. / Abrahamson, M. / Jaskolski, M. #1:  ジャーナル: Acta Crystallogr.,Sect.D / 年: 1999 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1999タイトル: Expression of selenomethionyl derivative and preliminary crystallographic studies of human cystatin C 著者: Kozak, M. / Jankowska, E. / Janowski, R. / Grzonka, Z. / Grubb, A. / Alvarez Fernandez, M. / Abrahamson, M. / Jaskolski, M. #2:  ジャーナル: Embo J. / 年: 1988 ジャーナル: Embo J. / 年: 1988タイトル: The 2.0 angstrom X-ray crystal structure of chicken egg white cystatin and its possible mode of interaction with cysteine proteases 著者: Bode, W. / Engh, R. / Musil, D. / Thiele, U. / Huber, R. / Karshikov, A. / Brzin, J. / Kos, J. / Turk, V. #3:  ジャーナル: J.Mol.Biol. / 年: 1997 ジャーナル: J.Mol.Biol. / 年: 1997タイトル: NMR structural studies of human cystatin C dimers and monomers 著者: Ekiel, I. / Abrahamson, M. / Fulton, D.B. / Lindahl, P. / Storer, A.C. / Levadoux, W. / Lafrance, M. / Labelle, S. / Pomerleau, Y. / Groleau, D. / LeSauter, L. / Gehring, K. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1g96.cif.gz 1g96.cif.gz | 36.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1g96.ent.gz pdb1g96.ent.gz | 25.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1g96.json.gz 1g96.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1g96_validation.pdf.gz 1g96_validation.pdf.gz | 426.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1g96_full_validation.pdf.gz 1g96_full_validation.pdf.gz | 428.5 KB | 表示 | |
| XML形式データ |  1g96_validation.xml.gz 1g96_validation.xml.gz | 7.1 KB | 表示 | |
| CIF形式データ |  1g96_validation.cif.gz 1g96_validation.cif.gz | 8.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/g9/1g96 https://data.pdbj.org/pub/pdb/validation_reports/g9/1g96 ftp://data.pdbj.org/pub/pdb/validation_reports/g9/1g96 ftp://data.pdbj.org/pub/pdb/validation_reports/g9/1g96 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1cewS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||
| 2 | x 8
| ||||||||||
| 単位格子 |
| ||||||||||
| Components on special symmetry positions |
| ||||||||||
| 詳細 | Human cystatin C in the present structure forms crystallographic dimers with 3D swapped domains. The dimer is generated by two-fold rotation of the 4(2) axis using the following transformation: 1/2-x, -y, z. |
- 要素
要素
| #1: タンパク質 | 分子量: 13365.141 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / プラスミド: PHD 313 / 発現宿主: Homo sapiens (ヒト) / プラスミド: PHD 313 / 発現宿主:  |
|---|---|
| #2: 化合物 | ChemComp-CL / |
| #3: 化合物 | ChemComp-GOL / |
| #4: 水 | ChemComp-HOH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 4.3 Å3/Da / 溶媒含有率: 71 % | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 277 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.8 詳細: Lyophilized protein was dissolved in 100 mM sodium acetate buffer pH 4.8 containing 20 mM CaCl2. Droplets were equilibrated against 1 ml of reservoir with analogous buffer/CaCl2 solution. ...詳細: Lyophilized protein was dissolved in 100 mM sodium acetate buffer pH 4.8 containing 20 mM CaCl2. Droplets were equilibrated against 1 ml of reservoir with analogous buffer/CaCl2 solution. After 2 and 4 days, the reservoir solution was supplemented with 100 microliters of MPD, VAPOR DIFFUSION, HANGING DROP, temperature 277K | ||||||||||||||||||||||||||||||
| 結晶化 | *PLUS | ||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / ビームライン: BW7B / 波長: 0.8423 Å / ビームライン: BW7B / 波長: 0.8423 Å |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 2000年9月30日 |
| 放射 | モノクロメーター: monochromator / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.8423 Å / 相対比: 1 |
| 反射 | 解像度: 2.5→40 Å / Num. all: 8429 / Num. obs: 8429 / % possible obs: 99.2 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 19.9 % / Biso Wilson estimate: 40.6 Å2 / Rmerge(I) obs: 0.07 / Net I/σ(I): 27.8 |
| 反射 シェル | 解像度: 2.5→2.59 Å / 冗長度: 4.1 % / Rmerge(I) obs: 0.413 / Mean I/σ(I) obs: 2.5 / % possible all: 93.5 |
| 反射 | *PLUS Num. measured all: 167936 |
| 反射 シェル | *PLUS % possible obs: 93.5 % |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: N-terminally truncated chicken cystatin (1cew.pdb) converted to a polyalanine chain and limited to those fragments that had been modeled in electron density (residues 86-90 not included) 解像度: 2.5→20 Å / Rfactor Rfree error: 0.008 / Isotropic thermal model: individual isotropic / 交差検証法: free R / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: Engh & Huber / 詳細: maximum likelihood
| ||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: flat model / Bsol: 38.73 Å2 / ksol: 0.33 e/Å3 | ||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.5→20 Å
| ||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.5→2.59 Å / Rfactor Rfree error: 0.04 / Total num. of bins used: 10
| ||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: CNS / バージョン: 0.9 / 分類: refinement | ||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj





