+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7367 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single Molecule 3D Image of a Hole-Hole Homodimer Species Generated in the Production of a Bispecific Recombinant IgG1 (No. 015) | |||||||||||||||
 マップデータ マップデータ | Single Molecule 3D Image of a Hole-Hole Homodimer Species Generated in the Production of a Bispecific Recombinant IgG1 (No. 015) | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 | unidentified (未定義) | |||||||||||||||
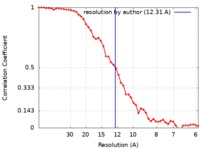
| 手法 | 電子線トモグラフィー法 / ネガティブ染色法 / 解像度: 12.31 Å | |||||||||||||||
 データ登録者 データ登録者 | Lei D / Liu J / Liu H / Lei M / Ren G | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2019 ジャーナル: Sci Rep / 年: 2019タイトル: Single-Molecule 3D Images of "Hole-Hole" IgG1 Homodimers by Individual-Particle Electron Tomography. 著者: Dongsheng Lei / Jianfang Liu / Hongbin Liu / Thomas E Cleveland / John P Marino / Ming Lei / Gang Ren /  要旨: The engineering of immunoglobulin-G molecules (IgGs) is of wide interest for improving therapeutics, for example by modulating the activity or multiplexing the specificity of IgGs to recognize more ...The engineering of immunoglobulin-G molecules (IgGs) is of wide interest for improving therapeutics, for example by modulating the activity or multiplexing the specificity of IgGs to recognize more than one antigen. Optimization of engineered IgG requires knowledge of three-dimensional (3D) structure of synthetic IgG. However, due to flexible nature of the molecules, their structural characterization is challenging. Here, we use our reported individual-particle electron tomography (IPET) method with optimized negative-staining (OpNS) for direct 3D reconstruction of individual IgG hole-hole homodimer molecules. The hole-hole homodimer is an undesired variant generated during the production of a bispecific antibody using the knob-into-hole heterodimer technology. A total of 64 IPET 3D density maps at ~15 Å resolutions were reconstructed from 64 individual molecules, revealing 64 unique conformations. In addition to the known Y-shaped conformation, we also observed an unusual X-shaped conformation. The 3D structure of the X-shaped conformation contributes to our understanding of the structural details of the interaction between two heavy chains in the Fc domain. The IPET approach, as an orthogonal technique to characterize the 3D structure of therapeutic antibodies, provides insight into the 3D structural variety and dynamics of heterogeneous IgG molecules. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7367.map.gz emd_7367.map.gz | 6.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7367-v30.xml emd-7367-v30.xml emd-7367.xml emd-7367.xml | 10.3 KB 10.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7367_fsc.xml emd_7367_fsc.xml | 4.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7367.png emd_7367.png | 12.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7367 http://ftp.pdbj.org/pub/emdb/structures/EMD-7367 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7367 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7367 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7367_validation.pdf.gz emd_7367_validation.pdf.gz | 77.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7367_full_validation.pdf.gz emd_7367_full_validation.pdf.gz | 77 KB | 表示 | |
| XML形式データ |  emd_7367_validation.xml.gz emd_7367_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7367 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7367 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7367 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7367 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7353C  7354C  7355C  7356C  7357C  7358C  7359C  7360C  7361C  7362C  7363C  7364C  7365C  7366C  7368C  7369C  7370C  7371C  7372C  7373C  7374C  7375C  7376C  7377C  7378C  7379C  7380C  7381C  7382C  7383C  7384C  7385C  7386C  7387C  7388C  7389C  7390C  7391C  7392C  7393C 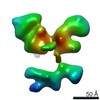 7394C  7395C  7396C  7397C 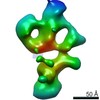 7398C  7399C 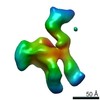 7400C  7401C  7402C  7404C  7405C  7406C  7407C  7408C  7409C 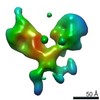 7410C  7411C  7412C 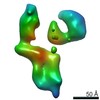 7413C  7414C  7415C  7416C  7417C  7418C  7419C  7420C  7421C  7422C  7423C 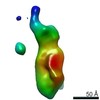 7424C  7425C  7426C 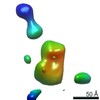 7427C  7428C  7429C  7430C  7431C  7432C  7433C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7367.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7367.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Single Molecule 3D Image of a Hole-Hole Homodimer Species Generated in the Production of a Bispecific Recombinant IgG1 (No. 015) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.96 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : IgG1 hole-hole homodimer
| 全体 | 名称: IgG1 hole-hole homodimer |
|---|---|
| 要素 |
|
-超分子 #1: IgG1 hole-hole homodimer
| 超分子 | 名称: IgG1 hole-hole homodimer / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 詳細: Dulbecco's phosphate buffered saline (DPBS) |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate / 詳細: The grid was stained with 1% (w/v) uranyl formate |
| グリッド | 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa |
| 切片作成 | その他: NO SECTIONING |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | ZEISS LIBRA120PLUS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均電子線量: 70.3 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 倍率(公称値): 80000 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)