+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the C. elegans Intron Lariat Spliceosome (Map 21) | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | mRNA / splicing / Intorn Lariat spliceosome / ILS / pre-mRNA | |||||||||
| 生物種 |  | |||||||||
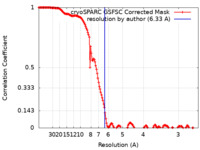
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.33 Å | |||||||||
 データ登録者 データ登録者 | Vorlaender MK / Rothe P / Plaschka C | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Mechanism for the initiation of spliceosome disassembly. 著者: Matthias K Vorländer / Patricia Rothe / Justus Kleifeld / Eric D Cormack / Lalitha Veleti / Daria Riabov-Bassat / Laura Fin / Alex W Phillips / Luisa Cochella / Clemens Plaschka /   要旨: Precursor-mRNA (pre-mRNA) splicing requires the assembly, remodelling and disassembly of the multi-megadalton ribonucleoprotein complex called the spliceosome. Recent studies have shed light on ...Precursor-mRNA (pre-mRNA) splicing requires the assembly, remodelling and disassembly of the multi-megadalton ribonucleoprotein complex called the spliceosome. Recent studies have shed light on spliceosome assembly and remodelling for catalysis, but the mechanism of disassembly remains unclear. Here we report cryo-electron microscopy structures of nematode and human terminal intron lariat spliceosomes along with biochemical and genetic data. Our results uncover how four disassembly factors and the conserved RNA helicase DHX15 initiate spliceosome disassembly. The disassembly factors probe large inner and outer spliceosome surfaces to detect the release of ligated mRNA. Two of these factors, TFIP11 and C19L1, and three general spliceosome subunits, SYF1, SYF2 and SDE2, then dock and activate DHX15 on the catalytic U6 snRNA to initiate disassembly. U6 therefore controls both the start and end of pre-mRNA splicing. Taken together, our results explain the molecular basis of the initiation of canonical spliceosome disassembly and provide a framework to understand general spliceosomal RNA helicase control and the discard of aberrant spliceosomes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50468.map.gz emd_50468.map.gz | 322.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50468-v30.xml emd-50468-v30.xml emd-50468.xml emd-50468.xml | 19.9 KB 19.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_50468_fsc.xml emd_50468_fsc.xml | 14.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_50468.png emd_50468.png | 68.9 KB | ||
| Filedesc metadata |  emd-50468.cif.gz emd-50468.cif.gz | 4.1 KB | ||
| その他 |  emd_50468_additional_1.map.gz emd_50468_additional_1.map.gz emd_50468_half_map_1.map.gz emd_50468_half_map_1.map.gz emd_50468_half_map_2.map.gz emd_50468_half_map_2.map.gz | 166.2 MB 318.7 MB 318.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50468 http://ftp.pdbj.org/pub/emdb/structures/EMD-50468 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50468 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50468 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_50468_validation.pdf.gz emd_50468_validation.pdf.gz | 899.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_50468_full_validation.pdf.gz emd_50468_full_validation.pdf.gz | 899.1 KB | 表示 | |
| XML形式データ |  emd_50468_validation.xml.gz emd_50468_validation.xml.gz | 23.5 KB | 表示 | |
| CIF形式データ |  emd_50468_validation.cif.gz emd_50468_validation.cif.gz | 31.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50468 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50468 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50468 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50468 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50468.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50468.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3013 Å | ||||||||||||||||||||||||||||||||||||
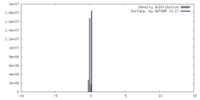
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_50468_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
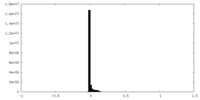
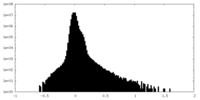
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_50468_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
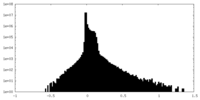
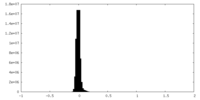
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_50468_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
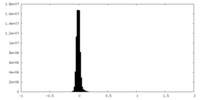
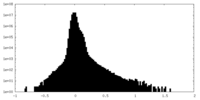
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Intron lariat spliceosome
| 全体 | 名称: Intron lariat spliceosome |
|---|---|
| 要素 |
|
-超分子 #1: Intron lariat spliceosome
| 超分子 | 名称: Intron lariat spliceosome / タイプ: complex / ID: 1 / 親要素: 0 含まれる分子: #4, #2, #5-#6, #8, #3, #10, #1, #9, #11-#13, #16-#19, #21-#24, #15, #7, #26-#27, #14, #28-#35, #20, #36-#38, #25, #39-#40 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.9 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
| 詳細 | Crosslinked with glutaraledhyde |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | プロトコル: AB INITIO MODEL |
 ムービー
ムービー コントローラー
コントローラー


















































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)