+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4648 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Packaging of DNA Origami in Viral Capsids | |||||||||
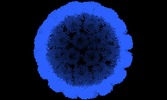
 マップデータ マップデータ | Empty capsid from DNA origami-filled SV40 (VP1) preparation. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Simian virus 40 (ウイルス) Simian virus 40 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.8 Å | |||||||||
 データ登録者 データ登録者 | Kopatz I / Zalk R / Levi-Kalisman Y / Zlotkin-Rivkin E / Frank GA / Kler S | |||||||||
 引用 引用 |  ジャーナル: Nanoscale / 年: 2019 ジャーナル: Nanoscale / 年: 2019タイトル: Packaging of DNA origami in viral capsids. 著者: Idit Kopatz / Ran Zalk / Yael Levi-Kalisman / Efrat Zlotkin-Rivkin / Gabriel A Frank / Stanislav Kler /  要旨: Here we show the encapsulation of 35 nm diameter, nearly-spherical, DNA origami by self-assembly of SV40-like (simian virus 40) particles. The self-assembly of this new type of nanoparticles is ...Here we show the encapsulation of 35 nm diameter, nearly-spherical, DNA origami by self-assembly of SV40-like (simian virus 40) particles. The self-assembly of this new type of nanoparticles is highly reproducible and efficient. The structure of these particles was determined by cryo-EM. The capsid forms a regular SV40 lattice of T = 7d icosahedral symmetry and the structural features of encapsulated DNA origami are fully visible. These particles are a promising biomaterial for use in various medical applications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4648.map.gz emd_4648.map.gz | 27.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4648-v30.xml emd-4648-v30.xml emd-4648.xml emd-4648.xml | 9.3 KB 9.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_4648.png emd_4648.png | 159.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4648 http://ftp.pdbj.org/pub/emdb/structures/EMD-4648 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4648 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4648 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4648_validation.pdf.gz emd_4648_validation.pdf.gz | 245 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4648_full_validation.pdf.gz emd_4648_full_validation.pdf.gz | 244.1 KB | 表示 | |
| XML形式データ |  emd_4648_validation.xml.gz emd_4648_validation.xml.gz | 6.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4648 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4648 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4648 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4648 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4648.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4648.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Empty capsid from DNA origami-filled SV40 (VP1) preparation. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Simian virus 40
| 全体 | 名称:  Simian virus 40 (ウイルス) Simian virus 40 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Simian virus 40
| 超分子 | 名称: Simian virus 40 / タイプ: virus / ID: 1 / 親要素: 0 / NCBI-ID: 10633 / 生物種: Simian virus 40 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| Host system | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 96 % / チャンバー内温度: 293 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 100.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / 解像度のタイプ: BY AUTHOR / 解像度: 6.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0 beta2) / 使用した粒子像数: 13818 |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















