+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | rat GluN1a-2B Fab 003-102 local refinement | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Channel / heterotetramer / receptor / antibody / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to curcumin / cellular response to corticosterone stimulus / cellular response to magnesium starvation / sensory organ development / pons maturation / positive regulation of Schwann cell migration / regulation of cell communication / EPHB-mediated forward signaling / sensitization / auditory behavior ...cellular response to curcumin / cellular response to corticosterone stimulus / cellular response to magnesium starvation / sensory organ development / pons maturation / positive regulation of Schwann cell migration / regulation of cell communication / EPHB-mediated forward signaling / sensitization / auditory behavior / Assembly and cell surface presentation of NMDA receptors / response to hydrogen sulfide / olfactory learning / conditioned taste aversion / dendritic branch / regulation of respiratory gaseous exchange / response to other organism / protein localization to postsynaptic membrane / regulation of ARF protein signal transduction / transmitter-gated monoatomic ion channel activity / fear response / apical dendrite / positive regulation of inhibitory postsynaptic potential / suckling behavior / response to methylmercury / response to glycine / propylene metabolic process / response to manganese ion / response to carbohydrate / interleukin-1 receptor binding / cellular response to dsRNA / negative regulation of dendritic spine maintenance / cellular response to lipid / response to growth hormone / regulation of monoatomic cation transmembrane transport / heterocyclic compound binding / NMDA glutamate receptor activity / positive regulation of glutamate secretion / Synaptic adhesion-like molecules / RAF/MAP kinase cascade / voltage-gated monoatomic cation channel activity / response to glycoside / NMDA selective glutamate receptor complex / neurotransmitter receptor complex / ligand-gated sodium channel activity / glutamate binding / response to morphine / calcium ion transmembrane import into cytosol / regulation of axonogenesis / neuromuscular process / regulation of dendrite morphogenesis / protein heterotetramerization / male mating behavior / glycine binding / regulation of synapse assembly / response to amine / receptor clustering / parallel fiber to Purkinje cell synapse / small molecule binding / regulation of cAMP/PKA signal transduction / startle response / positive regulation of reactive oxygen species biosynthetic process / monoatomic cation transmembrane transport / behavioral response to pain / positive regulation of calcium ion transport into cytosol / response to magnesium ion / regulation of postsynaptic membrane potential / regulation of neuron apoptotic process / regulation of MAPK cascade / action potential / associative learning / cellular response to glycine / extracellularly glutamate-gated ion channel activity / monoatomic cation transport / excitatory synapse / social behavior / positive regulation of dendritic spine maintenance / response to electrical stimulus / regulation of neuronal synaptic plasticity / monoatomic ion channel complex / positive regulation of excitatory postsynaptic potential / glutamate receptor binding / Unblocking of NMDA receptors, glutamate binding and activation / response to mechanical stimulus / long-term memory / detection of mechanical stimulus involved in sensory perception of pain / positive regulation of synaptic transmission, glutamatergic / behavioral fear response / synaptic cleft / multicellular organismal response to stress / neuron development / prepulse inhibition / postsynaptic density, intracellular component / phosphatase binding / monoatomic cation channel activity / calcium ion homeostasis / response to fungicide / glutamate-gated receptor activity / cell adhesion molecule binding / D2 dopamine receptor binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.95 Å | |||||||||
 データ登録者 データ登録者 | Michalski K / Furukawa H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural and functional mechanisms of anti-NMDAR autoimmune encephalitis. 著者: Kevin Michalski / Taha Abdulla / Sam Kleeman / Lars Schmidl / Ricardo Gómez / Noriko Simorowski / Francesca Vallese / Harald Prüss / Manfred Heckmann / Christian Geis / Hiro Furukawa /   要旨: Autoantibodies against neuronal membrane proteins can manifest in autoimmune encephalitis, inducing seizures, cognitive dysfunction and psychosis. Anti-N-methyl-D-aspartate receptor (NMDAR) ...Autoantibodies against neuronal membrane proteins can manifest in autoimmune encephalitis, inducing seizures, cognitive dysfunction and psychosis. Anti-N-methyl-D-aspartate receptor (NMDAR) encephalitis is the most dominant autoimmune encephalitis; however, insights into how autoantibodies recognize and alter receptor functions remain limited. Here we determined structures of human and rat NMDARs bound to three distinct patient-derived antibodies using single-particle electron cryo-microscopy. These antibodies bind different regions within the amino-terminal domain of the GluN1 subunit. Through electrophysiology, we show that all three autoantibodies acutely and directly reduced NMDAR channel functions in primary neurons. Antibodies show different stoichiometry of binding and antibody-receptor complex formation, which in one antibody, 003-102, also results in reduced synaptic localization of NMDARs. These studies demonstrate mechanisms of diverse epitope recognition and direct channel regulation of anti-NMDAR autoantibodies underlying autoimmune encephalitis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43559.map.gz emd_43559.map.gz | 229.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43559-v30.xml emd-43559-v30.xml emd-43559.xml emd-43559.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_43559.png emd_43559.png | 69.4 KB | ||
| Filedesc metadata |  emd-43559.cif.gz emd-43559.cif.gz | 6.6 KB | ||
| その他 |  emd_43559_half_map_1.map.gz emd_43559_half_map_1.map.gz emd_43559_half_map_2.map.gz emd_43559_half_map_2.map.gz | 226.4 MB 226.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43559 http://ftp.pdbj.org/pub/emdb/structures/EMD-43559 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43559 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43559 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43559_validation.pdf.gz emd_43559_validation.pdf.gz | 937.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43559_full_validation.pdf.gz emd_43559_full_validation.pdf.gz | 937.5 KB | 表示 | |
| XML形式データ |  emd_43559_validation.xml.gz emd_43559_validation.xml.gz | 15.8 KB | 表示 | |
| CIF形式データ |  emd_43559_validation.cif.gz emd_43559_validation.cif.gz | 18.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43559 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43559 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43559 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43559 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8vvhMC  8vuhC  8vujC  8vulC  8vunC  8vuqC  8vurC  8vusC  8vutC  8vuuC  8vuvC  8vuyC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43559.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43559.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.856 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_43559_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
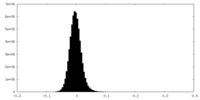
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_43559_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
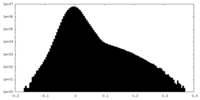
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : rat GluN1a-2B Fab 003-102 local refinement
| 全体 | 名称: rat GluN1a-2B Fab 003-102 local refinement |
|---|---|
| 要素 |
|
-超分子 #1: rat GluN1a-2B Fab 003-102 local refinement
| 超分子 | 名称: rat GluN1a-2B Fab 003-102 local refinement / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Glutamate receptor ionotropic, NMDA 1
| 分子 | 名称: Glutamate receptor ionotropic, NMDA 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 41.352012 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KIVNIGAVLS TRKHEQMFRE AVNQANKRHG SWKIQLQATS VTHKPNAIQM ALSVCEDLIS SQVYAILVSH PPTPNDHFTP TPVSYTAGF YRIPVLGLTT RMSIYSDKSI HLSFLRTVPP YSHQSSVWFE MMRVYNWNHI ILLVSDDHEG RAAQKRLETL L EERESKAE ...文字列: KIVNIGAVLS TRKHEQMFRE AVNQANKRHG SWKIQLQATS VTHKPNAIQM ALSVCEDLIS SQVYAILVSH PPTPNDHFTP TPVSYTAGF YRIPVLGLTT RMSIYSDKSI HLSFLRTVPP YSHQSSVWFE MMRVYNWNHI ILLVSDDHEG RAAQKRLETL L EERESKAE KVLQFDPGTK NVTALLMEAR ELEARVIILS ASEDDAATVY RAAAMLDMTG SGYVWLVGER EISGNALRYA PD GIIGLQL INGKNESAHI SDAVGVVAQA VHELLEKENI TDPPRGCVGN TNIWKTGPLF KRVLMSSKYA DGVTGRVEFN EDG DRKFAQ YSIMNLQNRK LVQVGIYNGT HVIPNDRKII WPGGETEKPR GYQ UniProtKB: Glutamate receptor ionotropic, NMDA 1 |
-分子 #2: Glutamate receptor ionotropic, NMDA 2B
| 分子 | 名称: Glutamate receptor ionotropic, NMDA 2B / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 40.103426 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SIGIAVILVG TSDEVAIKDA HEKDDFHHLS VVPRVELVAM NETDPKSIIT RICDLMSDRK IQGVVFADDT DQEAIAQILD FISAQTLTP ILGIHGGSSM IMADKDESSM FFQFGPSIEQ QASVMLNIME EYDWYIFSIV TTYFPGYQDF VNKIRSTIEN S FVGWELEE ...文字列: SIGIAVILVG TSDEVAIKDA HEKDDFHHLS VVPRVELVAM NETDPKSIIT RICDLMSDRK IQGVVFADDT DQEAIAQILD FISAQTLTP ILGIHGGSSM IMADKDESSM FFQFGPSIEQ QASVMLNIME EYDWYIFSIV TTYFPGYQDF VNKIRSTIEN S FVGWELEE VLLLDMSLDD GDSKIQNQLK KLQSPIILLY CTKEEATYIF EVANSVGLTG YGYTWIVPSL VAGDTDTVPS EF PTGLISV SYDEWDYGLP ARVRDGIAII TTAASDMLSE HSFIPEPKSS CYNTHEKRIY QSNMLNRYLI NVTFEGRDLS FSE EGYQMH PKLVIILLNK ERKWERVGKW KDKSLQMK UniProtKB: Glutamate receptor ionotropic, NMDA 2B |
-分子 #3: 003-102 Heavy
| 分子 | 名称: 003-102 Heavy / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.477862 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LQLQESGPGL VKPSQTLSLT CTVSGGSISS SNWWSWVRQP PGKGLEWIGE IYHSGNTNYN PSLKSRVTVS VDKSKNQFSL KLTSVTAAD TAVYYCARDV SGGVNWFDPW GQGTLV |
-分子 #4: 003-102 Light
| 分子 | 名称: 003-102 Light / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.582475 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: NFMLTQPHSV SESPGKTVTI SCTRSSGSIA SNYVQWYQQR PGSAPTTVIY EDNQRPSGVP DRFSGSIDSS SNSASLTISG LKTEDEADY YCQSYDSSTV VFGGGTKLT |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)