+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Locally refined cryoEM structure of G protein heterotrimer from beta-2-adrenergic receptor in complex with GTP-bound Gs heterotrimer (transition intermediate #1 of 20) | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | GPCR / Adrenergic / Receptor / G protein / SIGNALING PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Papasergi-Scott MM / Skiniotis G | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Time-resolved cryo-EM of G-protein activation by a GPCR. 著者: Makaía M Papasergi-Scott / Guillermo Pérez-Hernández / Hossein Batebi / Yang Gao / Gözde Eskici / Alpay B Seven / Ouliana Panova / Daniel Hilger / Marina Casiraghi / Feng He / Luis Maul / ...著者: Makaía M Papasergi-Scott / Guillermo Pérez-Hernández / Hossein Batebi / Yang Gao / Gözde Eskici / Alpay B Seven / Ouliana Panova / Daniel Hilger / Marina Casiraghi / Feng He / Luis Maul / Peter Gmeiner / Brian K Kobilka / Peter W Hildebrand / Georgios Skiniotis /    要旨: G-protein-coupled receptors (GPCRs) activate heterotrimeric G proteins by stimulating guanine nucleotide exchange in the Gα subunit. To visualize this mechanism, we developed a time-resolved cryo-EM ...G-protein-coupled receptors (GPCRs) activate heterotrimeric G proteins by stimulating guanine nucleotide exchange in the Gα subunit. To visualize this mechanism, we developed a time-resolved cryo-EM approach that examines the progression of ensembles of pre-steady-state intermediates of a GPCR-G-protein complex. By monitoring the transitions of the stimulatory G protein in complex with the β-adrenergic receptor at short sequential time points after GTP addition, we identified the conformational trajectory underlying G-protein activation and functional dissociation from the receptor. Twenty structures generated from sequential overlapping particle subsets along this trajectory, compared to control structures, provide a high-resolution description of the order of main events driving G-protein activation in response to GTP binding. Structural changes propagate from the nucleotide-binding pocket and extend through the GTPase domain, enacting alterations to Gα switch regions and the α5 helix that weaken the G-protein-receptor interface. Molecular dynamics simulations with late structures in the cryo-EM trajectory support that enhanced ordering of GTP on closure of the α-helical domain against the nucleotide-bound Ras-homology domain correlates with α5 helix destabilization and eventual dissociation of the G protein from the GPCR. These findings also highlight the potential of time-resolved cryo-EM as a tool for mechanistic dissection of GPCR signalling events. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40156.map.gz emd_40156.map.gz | 127.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40156-v30.xml emd-40156-v30.xml emd-40156.xml emd-40156.xml | 18.4 KB 18.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
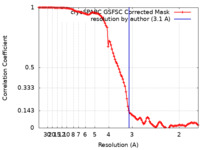
| FSC (解像度算出) |  emd_40156_fsc.xml emd_40156_fsc.xml | 10.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40156.png emd_40156.png | 113.1 KB | ||
| マスクデータ |  emd_40156_msk_1.map emd_40156_msk_1.map | 134.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40156.cif.gz emd-40156.cif.gz | 5 KB | ||
| その他 |  emd_40156_additional_1.map.gz emd_40156_additional_1.map.gz emd_40156_half_map_1.map.gz emd_40156_half_map_1.map.gz emd_40156_half_map_2.map.gz emd_40156_half_map_2.map.gz | 67.4 MB 125.1 MB 125.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40156 http://ftp.pdbj.org/pub/emdb/structures/EMD-40156 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40156 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40156 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40156_validation.pdf.gz emd_40156_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40156_full_validation.pdf.gz emd_40156_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_40156_validation.xml.gz emd_40156_validation.xml.gz | 19.5 KB | 表示 | |
| CIF形式データ |  emd_40156_validation.cif.gz emd_40156_validation.cif.gz | 25 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40156 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40156 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40156 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40156 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8gdzC  8ge1C  8ge2C  8ge3C  8ge4C  8ge5C  8ge6C  8ge7C  8ge8C  8ge9C  8geaC  8gebC  8gecC  8gedC  8geeC  8gefC  8gegC  8gehC  8geiC  8gejC  8gfvC  8gfwC  8gfxC  8gfyC  8gfzC  8gg0C  8gg1C  8gg2C  8gg3C  8gg4C  8gg5C  8gg6C  8gg7C  8gg8C  8gg9C  8ggaC  8ggbC  8ggcC  8ggeC  8ggfC  8ggiC  8ggjC  8ggkC  8gglC  8ggmC  8ggnC  8ggoC  8ggpC  8ggqC  8ggrC  8ggsC  8ggtC  8gguC  8ggvC  8ggwC  8ggxC  8ggyC  8ggzC  8gh0C  8gh1C  8unlC  8unmC  8unnC  8unoC  8unpC  8unqC  8unrC  8unsC  8untC  8unuC  8unvC  8unwC  8unxC  8unyC  8unzC  8uo0C  8uo1C  8uo2C  8uo3C  8uo4C C: 同じ文献を引用 ( |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40156.map.gz / 形式: CCP4 / 大きさ: 134.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40156.map.gz / 形式: CCP4 / 大きさ: 134.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8677 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40156_msk_1.map emd_40156_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unsharpened map
| ファイル | emd_40156_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map
| ファイル | emd_40156_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map
| ファイル | emd_40156_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of beta-2 adrenergic receptor and Gs heterotrimer with GTP
| 全体 | 名称: Complex of beta-2 adrenergic receptor and Gs heterotrimer with GTP |
|---|---|
| 要素 |
|
-超分子 #1: Complex of beta-2 adrenergic receptor and Gs heterotrimer with GTP
| 超分子 | 名称: Complex of beta-2 adrenergic receptor and Gs heterotrimer with GTP タイプ: complex / ID: 1 / 親要素: 0 詳細: Combined datasets of the protein complex from samples plunge frozen at 5 sec, 10 sec, or 17 sec after GTP addition to the sample. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 詳細: GTP was added just prior to freezing at 5 sec, 10 sec, or 17 sec before plunging. |
|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV 詳細: GTP was added just prior to freezing at 5 sec, 10 sec, or 17 sec before plunging.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 詳細: The map is the result of combining multiple datasets: cryo-EM imaging of the beta-2AR-Gs + GTP (5 sec) complex was performed on a Titan Krios electron microscope equipped with a K3 Summit ...詳細: The map is the result of combining multiple datasets: cryo-EM imaging of the beta-2AR-Gs + GTP (5 sec) complex was performed on a Titan Krios electron microscope equipped with a K3 Summit direct electron detector (Gatan). The microscope was operated at 300 kV accelerating voltage, with a nominal magnification of 105,000x in counting mode resulting in a magnified pixel size of 0.8677 Angstrom. A total exposure of 60.48 electrons/ Angstrom^2 over 63 frames with defocus ranging from -1.0 - -2.0 micrometers was used. Cryo-EM imaging of beta-2AR-Gs + GTP (10 sec) complex was performed on four separate grids over three collection sessions. The microscope was operated at 300 kV accelerating voltage, with a magnification at the camera of 58,679x in counting mode resulting in a magnified pixel size of 0.8521 Angstrom. For the first and second grids, movies were obtained at an exposure rate of 21.13 electrons/Angstrum^2/sec with defocus ranging from -0.4 - -2.0 micrometers. The total exposure time was 2.717 sec over 77 frames per movie stack. For an additional collection of the first grid, movies were obtained at an exposure rate of 20.95 electrons/ Angstrum^2/sec with defocus ranging from -0.4 -2.0 micrometers. The total exposure time was 2.717 sec over 77 frames per movie stack. For the third and fourth grids, movies were obtained at an exposure rate of 30.71 electrons/ Angstrum^2/sec with defocus ranging from -0.5 - -1.6 micrometers. The total exposure time was 2.008 sec over 79 frames per movie stack. Cryo-EM imaging of beta-2AR-Gs + GTP (17 sec) was performed on a Titan Krios equipped with a post-column energy filter, with a magnification of 105,000x in counting mode resulting in a magnified pixel size of 0.8677 Angstrom. Movies were obtained at an exposure rate of 32.46 electrons/Angstrum^2/sec with defocus ranging from -0.4 - -0.9 micrometers. The total exposure time was 1.999 sec over 79 frames per movie stack. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.4 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


































































































































































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)