+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
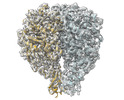
| タイトル | Single particle cryo-EM of the P140-P110 heterodimer of Mycoplasma genitalium at 3.3 Angstrom resolution. | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Adhesion / Mycoplasma genitalium / CELL ADHESION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Mycoplasmoides genitalium G37 (バクテリア) Mycoplasmoides genitalium G37 (バクテリア) | |||||||||
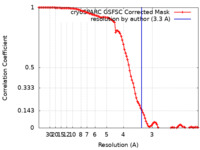
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Sprankel L / Scheffer MP / Frangakis AS | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2023 ジャーナル: PLoS Pathog / 年: 2023タイトル: Cryo-electron tomography reveals the binding and release states of the major adhesion complex from Mycoplasma genitalium. 著者: Lasse Sprankel / Margot P Scheffer / Sina Manger / Utz H Ermel / Achilleas S Frangakis /  要旨: The nap particle is an immunogenic surface adhesion complex from Mycoplasma genitalium. It is essential for motility and responsible for binding sialylated oligosaccharides on the surface of the host ...The nap particle is an immunogenic surface adhesion complex from Mycoplasma genitalium. It is essential for motility and responsible for binding sialylated oligosaccharides on the surface of the host cell. The nap particle is composed of two P140-P110 heterodimers, the structure of which was recently solved. However, the interpretation of the mechanism by which the mycoplasma cells orchestrate adhesion remained challenging. Here, we provide cryo-electron tomography structures at ~11 Å resolution, which allow for the distinction between the bound and released state of the nap particle, displaying the in vivo conformational states. Fitting of the atomically resolved structures reveals that bound sialylated oligosaccharides are stabilized by both P110 and P140. Movement of the stalk domains allows for the transfer of conformational changes from the interior of the cell to the binding pocket, thus having the capability of an active release process. It is likely that the same mechanism can be transferred to other Mycoplasma species that belong to the pneumoniae cluster. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17587.map.gz emd_17587.map.gz | 97.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17587-v30.xml emd-17587-v30.xml emd-17587.xml emd-17587.xml | 21.7 KB 21.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17587_fsc.xml emd_17587_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17587.png emd_17587.png | 148.5 KB | ||
| マスクデータ |  emd_17587_msk_1.map emd_17587_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17587.cif.gz emd-17587.cif.gz | 7.6 KB | ||
| その他 |  emd_17587_half_map_1.map.gz emd_17587_half_map_1.map.gz emd_17587_half_map_2.map.gz emd_17587_half_map_2.map.gz | 95.6 MB 95.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17587 http://ftp.pdbj.org/pub/emdb/structures/EMD-17587 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17587 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17587 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17587_validation.pdf.gz emd_17587_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17587_full_validation.pdf.gz emd_17587_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_17587_validation.xml.gz emd_17587_validation.xml.gz | 18.1 KB | 表示 | |
| CIF形式データ |  emd_17587_validation.cif.gz emd_17587_validation.cif.gz | 23.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17587 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17587 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17587 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17587 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17587.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17587.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17587_msk_1.map emd_17587_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
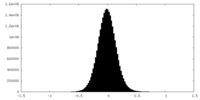
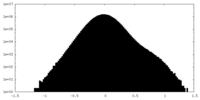
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17587_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17587_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : P140-P110 Heterodimer
| 全体 | 名称: P140-P110 Heterodimer |
|---|---|
| 要素 |
|
-超分子 #1: P140-P110 Heterodimer
| 超分子 | 名称: P140-P110 Heterodimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Mgp-operon protein 3
| 超分子 | 名称: Mgp-operon protein 3 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Mycoplasmoides genitalium G37 (バクテリア) Mycoplasmoides genitalium G37 (バクテリア) |
-超分子 #3: Adhesin P1
| 超分子 | 名称: Adhesin P1 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Mycoplasmoides genitalium G37 (バクテリア) Mycoplasmoides genitalium G37 (バクテリア) |
-分子 #1: Mgp-operon protein 3
| 分子 | 名称: Mgp-operon protein 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycoplasmoides genitalium G37 (バクテリア) Mycoplasmoides genitalium G37 (バクテリア) |
| 分子量 | 理論値: 115.382062 KDa |
| 組換発現 | 生物種:  Mycoplasmoides genitalium G37 (バクテリア) Mycoplasmoides genitalium G37 (バクテリア) |
| 配列 | 文字列: MKTMRKQIYK KAYWLLLPFL PLALANTFLV KEDSKNVTAY TPFATPITDS KSDLVSLAQL DSSYQIADQT IHNTNLFVLF KSRDVKVKY ESSGSNNISF DSTSQGEKPS YVVEFTNSTN IGIKWTMVKK YQLDVPNVSS DMNQVLKNLI LEQPLTKYTL N SSLAKEKG ...文字列: MKTMRKQIYK KAYWLLLPFL PLALANTFLV KEDSKNVTAY TPFATPITDS KSDLVSLAQL DSSYQIADQT IHNTNLFVLF KSRDVKVKY ESSGSNNISF DSTSQGEKPS YVVEFTNSTN IGIKWTMVKK YQLDVPNVSS DMNQVLKNLI LEQPLTKYTL N SSLAKEKG KTQREVHLGS GQANQWTSQR NQHDLNNNPS PNASTGFKLT TGNAYRKLSE SWPIYEPIDG TKQGKGKDSS GW SSTEENE AKNDAPSVSG GGSSSGTFNK YLNTKQALES IGILFDDQTP RNVITQLYYA STSKLAVTNN HIVVMGNSFL PSM WYWVVE RSAQENASNK PTWFANTNLD WGEDKQKQFV ENQLGYKETT STNSHNFHSK SFTQPAYLIS GIDSVNDQII FSGF KAGSV GYDSSSSSSS SSSSSTKDQA LAWSTTTSLD SKTGYKDLVT NDTGLNGPIN GSFSIQDTFS FVVPYSGNHT NNGTT GPIK TAYPVKKDQK STVKINSLIN ATPLNSYGDE GIGVFDALGL NYNFKSNQER LPSRTDQIFV YGIVSPNELR SAKSSA DST GSDTKVNWSN TQSRYLPVPY NYSEGIIDAD GFKRPENRGA SVTTFSGLKS IAPDGFANSI ANFSVGLKAG IDPNPVM SG KKANYGAVVL TRGGVVRLNF NPGNDSLLST TDNNIAPISF SFTPFTAAES AVDLTTFKEV TYNQESGLWS YIFDSSLK P SHDGKQTPVT DNMGFSVITV SRTGIELNQD QATTTLDVAP SALAVQSGIQ STTQTLTGVL PLSEEFSAVI AKDSDQNKI DIYKNNNGLF EIDTQLSNSV ATNNGGLAPS YTENRVDAWG KVEFADNSVL QARNLVDKTV DEIINTPEIL NSFFRFTPAF EDQKATLVA TKQSDTSLSV SPRIQFLDGN FYDLNSTIAG VPLNIGFPSR VFAGFAALPA WVIPVSVGSS VGILFILLVL G LGIGIPMY RVRKLQDASF VNVFKKVDTL TTAVGSVYKK IITQTGVVKK APSALKAANP SVKKPAAFLK PPVQPPSKPE GE QKAVEVK SEETKSHHHH HH UniProtKB: Mgp-operon protein 3 |
-分子 #2: Adhesin P1
| 分子 | 名称: Adhesin P1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycoplasmoides genitalium G37 (バクテリア) Mycoplasmoides genitalium G37 (バクテリア) |
| 分子量 | 理論値: 159.809297 KDa |
| 配列 | 文字列: MHQPKKRLAK KSWAFLTAAL TLGVITGVGG YFLFNQNKQR SSVSNFAYQP KQLSVKHQQA VDETLTPWTW NNNNFSSLKI TGENPGSFG LVRSQNDNLN ISSVTKNSSD DNLKYLNAVE KYLDGQQNFA IRRYDNNGRA LYDINLAKME NPSTVQRGLN G EPIFDPFK ...文字列: MHQPKKRLAK KSWAFLTAAL TLGVITGVGG YFLFNQNKQR SSVSNFAYQP KQLSVKHQQA VDETLTPWTW NNNNFSSLKI TGENPGSFG LVRSQNDNLN ISSVTKNSSD DNLKYLNAVE KYLDGQQNFA IRRYDNNGRA LYDINLAKME NPSTVQRGLN G EPIFDPFK GFGLTGNAPT DWNEIKGKVP VEVVQSPHSP NLYFVLLVPK VALEYHNLNN QVVKESLEVK ATQSSFNPTQ RL QKDSPVK DSSKQGEKLS ETTASSMSSG MATSTRAKAL KVEVERGSQS DSLLKNDFAK KPLKHKNSSG EVKLEAEKEF TEA WKPLLT TDQIAREKGM GATVVSFYDA PYSENHTAFG LVDHIDPKKM VENYPPSWKT PKWNHHGIWD YNARNLLLQT TGFF NPRRH PEWFDEGQAK ADNTSPGFKV GDTDHKKDGF KKNSSSPIAL PFEAYFANIG NMVAIGNSVF IFGGNGHATK MFTTN PLSI GVFRIKYTDN FSKSSVTGWP YAVLFGGLIN PQTNGLKDLP LGTNRWFEYV PRMAVSGVKW VGNQLVLAGT LTMGDT ATV PRLKYDQLEK HLNLVAQGQG LLREDLQIFT PYGWANRPDI PVGAWLQDEM GSKFGPHYFL NNPDIQDNVN NDTVEAL IS SYKNTDKLKH VYPYRYSGLY AWQLFNWSNK LTNTPLSANF VNENSYAPNS LFAAILNEDL LTGLSDKIFY GKENEFAE N EADRFNQLLS LNPNPNTNWA RYLNVVQRFT TGPNLDSSTF DQFLDFLPWI GNGKPFSNSP SPSTSASSST PLPTFSNIN VGVKSMITQH LNKENTRWVF IPNFSPDIWT GAGYRVQSAN QKNGIPFEQV KPSNNSTPFD PNSDDNKVTP SGGSSKPTTY PALPNSISP TSDWINALTF TNKNNPQRNQ LLLRSLLGTI PVLINKSGDS NDQFNKDSEQ KWDKTETNEG NLPGFGEVNG L YNAALLHT YGFFGTNTNS TDPKIGFKAD SSSSSSSTLV GSGLNWTSQD VGNLVVINDT SFGFQLGGWF ITFTDFIRPR TG YLGITLS SLQDQTIIWA DQPWTSFKGS YLDSDGTPKS LWDPTALKSL PNSSTTYDTN PTLSPSFQLY QPNKVKAYQT TNT YNKLIE PVDATSAATN MTSLLKLLTT KNIKAKLGKG TASSQGNNNG GGVSQTINTI TTTGNISEGL KEETSIQAET LKKF FDSKQ NNKSEIGIGD STFTKMDGKL TGVVSTPLVN LINGQGATSD SDTEKISFKP GNQIDFNRLF TLPVTELFDP NTMFV YDQY VPLLVNLPSG FDQASIRLKV ISYSVENQTL GVRLEFKDPQ TQQFIPVLNA SSTGPQTVFQ PFNQWADYVL PLIVTV PIV VIILSVTLGL TIGIPMHRNK KALQAGFDLS NKKVDVLTKA VGSVFKEIIN RTGISNAPKK LKQATPTKPT PKTPPKP PV KQ UniProtKB: Adhesin P1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.025 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum SE / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 4.0 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 130000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL | ||||||
| 得られたモデル |  PDB-8pbx: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)