+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13590 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the dimeric Rhodobacter sphaeroides RC-LH1 core complex at 2.9 A: the structural basis for dimerisation | ||||||||||||
 マップデータ マップデータ | light harvesting core complex | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | light harvesting complex / photosynthesis / Cryo-EM / purple bacteria / RC-LH1 / RC-LH1-PufXYZ / dimer / dimeric core complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthetic electron transport in photosystem II / : / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Cereibacter sphaeroides 2.4.1 (バクテリア) Cereibacter sphaeroides 2.4.1 (バクテリア) | ||||||||||||
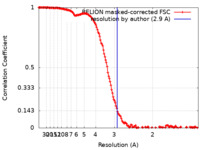
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | ||||||||||||
 データ登録者 データ登録者 | Qian P / Hunter CN | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Biochem J / 年: 2021 ジャーナル: Biochem J / 年: 2021タイトル: Cryo-EM structure of the dimeric Rhodobacter sphaeroides RC-LH1 core complex at 2.9 Å: the structural basis for dimerisation. 著者: Pu Qian / Tristan I Croll / Andrew Hitchcock / Philip J Jackson / Jack H Salisbury / Pablo Castro-Hartmann / Kasim Sader / David J K Swainsbury / C Neil Hunter /   要旨: The dimeric reaction centre light-harvesting 1 (RC-LH1) core complex of Rhodobacter sphaeroides converts absorbed light energy to a charge separation, and then it reduces a quinone electron and ...The dimeric reaction centre light-harvesting 1 (RC-LH1) core complex of Rhodobacter sphaeroides converts absorbed light energy to a charge separation, and then it reduces a quinone electron and proton acceptor to a quinol. The angle between the two monomers imposes a bent configuration on the dimer complex, which exerts a major influence on the curvature of the membrane vesicles, known as chromatophores, where the light-driven photosynthetic reactions take place. To investigate the dimerisation interface between two RC-LH1 monomers, we determined the cryogenic electron microscopy structure of the dimeric complex at 2.9 Å resolution. The structure shows that each monomer consists of a central RC partly enclosed by a 14-subunit LH1 ring held in an open state by PufX and protein-Y polypeptides, thus enabling quinones to enter and leave the complex. Two monomers are brought together through N-terminal interactions between PufX polypeptides on the cytoplasmic side of the complex, augmented by two novel transmembrane polypeptides, designated protein-Z, that bind to the outer faces of the two central LH1 β polypeptides. The precise fit at the dimer interface, enabled by PufX and protein-Z, by C-terminal interactions between opposing LH1 αβ subunits, and by a series of interactions with a bound sulfoquinovosyl diacylglycerol lipid, bring together each monomer creating an S-shaped array of 28 bacteriochlorophylls. The seamless join between the two sets of LH1 bacteriochlorophylls provides a path for excitation energy absorbed by one half of the complex to migrate across the dimer interface to the other half. #1:  ジャーナル: Acta Crystallogr., Sect. D: Biol. Crystallogr. ジャーナル: Acta Crystallogr., Sect. D: Biol. Crystallogr.年: 2018 タイトル: Real-space refinement in PHENIX for cryo-EM and crystallography 著者: Qian P / Hunter CN | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13590.map.gz emd_13590.map.gz | 51.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13590-v30.xml emd-13590-v30.xml emd-13590.xml emd-13590.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13590_fsc.xml emd_13590_fsc.xml | 15.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13590.png emd_13590.png | 72.3 KB | ||
| Filedesc metadata |  emd-13590.cif.gz emd-13590.cif.gz | 7.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13590 http://ftp.pdbj.org/pub/emdb/structures/EMD-13590 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13590 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13590 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13590_validation.pdf.gz emd_13590_validation.pdf.gz | 457.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13590_full_validation.pdf.gz emd_13590_full_validation.pdf.gz | 456.8 KB | 表示 | |
| XML形式データ |  emd_13590_validation.xml.gz emd_13590_validation.xml.gz | 15.1 KB | 表示 | |
| CIF形式データ |  emd_13590_validation.cif.gz emd_13590_validation.cif.gz | 20.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13590 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13590 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13590 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13590 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13590.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13590.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | light harvesting core complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.65 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Light harvesting core complex
+超分子 #1: Light harvesting core complex
+分子 #1: LH1-alpha
+分子 #2: LH1-beta
+分子 #3: RC-H
+分子 #4: RC-L
+分子 #5: Reaction center protein M chain
+分子 #6: PufZ
+分子 #7: PufY
+分子 #8: PufX
+分子 #9: BACTERIOCHLOROPHYLL A
+分子 #10: 3,4-DIHYDROSPHEROIDENE
+分子 #11: 1,2-Distearoyl-sn-glycerophosphoethanolamine
+分子 #12: (2R,5R,11R,14R)-5,8,11-trihydroxy-5,11-dioxido-17-oxo-2,14-bis(te...
+分子 #13: UBIQUINONE-1
+分子 #14: UBIQUINONE-10
+分子 #15: BACTERIOPHEOPHYTIN A
+分子 #16: 1,2-DI-O-ACYL-3-O-[6-DEOXY-6-SULFO-ALPHA-D-GLUCOPYRANOSYL]-SN-GLYCEROL
+分子 #17: DODECYL-BETA-D-MALTOSIDE
+分子 #18: FE (III) ION
+分子 #19: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 / 構成要素 - 濃度: 20.0 mMol / 構成要素 - 式: HEPES / 詳細: 20 mM HEPES, pH 7.8, 0.03% beta-DDM |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 99 % / チャンバー内温度: 277 K / 詳細: QF R1.2/1.3 300 mesh Cu grid. |
| 詳細 | In 20 mM HEPES, pH 7.8, 0.03% beta-DDM buffer solution |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最高: 80.0 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 5058 / 平均露光時間: 12.21 sec. / 平均電子線量: 45.36 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 120000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)