+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8c1p | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
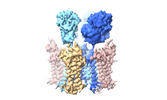
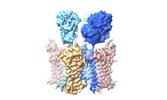
| タイトル | Active state homomeric GluA1 AMPA receptor in complex with TARP gamma 3 | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | MEMBRANE PROTEIN / AMPAR / ion channels / neurotransmission | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Cargo concentration in the ER / cellular response to amine stimulus / axonal spine / COPII-mediated vesicle transport / positive regulation of membrane potential / cellular response to ammonium ion / neurotransmitter receptor transport, postsynaptic endosome to lysosome / LGI-ADAM interactions / myosin V binding / neurotransmitter receptor activity involved in regulation of postsynaptic cytosolic calcium ion concentration ...Cargo concentration in the ER / cellular response to amine stimulus / axonal spine / COPII-mediated vesicle transport / positive regulation of membrane potential / cellular response to ammonium ion / neurotransmitter receptor transport, postsynaptic endosome to lysosome / LGI-ADAM interactions / myosin V binding / neurotransmitter receptor activity involved in regulation of postsynaptic cytosolic calcium ion concentration / Trafficking of AMPA receptors / neuron spine / regulation of AMPA receptor activity / neurotransmitter receptor internalization / response to arsenic-containing substance / cellular response to dsRNA / postsynaptic neurotransmitter receptor diffusion trapping / dendritic spine membrane / Synaptic adhesion-like molecules / long-term synaptic depression / cellular response to peptide hormone stimulus / neurotransmitter receptor localization to postsynaptic specialization membrane / protein kinase A binding / spinal cord development / neuronal cell body membrane / Activation of AMPA receptors / perisynaptic space / transmission of nerve impulse / AMPA glutamate receptor activity / channel regulator activity / Trafficking of GluR2-containing AMPA receptors / response to lithium ion / immunoglobulin binding / AMPA glutamate receptor complex / adenylate cyclase binding / cellular response to organic cyclic compound / ionotropic glutamate receptor complex / excitatory synapse / neuronal action potential / asymmetric synapse / G-protein alpha-subunit binding / regulation of receptor recycling / voltage-gated calcium channel activity / regulation of postsynaptic membrane potential / Unblocking of NMDA receptors, glutamate binding and activation / postsynaptic density, intracellular component / protein targeting / positive regulation of synaptic transmission / glutamate receptor binding / long-term memory / beta-2 adrenergic receptor binding / response to electrical stimulus / glutamate-gated calcium ion channel activity / glutamate-gated receptor activity / presynaptic active zone membrane / response to fungicide / somatodendritic compartment / synapse assembly / cellular response to brain-derived neurotrophic factor stimulus / dendrite membrane / ligand-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / positive regulation of synaptic transmission, glutamatergic / response to cocaine / dendritic shaft / synaptic membrane / synaptic transmission, glutamatergic / PDZ domain binding / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / long-term synaptic potentiation / cellular response to amino acid stimulus / postsynaptic density membrane / ionotropic glutamate receptor binding / modulation of chemical synaptic transmission / neuromuscular junction / regulation of synaptic plasticity / Schaffer collateral - CA1 synapse / response to organic cyclic compound / receptor internalization / recycling endosome / response to toxic substance / cellular response to growth factor stimulus / cerebral cortex development / response to peptide hormone / small GTPase binding / synaptic vesicle membrane / recycling endosome membrane / G-protein beta-subunit binding / protein localization / cell-cell junction / synaptic vesicle / response to estradiol / presynapse / presynaptic membrane / amyloid-beta binding / early endosome membrane / cell body / scaffold protein binding / chemical synaptic transmission / postsynaptic membrane / postsynapse 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Zhang, D. / Ivica, J. / Krieger, J.M. / Ho, H. / Yamashita, K. / Cais, O. / Greger, I. | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Structural mobility tunes signalling of the GluA1 AMPA glutamate receptor. 著者: Danyang Zhang / Josip Ivica / James M Krieger / Hinze Ho / Keitaro Yamashita / Imogen Stockwell / Rozbeh Baradaran / Ondrej Cais / Ingo H Greger /   要旨: AMPA glutamate receptors (AMPARs), the primary mediators of excitatory neurotransmission in the brain, are either GluA2 subunit-containing and thus Ca-impermeable, or GluA2-lacking and Ca-permeable. ...AMPA glutamate receptors (AMPARs), the primary mediators of excitatory neurotransmission in the brain, are either GluA2 subunit-containing and thus Ca-impermeable, or GluA2-lacking and Ca-permeable. Despite their prominent expression throughout interneurons and glia, their role in long-term potentiation and their involvement in a range of neuropathologies, structural information for GluA2-lacking receptors is currently absent. Here we determine and characterize cryo-electron microscopy structures of the GluA1 homotetramer, fully occupied with TARPγ3 auxiliary subunits (GluA1/γ3). The gating core of both resting and open-state GluA1/γ3 closely resembles GluA2-containing receptors. However, the sequence-diverse N-terminal domains (NTDs) give rise to a highly mobile assembly, enabling domain swapping and subunit re-alignments in the ligand-binding domain tier that are pronounced in desensitized states. These transitions underlie the unique kinetic properties of GluA1. A GluA2 mutant (F231A) increasing NTD dynamics phenocopies this behaviour, and exhibits reduced synaptic responses, reflecting the anchoring function of the AMPAR NTD at the synapse. Together, this work underscores how the subunit-diverse NTDs determine subunit arrangement, gating properties and ultimately synaptic signalling efficiency among AMPAR subtypes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8c1p.cif.gz 8c1p.cif.gz | 504 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8c1p.ent.gz pdb8c1p.ent.gz | 377.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8c1p.json.gz 8c1p.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/c1/8c1p https://data.pdbj.org/pub/pdb/validation_reports/c1/8c1p ftp://data.pdbj.org/pub/pdb/validation_reports/c1/8c1p ftp://data.pdbj.org/pub/pdb/validation_reports/c1/8c1p | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  16379MC  8c1qC  8c1rC  8c1sC  8c2hC  8c2iC  8p3qC  8p3sC  8p3tC  8p3uC  8p3vC  8p3wC  8p3xC  8p3yC  8p3zC  8pivC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Refine code: 1
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj












