+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7048 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Type I-F CRISPR crRNA-guided Csy surveillance complex with bound target dsDNA | |||||||||
 マップデータ マップデータ | Type I-F CRISPR crRNA-guided Csy surveillance complex with bound target dsDNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-Cas / IMMUNE SYSTEM-RNA-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / defense response to virus / endonuclease activity / 加水分解酵素; エステル加水分解酵素 / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) / synthetic construct (人工物) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Guo TW / Bartesaghi A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2017 ジャーナル: Cell / 年: 2017タイトル: Cryo-EM Structures Reveal Mechanism and Inhibition of DNA Targeting by a CRISPR-Cas Surveillance Complex. 著者: Tai Wei Guo / Alberto Bartesaghi / Hui Yang / Veronica Falconieri / Prashant Rao / Alan Merk / Edward T Eng / Ashleigh M Raczkowski / Tara Fox / Lesley A Earl / Dinshaw J Patel / Sriram Subramaniam /  要旨: Prokaryotic cells possess CRISPR-mediated adaptive immune systems that protect them from foreign genetic elements, such as invading viruses. A central element of this immune system is an RNA-guided ...Prokaryotic cells possess CRISPR-mediated adaptive immune systems that protect them from foreign genetic elements, such as invading viruses. A central element of this immune system is an RNA-guided surveillance complex capable of targeting non-self DNA or RNA for degradation in a sequence- and site-specific manner analogous to RNA interference. Although the complexes display considerable diversity in their composition and architecture, many basic mechanisms underlying target recognition and cleavage are highly conserved. Using cryoelectron microscopy (cryo-EM), we show that the binding of target double-stranded DNA (dsDNA) to a type I-F CRISPR system yersinia (Csy) surveillance complex leads to large quaternary and tertiary structural changes in the complex that are likely necessary in the pathway leading to target dsDNA degradation by a trans-acting helicase-nuclease. Comparison of the structure of the surveillance complex before and after dsDNA binding, or in complex with three virally encoded anti-CRISPR suppressors that inhibit dsDNA binding, reveals mechanistic details underlying target recognition and inhibition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7048.map.gz emd_7048.map.gz | 47.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7048-v30.xml emd-7048-v30.xml emd-7048.xml emd-7048.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7048.png emd_7048.png | 173.2 KB | ||
| Filedesc metadata |  emd-7048.cif.gz emd-7048.cif.gz | 7.3 KB | ||
| その他 |  emd_7048_additional.map.gz emd_7048_additional.map.gz | 47.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7048 http://ftp.pdbj.org/pub/emdb/structures/EMD-7048 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7048 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7048 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7048_validation.pdf.gz emd_7048_validation.pdf.gz | 655.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7048_full_validation.pdf.gz emd_7048_full_validation.pdf.gz | 655.3 KB | 表示 | |
| XML形式データ |  emd_7048_validation.xml.gz emd_7048_validation.xml.gz | 6.2 KB | 表示 | |
| CIF形式データ |  emd_7048_validation.cif.gz emd_7048_validation.cif.gz | 7.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7048 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7048 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7048 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7048 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6b44MC  7049C  7050C  7051C  7052C  6anvC  6anwC  6b45C  6b46C  6b47C  6b48C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7048.map.gz / 形式: CCP4 / 大きさ: 51.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7048.map.gz / 形式: CCP4 / 大きさ: 51.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Type I-F CRISPR crRNA-guided Csy surveillance complex with bound target dsDNA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8254 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
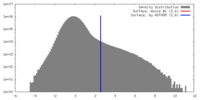
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened map of Type I-F CRISPR crRNA-guided Csy...
| ファイル | emd_7048_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map of Type I-F CRISPR crRNA-guided Csy surveillance complex with bound target dsDNA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
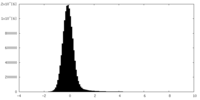
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Type I-F CRISPR crRNA-guided Csy surveillance complex with bound ...
| 全体 | 名称: Type I-F CRISPR crRNA-guided Csy surveillance complex with bound target dsDNA |
|---|---|
| 要素 |
|
-超分子 #1: Type I-F CRISPR crRNA-guided Csy surveillance complex with bound ...
| 超分子 | 名称: Type I-F CRISPR crRNA-guided Csy surveillance complex with bound target dsDNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 350 KDa |
-分子 #1: CRISPR-associated protein Csy1
| 分子 | 名称: CRISPR-associated protein Csy1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 49.338297 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSMTSPLPTP TWQELRQFIE SFIQERLQGK LDKLQPDEDD KRQTLLATHR REAWLADAAR RVGQLQLVTH TLKPIHPDAR GSNLHSLPQ APGQPGLAGS HELGDRLVSD VVGNAAALDV FKFLSLQYQG KNLLNWLTED SAEALQALSD NAEQAREWRQ A FIGITTVK ...文字列: GSMTSPLPTP TWQELRQFIE SFIQERLQGK LDKLQPDEDD KRQTLLATHR REAWLADAAR RVGQLQLVTH TLKPIHPDAR GSNLHSLPQ APGQPGLAGS HELGDRLVSD VVGNAAALDV FKFLSLQYQG KNLLNWLTED SAEALQALSD NAEQAREWRQ A FIGITTVK GAPASHSLAK QLYFPLPGSG YHLLAPLFPT SLVHHVHALL REARFGDAAK AAREARSRQE SWPHGFSEYP NL AIQKFGG TKPQNISQLN NERRGENWLL PSLPPNWQRQ NVNAPMRHSS VFEHDFGRTP EVSRLTRTLQ RFLAKTVHNN LAI RQRRAQ LVAQICDEAL QYAARLRELE PGWSATPGCQ LHDAEQLWLD PLRAQTDETF LQRRLRGDWP AEVGNRFANW LNRA VSSDS QILGSPEAAQ WSQELSKELT MFKEILEDER D UniProtKB: CRISPR-associated protein Csy1 |
-分子 #2: CRISPR-associated protein Csy2
| 分子 | 名称: CRISPR-associated protein Csy2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 36.446352 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAMSVTDPEA LLLLPRLSIQ NANAISSPLT WGFPSPGAFT GFVHALQRRV GISLDIELDG VGIVCHRFEA QISQPAGKRT KVFNLTRNP LNRDGSTAAI VEEGRAHLEV SLLLGVHGDG LDDHPAQEIA RQVQEQAGAM RLAGGSILPW CNERFPAPNA E LLMLGGSD ...文字列: MAMSVTDPEA LLLLPRLSIQ NANAISSPLT WGFPSPGAFT GFVHALQRRV GISLDIELDG VGIVCHRFEA QISQPAGKRT KVFNLTRNP LNRDGSTAAI VEEGRAHLEV SLLLGVHGDG LDDHPAQEIA RQVQEQAGAM RLAGGSILPW CNERFPAPNA E LLMLGGSD EQRRKNQRRL TRRLLPGFAL VSREALLQQH LETLRTTLPE ATTLDALLDL CRINFEPPAT SSEEEASPPD AA WQVRDKP GWLVPIPAGY NALSPLYLPG EVRNARDRET PLRFVENLFG LGEWLSPHRV AALSDLLWYH HAEPDKGLYR WST PRFVEH AIA UniProtKB: CRISPR-associated protein Csy2 |
-分子 #3: CRISPR-associated protein Csy3
| 分子 | 名称: CRISPR-associated protein Csy3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 37.781547 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAMSKPILST ASVLAFERKL DPSDALMSAG AWAQRDASQE WPAVTVREKS VRGTISNRLK TKDRDPAKLD ASIQSPNLQT VDVANLPSD ADTLKVRFTL RVLGGAGTPS ACNDAAYRDK LLQTVATYVN DQGFAELARR YAHNLANARF LWRNRVGAEA V EVRINHIR ...文字列: MAMSKPILST ASVLAFERKL DPSDALMSAG AWAQRDASQE WPAVTVREKS VRGTISNRLK TKDRDPAKLD ASIQSPNLQT VDVANLPSD ADTLKVRFTL RVLGGAGTPS ACNDAAYRDK LLQTVATYVN DQGFAELARR YAHNLANARF LWRNRVGAEA V EVRINHIR QGEVARAWRF DALAIGLRDF KADAELDALA ELIASGLSGS GHVLLEVVAF ARIGDGQEVF PSQELILDKG DK KGQKSKT LYSVRDAAAI HSQKIGNALR TIDTWYPDED GLGPIAVEPY GSVTSQGKAY RQPKQKLDFY TLLDNWVLRD EAP AVEQQH YVIANLIRGG VFGEAEEK UniProtKB: CRISPR-associated protein Csy3 |
-分子 #4: CRISPR-associated endonuclease Cas6/Csy4
| 分子 | 名称: CRISPR-associated endonuclease Cas6/Csy4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 21.629777 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAMDHYLDIR LRPDPEFPPA QLMSVLFGKL HQALVAQGGD RIGVSFPDLD ESRSRLGERL RIHASADDLR ALLARPWLEG LRDHLQFGE PAVVPHPTPY RQVSRVQAKS NPERLRRRLM RRHDLSEEEA RKRIPDTVAR ALDLPFVTLR SQSTGQHFRL F IRHGPLQV ...文字列: MAMDHYLDIR LRPDPEFPPA QLMSVLFGKL HQALVAQGGD RIGVSFPDLD ESRSRLGERL RIHASADDLR ALLARPWLEG LRDHLQFGE PAVVPHPTPY RQVSRVQAKS NPERLRRRLM RRHDLSEEEA RKRIPDTVAR ALDLPFVTLR SQSTGQHFRL F IRHGPLQV TAEEGGFTCY GLSKGGFVPW F UniProtKB: CRISPR-associated endonuclease Cas6/Csy4 |
-分子 #5: Pseudomonas aeruginosa strain SMC4485 CRISPR repeat sequence
| 分子 | 名称: Pseudomonas aeruginosa strain SMC4485 CRISPR repeat sequence タイプ: rna / ID: 5 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 19.265404 KDa |
| 配列 | 文字列: CUAAGAAAUU CACGGCGGGC UUGAUGUCCG CGUCUACCUG GUUCACUGCC GUGUAGGCAG GENBANK: GENBANK: HQ326201.1 |
-分子 #6: Target DNA strand (41-MER)
| 分子 | 名称: Target DNA strand (41-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.191747 KDa |
| 配列 | 文字列: (DC)(DA)(DG)(DG)(DT)(DA)(DG)(DA)(DC)(DG) (DC)(DG)(DG)(DA)(DC)(DA)(DT)(DC)(DA)(DA) (DG)(DC)(DC)(DC)(DG)(DC)(DC)(DG)(DT) (DG)(DA)(DA)(DG)(DG)(DT)(DG)(DA)(DT)(DG) (DA) (DC)(DT)(DG)(DC)(DA)(DC)(DA)(DG) (DA) |
-分子 #7: Non-target DNA strand (5'-D(P*CP*AP*GP*TP*CP*AP*TP*CP*AP*CP*CP*AP...
| 分子 | 名称: Non-target DNA strand (5'-D(P*CP*AP*GP*TP*CP*AP*TP*CP*AP*CP*CP*AP*A)-3') タイプ: dna / ID: 7 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 8.216318 KDa |
| 配列 | 文字列: (DT)(DC)(DT)(DG)(DT)(DG)(DC)(DA)(DG)(DT) (DC)(DA)(DT)(DC)(DA)(DC)(DC)(DA)(DA)(DT) (DT)(DT)(DA)(DT)(DT)(DT)(DA) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 詳細: 10 mM HEPES, pH 7.2, 150 mM NaCl, 2mM MgCl2, 1 mM DTT |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 98 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 3064 / 平均露光時間: 15.2 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: FREALIGN (ver. 9.11) / 使用した粒子像数: 39811 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: FREALIGN (ver. 9.11) |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: FREALIGN (ver. 9.11) |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)