+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7mua | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the adeno-associated virus 9 capsid at pH pH 5.5 in complex with terminal galactose | ||||||||||||||||||
 要素 要素 | Capsid protein VP1 | ||||||||||||||||||
 キーワード キーワード | VIRUS LIKE PARTICLE / adeno-associated virus / pH / phospholipase-A2 / capsid / gene therapy / AAV9 / dependoparvovirus / intracellular trafficking / endosome / galactose / glycan attachment | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||||||||
| 生物種 |   Adeno-associated virus 9 (アデノ随伴ウイルス) Adeno-associated virus 9 (アデノ随伴ウイルス) | ||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.68 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Penzes, J.J. / Chipman, P. / Bhattacharya, N. / Zeher, A. / Huang, R. / McKenna, R. / Agbandje-McKenna, M. | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2021 ジャーナル: J Virol / 年: 2021タイトル: Adeno-associated Virus 9 Structural Rearrangements Induced by Endosomal Trafficking pH and Glycan Attachment. 著者: Judit J Penzes / Paul Chipman / Nilakshee Bhattacharya / Allison Zeher / Rick Huang / Robert McKenna / Mavis Agbandje-McKenna /  要旨: Adeno-associated viruses (AAVs) are small nonenveloped single-stranded DNA (ssDNA) viruses that are currently being developed as gene therapy biologics. After cell entry, AAVs traffic to the nucleus ...Adeno-associated viruses (AAVs) are small nonenveloped single-stranded DNA (ssDNA) viruses that are currently being developed as gene therapy biologics. After cell entry, AAVs traffic to the nucleus using the endo-lysosomal pathway. The subsequent decrease in pH triggers conformational changes to the capsid that enable the externalization of the capsid protein (VP) N termini, including the unique domain of the minor capsid protein VP1 (VP1u), which permits the phospholipase activity required for the capsid lysosomal egress. Here, we report the AAV9 capsid structure, determined at the endosomal pHs (7.4, 6.0, 5.5, and 4.0), and terminal galactose-bound AAV9 capsids at pHs 7.4 and 5.5 using cryo-electron microscopy and three-dimensional image reconstruction. Taken together, these studies provide insight into AAV9 capsid conformational changes at the 5-fold pore during endosomal trafficking, in both the presence and absence of its cellular glycan receptor. We visualized, for the first time, that acidification induces the externalization of the VP3 and possibly VP2 N termini, presumably in prelude to the externalization of VP1u at pH 4.0, which is essential for lysosomal membrane disruption. In addition, the structural study of AAV9-galactose interactions demonstrates that AAV9 remains attached to its glycan receptor at the late endosome pH 5.5. This interaction significantly alters the conformational stability of the variable region I of the VPs, as well as the dynamics associated with VP N terminus externalization. There are 13 distinct Adeno-associated virus (AAV) serotypes that are structurally homologous and whose capsid proteins (VP1 to -3) are similar in amino acid sequence. However, AAV9 is one of the most commonly studied and is used as a gene therapy vector. This is partly because AAV9 is capable of crossing the blood-brain barrier and readily transduces a wide array of tissues, including the central nervous system. In this study, we provide AAV9 capsid structural insight during intracellular trafficking. Although the AAV capsid has been shown to externalize the N termini of its VPs, to enzymatically disrupt the lysosome membrane at low pH, there was no structural evidence to confirm this. By utilizing AAV9 as our model, we provide the first structural evidence that the externalization process occurs at the protein interface at the icosahedral 5-fold symmetry axis and can be triggered by lowering the pH. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7mua.cif.gz 7mua.cif.gz | 5.6 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7mua.ent.gz pdb7mua.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  7mua.json.gz 7mua.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7mua_validation.pdf.gz 7mua_validation.pdf.gz | 2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7mua_full_validation.pdf.gz 7mua_full_validation.pdf.gz | 2 MB | 表示 | |
| XML形式データ |  7mua_validation.xml.gz 7mua_validation.xml.gz | 632.8 KB | 表示 | |
| CIF形式データ |  7mua_validation.cif.gz 7mua_validation.cif.gz | 1 MB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/mu/7mua https://data.pdbj.org/pub/pdb/validation_reports/mu/7mua ftp://data.pdbj.org/pub/pdb/validation_reports/mu/7mua ftp://data.pdbj.org/pub/pdb/validation_reports/mu/7mua | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
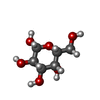
| #1: タンパク質 | 分子量: 58474.414 Da / 分子数: 60 / 断片: UNP residues 219-736 / 由来タイプ: 組換発現 由来: (組換発現)   Adeno-associated virus 9 (アデノ随伴ウイルス) Adeno-associated virus 9 (アデノ随伴ウイルス)遺伝子: cap / プラスミド: FastBac1-HM / 細胞株 (発現宿主): Sf9 発現宿主:  参照: UniProt: Q6JC40 #2: 糖 | ChemComp-GAL / 研究の焦点であるリガンドがあるか | Y | |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Adeno-associated virus 9 / タイプ: VIRUS / Entity ID: #1 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:   Adeno-associated virus 9 (アデノ随伴ウイルス) Adeno-associated virus 9 (アデノ随伴ウイルス) |
| 由来(組換発現) | 生物種:  細胞: Sf9 / プラスミド: FastBac1-HM |
| ウイルスについての詳細 | 中空か: YES / エンベロープを持つか: NO / 単離: SEROTYPE / タイプ: VIRUS-LIKE PARTICLE |
| 天然宿主 | 生物種: Homo sapiens |
| ウイルス殻 | 名称: AAV9 virus like particle / 直径: 240 nm / 三角数 (T数): 1 |
| 緩衝液 | pH: 5.5 詳細: 1x Universal buffer of 20 mM HEPES, 20 mM MES, 20 mM sodium acetate, 0.15 M NaCl, 3.7 mM CaCl2 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | グリッドの材料: COPPER / グリッドのタイプ: Quantifoil |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源: OTHER / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: OTHER / Cs: 2.7 mm |
| 撮影 | 電子線照射量: 63 e/Å2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 1445 |
| 画像スキャン | 動画フレーム数/画像: 50 |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.10-2155_2155: / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING ONLY | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 134697 | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: I (正20面体型対称) | ||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.68 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 110690 / アルゴリズム: FOURIER SPACE / クラス平均像の数: 15 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 3UX1 PDB chain-ID: A / Accession code: 3UX1 / Source name: PDB / タイプ: experimental model | ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera





























 PDBj
PDBj