+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7843 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of RAG in complex with 12-RSS and 23-RSS nicked DNA intermediates | |||||||||
 マップデータ マップデータ | RAG in complex with 12-RSS and 23-RSS nicked DNA intermediates, sharpened map with B factor -91 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | V(D)J recombination / synaptic RAG complex / nicked RSS intermediates / paired complex / RECOMBINATION-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報somatic diversification of immune receptors via germline recombination within a single locus / hematopoietic or lymphoid organ development / protein-DNA complex assembly / DNA recombinase complex / endodeoxyribonuclease complex / immunoglobulin V(D)J recombination / lymphocyte differentiation / V(D)J recombination / phosphatidylinositol-3,4-bisphosphate binding / phosphatidylinositol-3,5-bisphosphate binding ...somatic diversification of immune receptors via germline recombination within a single locus / hematopoietic or lymphoid organ development / protein-DNA complex assembly / DNA recombinase complex / endodeoxyribonuclease complex / immunoglobulin V(D)J recombination / lymphocyte differentiation / V(D)J recombination / phosphatidylinositol-3,4-bisphosphate binding / phosphatidylinositol-3,5-bisphosphate binding / detection of maltose stimulus / maltose transport complex / maltose binding / carbohydrate transport / phosphatidylinositol-3,4,5-trisphosphate binding / maltose transport / maltodextrin transmembrane transport / T cell differentiation / carbohydrate transmembrane transporter activity / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / phosphatidylinositol-4,5-bisphosphate binding / methylated histone binding / B cell differentiation / ATP-binding cassette (ABC) transporter complex / phosphatidylinositol binding / cell chemotaxis / thymus development / RING-type E3 ubiquitin transferase / ubiquitin-protein transferase activity / ubiquitin protein ligase activity / chromatin organization / outer membrane-bounded periplasmic space / histone binding / T cell differentiation in thymus / endonuclease activity / DNA recombination / sequence-specific DNA binding / adaptive immune response / 加水分解酵素; エステル加水分解酵素 / periplasmic space / DNA damage response / chromatin binding / magnesium ion binding / protein homodimerization activity / DNA binding / zinc ion binding / membrane / nucleus / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Wu H / Liao M | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2018 ジャーナル: Nat Struct Mol Biol / 年: 2018タイトル: DNA melting initiates the RAG catalytic pathway. 著者: Heng Ru / Wei Mi / Pengfei Zhang / Frederick W Alt / David G Schatz / Maofu Liao / Hao Wu /  要旨: The mechanism for initiating DNA cleavage by DDE-family enzymes, including the RAG endonuclease, which initiates V(D)J recombination, is not well understood. Here we report six cryo-EM structures of ...The mechanism for initiating DNA cleavage by DDE-family enzymes, including the RAG endonuclease, which initiates V(D)J recombination, is not well understood. Here we report six cryo-EM structures of zebrafish RAG in complex with one or two intact recombination signal sequences (RSSs), at up to 3.9-Å resolution. Unexpectedly, these structures reveal DNA melting at the heptamer of the RSSs, thus resulting in a corkscrew-like rotation of coding-flank DNA and the positioning of the scissile phosphate in the active site. Substrate binding is associated with dimer opening and a piston-like movement in RAG1, first outward to accommodate unmelted DNA and then inward to wedge melted DNA. These precleavage complexes show limited base-specific contacts of RAG at the conserved terminal CAC/GTG sequence of the heptamer, thus suggesting conservation based on a propensity to unwind. CA and TG overwhelmingly dominate terminal sequences in transposons and retrotransposons, thereby implicating a universal mechanism for DNA melting during the initiation of retroviral integration and DNA transposition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7843.map.gz emd_7843.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7843-v30.xml emd-7843-v30.xml emd-7843.xml emd-7843.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7843.png emd_7843.png | 68.5 KB | ||
| Filedesc metadata |  emd-7843.cif.gz emd-7843.cif.gz | 7.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7843 http://ftp.pdbj.org/pub/emdb/structures/EMD-7843 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7843 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7843 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7843_validation.pdf.gz emd_7843_validation.pdf.gz | 589.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7843_full_validation.pdf.gz emd_7843_full_validation.pdf.gz | 589 KB | 表示 | |
| XML形式データ |  emd_7843_validation.xml.gz emd_7843_validation.xml.gz | 6.1 KB | 表示 | |
| CIF形式データ |  emd_7843_validation.cif.gz emd_7843_validation.cif.gz | 6.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7843 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7843 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7843 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7843 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6dbiMC  7844C  7845C  7846C  7847C  7848C  7849C  7850C  7851C  7852C  7853C  6dbjC  6dblC  6dboC  6dbqC  6dbrC  6dbtC  6dbuC  6dbvC  6dbwC  6dbxC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7843.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7843.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RAG in complex with 12-RSS and 23-RSS nicked DNA intermediates, sharpened map with B factor -91 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
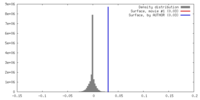
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : RAG in complex with 12-RSS and 23-RSS nicked DNA intermediates
+超分子 #1: RAG in complex with 12-RSS and 23-RSS nicked DNA intermediates
+分子 #1: Recombination activating gene 1 - MBP chimera
+分子 #2: Recombination activating gene 2
+分子 #3: Forward strand of 12-RSS signal end
+分子 #4: Reverse strand of 12-RSS
+分子 #5: Reverse strand of 23-RSS
+分子 #6: Forward strand of 23-RSS signal end
+分子 #7: Forward strand of coding flank
+分子 #8: ZINC ION
+分子 #9: CALCIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.38 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Solutions were made fresh from concentrated to avoid microbial contamination. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE | |||||||||||||||
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 53109 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)