+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24625 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Archaeal DNA ligase and heterotrimeric PCNA in complex with end-joined DNA | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA ligase / PCNA / cryo-EM / LIGASE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA ligase (ATP) / DNA ligase (ATP) activity / DNA ligation / lagging strand elongation / DNA biosynthetic process / leading strand elongation / DNA polymerase processivity factor activity / regulation of DNA replication / DNA recombination / cell division ...DNA ligase (ATP) / DNA ligase (ATP) activity / DNA ligation / lagging strand elongation / DNA biosynthetic process / leading strand elongation / DNA polymerase processivity factor activity / regulation of DNA replication / DNA recombination / cell division / DNA repair / DNA binding / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharolobus solfataricus (古細菌) / Saccharolobus solfataricus (古細菌) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Sverzhinsky A / Pascal JM | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Cryo-EM structures and biochemical insights into heterotrimeric PCNA regulation of DNA ligase. 著者: Aleksandr Sverzhinsky / Alan E Tomkinson / John M Pascal /   要旨: DNA ligases act in the final step of many DNA repair pathways and are commonly regulated by the DNA sliding clamp proliferating cell nuclear antigen (PCNA), but there are limited insights into the ...DNA ligases act in the final step of many DNA repair pathways and are commonly regulated by the DNA sliding clamp proliferating cell nuclear antigen (PCNA), but there are limited insights into the physical basis for this regulation. Here, we use single-particle cryoelectron microscopy (cryo-EM) to analyze an archaeal DNA ligase and heterotrimeric PCNA in complex with a single-strand DNA break. The cryo-EM structures highlight a continuous DNA-binding surface formed between DNA ligase and PCNA that supports the distorted conformation of the DNA break undergoing repair and contributes to PCNA stimulation of DNA ligation. DNA ligase is conformationally flexible within the complex, with its domains fully ordered only when encircling the repaired DNA to form a stacked ring structure with PCNA. The structures highlight DNA ligase structural transitions while docked on PCNA, changes in DNA conformation during ligation, and the potential for DNA ligase domains to regulate PCNA accessibility to other repair factors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24625.map.gz emd_24625.map.gz | 22 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24625-v30.xml emd-24625-v30.xml emd-24625.xml emd-24625.xml | 22.6 KB 22.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24625.png emd_24625.png | 135 KB | ||
| Filedesc metadata |  emd-24625.cif.gz emd-24625.cif.gz | 7.1 KB | ||
| その他 |  emd_24625_additional_1.map.gz emd_24625_additional_1.map.gz | 22 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24625 http://ftp.pdbj.org/pub/emdb/structures/EMD-24625 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24625 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24625 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24625_validation.pdf.gz emd_24625_validation.pdf.gz | 465.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24625_full_validation.pdf.gz emd_24625_full_validation.pdf.gz | 465.4 KB | 表示 | |
| XML形式データ |  emd_24625_validation.xml.gz emd_24625_validation.xml.gz | 4.9 KB | 表示 | |
| CIF形式データ |  emd_24625_validation.cif.gz emd_24625_validation.cif.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24625 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24625 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24625 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24625 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24625.map.gz / 形式: CCP4 / 大きさ: 24.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24625.map.gz / 形式: CCP4 / 大きさ: 24.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
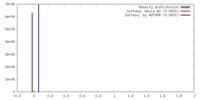
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_24625_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
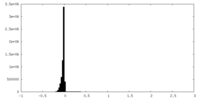
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
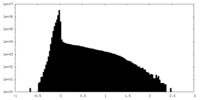
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of DNA Ligase with PCNA1-2-3 and end-joined DNA
| 全体 | 名称: Ternary complex of DNA Ligase with PCNA1-2-3 and end-joined DNA |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of DNA Ligase with PCNA1-2-3 and end-joined DNA
| 超分子 | 名称: Ternary complex of DNA Ligase with PCNA1-2-3 and end-joined DNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:   Saccharolobus solfataricus (古細菌) Saccharolobus solfataricus (古細菌) |
| 分子量 | 理論値: 175 KDa |
-分子 #1: DNA polymerase sliding clamp 1
| 分子 | 名称: DNA polymerase sliding clamp 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharolobus solfataricus (古細菌) / 株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 Saccharolobus solfataricus (古細菌) / 株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 |
| 分子量 | 理論値: 28.711961 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAFKIVYPNA KDFFSFINSI TNVTDSIILN FTEDGIFSRH LTEDKVLMAI MRIPKDVLSE YSIDSPTSVK LDVSSVKKIL SKASSKKAT IELTETDSGL KIIIRDEKSG AKSTIYIKAE KGQVEQLTEP KVNLAVNFTT DESVLNVIAA DVTLVGEEMR I STEEDKIK ...文字列: MAFKIVYPNA KDFFSFINSI TNVTDSIILN FTEDGIFSRH LTEDKVLMAI MRIPKDVLSE YSIDSPTSVK LDVSSVKKIL SKASSKKAT IELTETDSGL KIIIRDEKSG AKSTIYIKAE KGQVEQLTEP KVNLAVNFTT DESVLNVIAA DVTLVGEEMR I STEEDKIK IEAGEEGKRY VAFLMKDKPL KELSIDTSAS SSYSAEMFKD AVKGLRGFSA PTMVSFGENL PMKIDVEAVS GG HMIFWIA PRLLEHHHHH H UniProtKB: DNA polymerase sliding clamp 1 |
-分子 #2: DNA polymerase sliding clamp 2
| 分子 | 名称: DNA polymerase sliding clamp 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharolobus solfataricus (古細菌) / 株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 Saccharolobus solfataricus (古細菌) / 株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 |
| 分子量 | 理論値: 27.461084 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKAKVIDAVS FSYILRTVGD FLSEANFIVT KEGIRVSGID PSRVVFLDIF LPSSYFEGFE VSQEKEIIGF KLEDVNDILK RVLKDDTLI LSSNESKLTL TFDGEFTRSF ELPLIQVEST QPPSVNLEFP FKAQLLTITF ADIIDELSDL GEVLNIHSKE N KLYFEVIG ...文字列: MKAKVIDAVS FSYILRTVGD FLSEANFIVT KEGIRVSGID PSRVVFLDIF LPSSYFEGFE VSQEKEIIGF KLEDVNDILK RVLKDDTLI LSSNESKLTL TFDGEFTRSF ELPLIQVEST QPPSVNLEFP FKAQLLTITF ADIIDELSDL GEVLNIHSKE N KLYFEVIG DLSTAKVELS TDNGTLLEAS GADVSSSYGM EYVANTTKMR RASDSMELYF GSQIPLKLRF KLPQEGYGDF YI APRAD UniProtKB: DNA polymerase sliding clamp 2 |
-分子 #3: DNA polymerase sliding clamp 3
| 分子 | 名称: DNA polymerase sliding clamp 3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharolobus solfataricus (古細菌) / 株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 Saccharolobus solfataricus (古細菌) / 株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 |
| 分子量 | 理論値: 28.560268 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKVVYDDVRV LKDIIQALAR LVDEAVLKFK QDSVELVALD RAHISLISVN LPREMFKEYD VNDEFKFGFN TQYLMKILKV AKRKEAIEI ASESPDSVII NIIGSTNREF NVRNLEVSEQ EIPEINLQFD ISATISSDGF KSAISEVSTV TDNVVVEGHE D RILIKAEG ...文字列: MKVVYDDVRV LKDIIQALAR LVDEAVLKFK QDSVELVALD RAHISLISVN LPREMFKEYD VNDEFKFGFN TQYLMKILKV AKRKEAIEI ASESPDSVII NIIGSTNREF NVRNLEVSEQ EIPEINLQFD ISATISSDGF KSAISEVSTV TDNVVVEGHE D RILIKAEG ESEVEVEFSK DTGGLQDLEF SKESKNSYSA EYLDDVLSLT KLSDYVKISF GNQKPLQLFF NMEGGGKVTY LL APKVLEH HHHHH UniProtKB: DNA polymerase sliding clamp 3 |
-分子 #6: DNA ligase
| 分子 | 名称: DNA ligase / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA ligase (ATP) |
|---|---|
| 由来(天然) | 生物種:   Saccharolobus solfataricus (古細菌) / 株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 Saccharolobus solfataricus (古細菌) / 株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 |
| 分子量 | 理論値: 69.998375 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MEFKVIAEYF DKLEKISSRL QLTALLADLL SKSDKTIIDK VVYIIQGKLW PDFLGYPELG IGEKFLIKA ISIATNTDEN SVENLYKTIG DLGEVARRLK SKQQSTGILG FLGTTSKESL TVDEVYSTLS KVALTTGEGS R DLKIRLLA ...文字列: MGSSHHHHHH SSGLVPRGSH MEFKVIAEYF DKLEKISSRL QLTALLADLL SKSDKTIIDK VVYIIQGKLW PDFLGYPELG IGEKFLIKA ISIATNTDEN SVENLYKTIG DLGEVARRLK SKQQSTGILG FLGTTSKESL TVDEVYSTLS KVALTTGEGS R DLKIRLLA GLLKKADPLE AKFLVRFVEG RLRVGIGDAT VLDAMAIAFG GGQSASEIIE RAYNLRADLG NIAKIIVEKG IE ALKTLKP QVGIPIRPML AERLSNPEEI LKKMGGNAIV DYKYDGERAQ IHKKEDKIFI FSRRLENITS QYPDVVDYVS KYI EGKEFI IEGEIVAIDP ESGEMRPFQE LMHRKRKSDI YEAIKEYPVN VFLFDLMYYE DVDYTTKPLE ARRKLLESIV KPND YVKIA HHIQANNVED LKSFFYRAIS EGGEGVMVKA IGKDAIYQAG ARGWLWIKLK RDYQSEMADT VDLVVVGGFY GKGKR GGKI SSLLMAAYNP KTDSFESVCK VASGFSDEQL DELQKKLMEI KRDVKHPRVN SKMEPDIWVE PVYVAEIIGS EITISP LHT CCQDVVEKDA GLSIRFPRFI RWRDDKSPED ATTTDEILEM YNKQPKKKIE SPAVDESV UniProtKB: DNA ligase |
-分子 #4: End-joined DNA
| 分子 | 名称: End-joined DNA / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 14.549299 KDa |
| 配列 | 文字列: (DG)(DT)(DA)(DT)(DC)(DC)(DT)(DC)(DG)(DT) (DA)(DG)(DT)(DG)(DC)(DA)(DG)(DA)(DT)(DG) (DC)(DG)(DT)(DC)(DG)(DT)(DC)(DG)(DG) (DA)(DC)(DT)(DG)(DA)(DT)(DT)(DC)(DG)(DG) (DT) (DA)(DG)(DA)(DT)(DC)(DT)(DG) |
-分子 #5: Template strand DNA
| 分子 | 名称: Template strand DNA / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 14.403264 KDa |
| 配列 | 文字列: (DC)(DA)(DG)(DA)(DT)(DC)(DT)(DA)(DC)(DC) (DG)(DA)(DA)(DT)(DC)(DA)(DG)(DT)(DC)(DC) (DG)(DA)(DC)(DG)(DA)(DC)(DG)(DC)(DA) (DT)(DC)(DT)(DG)(DC)(DA)(DC)(DT)(DA)(DC) (DG) (DA)(DG)(DG)(DA)(DT)(DA)(DC) |
-分子 #7: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 7 / コピー数: 3 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.35 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: wait time 0, blot force 1, blot time 1, drain time 0. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 8060 / 平均電子線量: 100.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 4.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.1) / 使用した粒子像数: 192490 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.1) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.1) |
-原子モデル構築 1
| 初期モデル |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | プロトコル: RIGID BODY FIT | ||||||||||||
| 得られたモデル |  PDB-7rpx: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)