+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23036 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Yeast TFIIK (Kin28/Ccl1/Tfb3) Complex | |||||||||
 マップデータ マップデータ | CryoEM structure of Yeast TFIIK (Kin28/Ccl1/Tfb3) Complex | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / : / : / positive regulation of Atg1/ULK1 kinase complex assembly / : / : / transcription factor TFIIK complex / cellular response to nitrogen starvation / cyclin-dependent protein serine/threonine kinase activator activity / transcription factor TFIIH holo complex ...: / : / : / positive regulation of Atg1/ULK1 kinase complex assembly / : / : / transcription factor TFIIK complex / cellular response to nitrogen starvation / cyclin-dependent protein serine/threonine kinase activator activity / transcription factor TFIIH holo complex / [RNA-polymerase]-subunit kinase / cyclin-dependent protein serine/threonine kinase regulator activity / RNA Pol II CTD phosphorylation and interaction with CE / Formation of the Early Elongation Complex / mRNA Capping / RNA polymerase II transcribes snRNA genes / TP53 Regulates Transcription of DNA Repair Genes / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / RNA Polymerase II Pre-transcription Events / Formation of TC-NER Pre-Incision Complex / RNA Polymerase I Promoter Escape / regulation of cyclin-dependent protein serine/threonine kinase activity / Gap-filling DNA repair synthesis and ligation in TC-NER / Dual incision in TC-NER / 7-methylguanosine mRNA capping / transcription by RNA polymerase I / cyclin-dependent protein serine/threonine kinase activity / positive regulation of autophagy / RNA polymerase II CTD heptapeptide repeat kinase activity /  transcription initiation at RNA polymerase II promoter / nucleotide-excision repair / transcription by RNA polymerase II / transcription initiation at RNA polymerase II promoter / nucleotide-excision repair / transcription by RNA polymerase II /  protein kinase activity / protein kinase activity /  細胞周期 / 細胞周期 /  細胞分裂 / 細胞分裂 /  protein phosphorylation / protein phosphorylation /  DNA修復 / regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / DNA修復 / regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /  ミトコンドリア / ミトコンドリア /  ATP binding / ATP binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) / Saccharomyces cerevisiae (パン酵母) /   Baker's yeast (パン酵母) Baker's yeast (パン酵母) | |||||||||
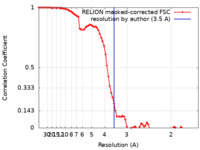
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | van Eeuwen T / Murakami K / Li T / Tsai KL | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Structure of TFIIK for phosphorylation of CTD of RNA polymerase II. 著者: Trevor van Eeuwen / Tao Li / Hee Jong Kim / Jose J Gorbea Colón / Mitchell I Parker / Roland L Dunbrack / Benjamin A Garcia / Kuang-Lei Tsai / Kenji Murakami /  要旨: During transcription initiation, the general transcription factor TFIIH marks RNA polymerase II by phosphorylating Ser5 of the carboxyl-terminal domain (CTD) of Rpb1, which is followed by extensive ...During transcription initiation, the general transcription factor TFIIH marks RNA polymerase II by phosphorylating Ser5 of the carboxyl-terminal domain (CTD) of Rpb1, which is followed by extensive modifications coupled to transcription elongation, mRNA processing, and histone dynamics. We have determined a 3.5-Å resolution cryo-electron microscopy (cryo-EM) structure of the TFIIH kinase module (TFIIK in yeast), which is composed of Kin28, Ccl1, and Tfb3, yeast homologs of CDK7, cyclin H, and MAT1, respectively. The carboxyl-terminal region of Tfb3 was lying at the edge of catalytic cleft of Kin28, where a conserved Tfb3 helix served to stabilize the activation loop in its active conformation. By combining the structure of TFIIK with the previous cryo-EM structure of the preinitiation complex, we extend the previously proposed model of the CTD path to the active site of TFIIK. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23036.map.gz emd_23036.map.gz | 38.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23036-v30.xml emd-23036-v30.xml emd-23036.xml emd-23036.xml | 24 KB 24 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23036_fsc.xml emd_23036_fsc.xml | 8.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23036.png emd_23036.png | 144.7 KB | ||
| その他 |  emd_23036_additional_1.map.gz emd_23036_additional_1.map.gz emd_23036_half_map_1.map.gz emd_23036_half_map_1.map.gz emd_23036_half_map_2.map.gz emd_23036_half_map_2.map.gz | 3.2 MB 33 MB 33 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23036 http://ftp.pdbj.org/pub/emdb/structures/EMD-23036 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23036 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23036 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23036.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23036.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of Yeast TFIIK (Kin28/Ccl1/Tfb3) Complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Additional map
| ファイル | emd_23036_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Additional map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 1
| ファイル | emd_23036_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 2
| ファイル | emd_23036_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of Kin28-Ccl1-Tfb3 from Saccharomyces cerevisiae.
| 全体 | 名称: Ternary complex of Kin28-Ccl1-Tfb3 from Saccharomyces cerevisiae. |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of Kin28-Ccl1-Tfb3 from Saccharomyces cerevisiae.
| 超分子 | 名称: Ternary complex of Kin28-Ccl1-Tfb3 from Saccharomyces cerevisiae. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: the yeast Cdk7 complex, that phosphorylates the RNA pol II C-terminal domain (CTD) in transcription initiation. |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 株: CB010 Tfb3-TAP Saccharomyces cerevisiae (パン酵母) / 株: CB010 Tfb3-TAP |
| 分子量 | 実験値: 73 KDa |
-分子 #1: RNA polymerase II transcription factor B subunit 3
| 分子 | 名称: RNA polymerase II transcription factor B subunit 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 38.128355 KDa |
| 配列 | 文字列: MLMDEYEENK DMCPICKTDR YLSPDVKFLV NPECYHRICE SCVDRIFSLG PAQCPYKGCD KILRKNKFKT QIFDDVEVEK EVDIRKRVF NVFNKTIDDF NGDLVEYNKY LEEVEDIIYK LDHGIDVAKT EEKLRTYEEL NKQLIMNNLE RSRTEIESFE Q RQKFEKEM ...文字列: MLMDEYEENK DMCPICKTDR YLSPDVKFLV NPECYHRICE SCVDRIFSLG PAQCPYKGCD KILRKNKFKT QIFDDVEVEK EVDIRKRVF NVFNKTIDDF NGDLVEYNKY LEEVEDIIYK LDHGIDVAKT EEKLRTYEEL NKQLIMNNLE RSRTEIESFE Q RQKFEKEM KLKKRLLERQ IEEEERMNKE WTKKEIVNRL STTTQDINET IEGVKNTVKL KKSSARRKLE ELNRVLKNNP YF NSNVNVQ NSRLKDAVPF TPFNGDREAH PPFTLKGSVY NDPFIKDLEH RKEFIASGFN TNYAYERVLT EAFMGLGCVI SEE L |
-分子 #2: Serine/threonine-protein kinase KIN28
| 分子 | 名称: Serine/threonine-protein kinase KIN28 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: [RNA-polymerase]-subunit kinase |
|---|---|
| 由来(天然) | 生物種:   Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 35.369867 KDa |
| 配列 | 文字列: MKVNMEYTKE KKVGEGTYAV VYLGCQHSTG RKIAIKEIKT SEFKDGLDMS AIREVKYLQE MQHPNVIELI DIFMAYDNLN LVLEFLPTD LEVVIKDKSI LFTPADIKAW MLMTLRGVYH CHRNFILHRD LKPNNLLFSP DGQIKVADFG LARAIPAPHE I L(TPO) ...文字列: MKVNMEYTKE KKVGEGTYAV VYLGCQHSTG RKIAIKEIKT SEFKDGLDMS AIREVKYLQE MQHPNVIELI DIFMAYDNLN LVLEFLPTD LEVVIKDKSI LFTPADIKAW MLMTLRGVYH CHRNFILHRD LKPNNLLFSP DGQIKVADFG LARAIPAPHE I L(TPO)SNVVTR WYRAPELLFG AKHYTSAIDI WSVGVIFAEL MLRIPYLPGQ NDVDQMEVTF RALGTPTDRD WPEVSSFM T YNKLQIYPPP SRDELRKRFI AASEYALDFM CGMLTMNPQK RWTAVQCLES DYFKELPPPS DPSSIKIRN |
-分子 #3: Cyclin CCL1
| 分子 | 名称: Cyclin CCL1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c Baker's yeast (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 45.259414 KDa |
| 配列 | 文字列: MTDIQLNGKS TLDTPSATMS AKEKEAKLKS ADENNKPPNY KRISDSQLYR HSSQYRMWSY TKDQLQEKRV DTNARAIAYI EENLLKFRE AHNLTEEEIK VLEAKAIPLT MEEELDLVNF YAKKVQVIAQ HLNLPTEVVA TAISFFRRFF LENSVMQIDP K SIVHTTIF ...文字列: MTDIQLNGKS TLDTPSATMS AKEKEAKLKS ADENNKPPNY KRISDSQLYR HSSQYRMWSY TKDQLQEKRV DTNARAIAYI EENLLKFRE AHNLTEEEIK VLEAKAIPLT MEEELDLVNF YAKKVQVIAQ HLNLPTEVVA TAISFFRRFF LENSVMQIDP K SIVHTTIF LACKSENYFI SVDSFAQKAK STRDSVLKFE FKLLESLKFS LLNHHPYKPL HGFFLDIQNV LYGKVDLNYM GQ IYDRCKK RITAALLTDV VYFYTPPQIT LATLLIEDEA LVTRYLETKF PSREGSQESV PGNEKEEPQN DASTTEKNKE KST ESEEYS IDSAKLLTII RECKSIIEDC KPPSTEEAKK IAAKNYYCQN PSTLIQKLKR KLNGEDTSST VEKKQKT |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #5: ALUMINUM FLUORIDE
| 分子 | 名称: ALUMINUM FLUORIDE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: AF3 |
|---|---|
| 分子量 | 理論値: 83.977 Da |
| Chemical component information |  ChemComp-AF3: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.08 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 0 % / チャンバー内温度: 293 K / 装置: LEICA EM CPC 詳細: blotted for 2 seconds with Whatman 41 ashless filter paper. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4620 / 平均露光時間: 2.24 sec. / 平均電子線量: 45.0 e/Å2 詳細: Images collected in super-resolution mode. Movies were 35 frames. Imaging 1 image/hole, image shift between 4 holes. Focus once per image shift |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


























 Z
Z Y
Y X
X