+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21600 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single-Particle Cryo-EM Structure of Arabinofuranosyltransferase AftD from Mycobacteria, Mutant R1389S Class 1 | |||||||||||||||||||||
 マップデータ マップデータ | Sharpened map | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
| 機能・相同性 | Alpha-(1->3)-arabinofuranosyltransferase / Alpha-(1->3)-arabinofuranosyltransferase / Coagulation factors 5/8 type C domain (FA58C) profile. / Coagulation factor 5/8 C-terminal domain / Galactose-binding-like domain superfamily / transferase activity / membrane / DUF3367 domain-containing protein 機能・相同性情報 機能・相同性情報 | |||||||||||||||||||||
| 生物種 |  Mycobacteroides abscessus (バクテリア) Mycobacteroides abscessus (バクテリア) | |||||||||||||||||||||
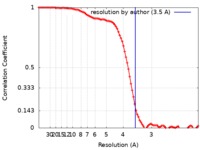
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Tan YZ / Zhang L / Rodrigues J / Zheng RB / Giacometti SI / Rosario AL / Kloss B / Dandey VP / Wei H / Brunton R ...Tan YZ / Zhang L / Rodrigues J / Zheng RB / Giacometti SI / Rosario AL / Kloss B / Dandey VP / Wei H / Brunton R / Raczkowski AM / Athayde D / Catalao MJ / Pimentel M / Clarke OB / Lowary TL / Archer M / Niederweis M / Potter CS / Carragher B / Mancia F | |||||||||||||||||||||
| 資金援助 |  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2020 ジャーナル: Mol Cell / 年: 2020タイトル: Cryo-EM Structures and Regulation of Arabinofuranosyltransferase AftD from Mycobacteria. 著者: Yong Zi Tan / Lei Zhang / José Rodrigues / Ruixiang Blake Zheng / Sabrina I Giacometti / Ana L Rosário / Brian Kloss / Venkata P Dandey / Hui Wei / Richard Brunton / Ashleigh M Raczkowski / ...著者: Yong Zi Tan / Lei Zhang / José Rodrigues / Ruixiang Blake Zheng / Sabrina I Giacometti / Ana L Rosário / Brian Kloss / Venkata P Dandey / Hui Wei / Richard Brunton / Ashleigh M Raczkowski / Diogo Athayde / Maria João Catalão / Madalena Pimentel / Oliver B Clarke / Todd L Lowary / Margarida Archer / Michael Niederweis / Clinton S Potter / Bridget Carragher / Filippo Mancia /     要旨: Mycobacterium tuberculosis causes tuberculosis, a disease that kills over 1 million people each year. Its cell envelope is a common antibiotic target and has a unique structure due, in part, to two ...Mycobacterium tuberculosis causes tuberculosis, a disease that kills over 1 million people each year. Its cell envelope is a common antibiotic target and has a unique structure due, in part, to two lipidated polysaccharides-arabinogalactan and lipoarabinomannan. Arabinofuranosyltransferase D (AftD) is an essential enzyme involved in assembling these glycolipids. We present the 2.9-Å resolution structure of M. abscessus AftD, determined by single-particle cryo-electron microscopy. AftD has a conserved GT-C glycosyltransferase fold and three carbohydrate-binding modules. Glycan array analysis shows that AftD binds complex arabinose glycans. Additionally, AftD is non-covalently complexed with an acyl carrier protein (ACP). 3.4- and 3.5-Å structures of a mutant with impaired ACP binding reveal a conformational change, suggesting that ACP may regulate AftD function. Mutagenesis experiments using a conditional knockout constructed in M. smegmatis confirm the essentiality of the putative active site and the ACP binding for AftD function. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21600.map.gz emd_21600.map.gz | 56.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21600-v30.xml emd-21600-v30.xml emd-21600.xml emd-21600.xml | 33.1 KB 33.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21600_fsc.xml emd_21600_fsc.xml | 9.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21600.png emd_21600.png | 495.9 KB | ||
| マスクデータ |  emd_21600_msk_1.map emd_21600_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_21600_additional_1.map.gz emd_21600_additional_1.map.gz emd_21600_additional_2.map.gz emd_21600_additional_2.map.gz emd_21600_additional_3.map.gz emd_21600_additional_3.map.gz emd_21600_additional_4.map.gz emd_21600_additional_4.map.gz emd_21600_half_map_1.map.gz emd_21600_half_map_1.map.gz emd_21600_half_map_2.map.gz emd_21600_half_map_2.map.gz | 16.8 MB 8 MB 6.8 MB 105.9 KB 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21600 http://ftp.pdbj.org/pub/emdb/structures/EMD-21600 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21600 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21600 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21600_validation.pdf.gz emd_21600_validation.pdf.gz | 842 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21600_full_validation.pdf.gz emd_21600_full_validation.pdf.gz | 841.6 KB | 表示 | |
| XML形式データ |  emd_21600_validation.xml.gz emd_21600_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_21600_validation.cif.gz emd_21600_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21600 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21600 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21600 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21600 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6wbxMC  6w98C  6wbyC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10391 (タイトル: Single-Particle Cryo-EM of Arabinofuranosyltransferase AftD from Mycobacteria, Mutant R1389S EMPIAR-10391 (タイトル: Single-Particle Cryo-EM of Arabinofuranosyltransferase AftD from Mycobacteria, Mutant R1389SData size: 2.3 TB Data #1: Unaligned and compressed multi-frame movies [micrographs - multiframe] Data #2: Aligned and dose-weighted micrographs [micrographs - single frame] Data #3: Final Particle Stacks with Refined Euler Angles and Shifts (Overall and Focused Refined on CBM3) [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21600.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21600.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0605 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21600_msk_1.map emd_21600_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Raw map
| ファイル | emd_21600_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: 3DFSC
| ファイル | emd_21600_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3DFSC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: 3DFSC - Thresholded
| ファイル | emd_21600_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3DFSC - Thresholded | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: 3DFSC - Thresholded, Binarized
| ファイル | emd_21600_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3DFSC - Thresholded, Binarized | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_21600_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_21600_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mutant R1389S Class 1 Mycobacterial Arabinofuranosyltransferase A...
| 全体 | 名称: Mutant R1389S Class 1 Mycobacterial Arabinofuranosyltransferase AftD Complexed with Acyl Carrier Protein |
|---|---|
| 要素 |
|
-超分子 #1: Mutant R1389S Class 1 Mycobacterial Arabinofuranosyltransferase A...
| 超分子 | 名称: Mutant R1389S Class 1 Mycobacterial Arabinofuranosyltransferase AftD Complexed with Acyl Carrier Protein タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Mycobacteroides abscessus (バクテリア) Mycobacteroides abscessus (バクテリア) |
| 分子量 | 理論値: 5 MDa |
| 組換発現 | 生物種:  |
-分子 #1: DUF3367 domain-containing protein
| 分子 | 名称: DUF3367 domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacteroides abscessus (バクテリア) Mycobacteroides abscessus (バクテリア) |
| 分子量 | 理論値: 152.818641 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDYKDDDDKH HHHHHHHHHE NLYFQSYVMT YRLDSSALSR RWLAVAAAVS LLLTFSQSPG QISPDTKLDL AINPLRFAAR ALNLWSSDL PFGQAQNQAY GYLFPHGAFF SLGHLLGVPA WVTQRLWWAL LIVAGFWGLI RVAEALGIGT RGSRIIAAVA F ALSPRVLT ...文字列: MDYKDDDDKH HHHHHHHHHE NLYFQSYVMT YRLDSSALSR RWLAVAAAVS LLLTFSQSPG QISPDTKLDL AINPLRFAAR ALNLWSSDL PFGQAQNQAY GYLFPHGAFF SLGHLLGVPA WVTQRLWWAL LIVAGFWGLI RVAEALGIGT RGSRIIAAVA F ALSPRVLT TLGAISSETL PMMLAPWVLL PLILTFQGRM SPRRAAALSA VAVALMGAVN AVATALACGV AVIWWLAHRP NR TWWRFTA WWIPCLALAS TWWIVALLIF GKISPKFLDF IESSGVTTQW TSLTEVLRGT DSWTPFVAPT ATAGSSLVTQ SAM VIATTM LAAAGMAGLA MRGMPARGRL VAVLLIGLVL LTAGYTGALG SPIAQQIQFF LDDGGTPLRN VHKLEPLIRL PLIL GLAHA LSRIPLPASV PVRQWLSALA RPERNRAVAF AIVLLVALAA STSLAWTGRL VPRGGFDAIP GYWNDTAHWL ADHDT GGRA LVVPGAPFAI QTWGLTRDEP LQALGQTPWG VRDSIPLTPP ETIRAIDSVQ QLFAAGRPSD GLADTLREQG ISYLVV RND LDPDTSRSAR PILVHHTIEG SPGLTKVAQF GDPVGAGAVE GFVADSDLRP QYPAVEIYAV GANDHDGEPY FTDIDTM PR VAGGPEALLR LNERRRQLNE PPLGPSLLAT DAAQAGLRPG PAVVTDTPLA RETDYGRVDD HSSAIRAPGD KRRTFNRV P DYPATGVPLV NGSWTGGTIT ASSSASDSTA LPNVAPGTST AAAIDRDNAT SWVSSSLEAA LGQWIRIDLD RPITNAILT VTPSATALGA QVRRLEVETD NGTTSVRFDE PGQPLNIALR PGETTWVKVT ATGTDDGTSG VQFGVTELSL TQYDAAGFAH TVDLRHSAT VPPPPAGDNP LGWDLGSPLQ GRSGCAPSPQ RLRCAATLSL APEEPGTFIR TLTVPQPVSL TPRLWVRARP G PQLRDLIQ QPGTTVATGD SDVIDPQGSS YAATDGDPGT VWTAPQDSVQ RLHLPSLVIK LPKPTAIGAI RLRPSRTEVP AH PKQVAIN LGDGPQLRSI DPKADVTELA LHPSITDTIT VTVTDWTDII DRTALGFDQL KPPGIAEVIA LDADHRPIAP ADN AANSKR KITIGCNRGP ILALAGRFVP MSITATVREL LDGTVIQATP CDTSPIATGA GIQDVTVNPS QQFIVDGVQL TAAA TEPAS ATMTVAPKGA WGPDRREVTA EPSAHERVLA VPESINPGWA ARDAQGHLLT PVRVNGWQQG WVLPAGDGGK ITLTF GLNT WYRAGLFGGL ALLPILACLA LLPARGRTTL PPVAPWCAGP AAGVAVLAAL TAISGISGMA VGLAALAFKV WTRWPL RAV TAAGVYLAGG SLLLAGAALS RHPWRSVGGY TGHSWWIQLL ALISVASVAL AAVSLPSRRC WKRRSASREG DSTSA |
-分子 #2: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Solution was filtered and degassed. | |||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 298 K / 装置: LEICA EM GP | |||||||||
| 詳細 | Protein was incorporated into lipid nanodiscs. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 15 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-90 / 撮影したグリッド数: 1 / 実像数: 4886 / 平均露光時間: 13.05 sec. / 平均電子線量: 96.78 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)