+Search query
-Structure paper
| Title | Structure-derived insights from blood factors binding to the surfaces of different adenoviruses. |
|---|---|
| Journal, issue, pages | Nat Commun, Vol. 15, Issue 1, Page 9768, Year 2024 |
| Publish date | Nov 11, 2024 |
 Authors Authors | Haley E Mudrick / Shao-Chia Lu / Janarjan Bhandari / Mary E Barry / Jack R Hemsath / Felix G M Andres / Olivia X Ma / Michael A Barry / Vijay S Reddy /  |
| PubMed Abstract | The tropism of adenoviruses (Ads) is significantly influenced by the binding of various blood factors. To investigate differences in their binding, we conducted cryo-EM analysis on complexes of ...The tropism of adenoviruses (Ads) is significantly influenced by the binding of various blood factors. To investigate differences in their binding, we conducted cryo-EM analysis on complexes of several human adenoviruses with human platelet factor-4 (PF4), coagulation factors FII (Prothrombin), and FX. While we observed EM densities for FII and FX bound to all the species-C adenoviruses examined, no densities were seen for PF4, even though PF4 can co-pellet with various Ads. Similar to FX, the γ-carboxyglutamic acid (Gla) domain of FII binds within the surface cavity of hexon trimers. While FII binds equally to species-C Ads: Ad5, Ad6, and Ad657, FX exhibits significantly better binding to Ad5 and Ad657 compared to Ad6. Although only the FX-Gla domain is observed at high-resolution (3.7 Å), the entire FX is visible at low-resolution bound to Ad5 in three equivalent binding modes consistent with the 3-fold symmetric hexon. Only the Gla and kringle-1 domains of FII are visible on all the species-C adenoviruses, where the rigid FII binds in an upright fashion, in contrast to the flexible and bent FX. These data suggest that differential binding of FII and FX may shield certain species-C adenoviruses differently against immune molecules, thereby modulating their tropism. |
 External links External links |  Nat Commun / Nat Commun /  PubMed:39528527 / PubMed:39528527 /  PubMed Central PubMed Central |
| Methods | EM (single particle) |
| Resolution | 3.61 - 5.01 Å |
| Structure data | EMDB-45675, PDB-9cli: EMDB-45726, PDB-9cln: EMDB-45729, PDB-9cls: EMDB-45737, PDB-9cm2: EMDB-45744, PDB-9cm9: EMDB-45751, PDB-9cmo:  EMDB-46615: Human adenovirus 5 (Ad5) in complex with coagulation Factor II (Prothrombin) at 4.13 resolution.  EMDB-46620: Human adenovirus 6 (Ad6) in complex with coagulation Factor FX at 5.0A resolution.  EMDB-46654: Human Adenovirus 6 (Ad6) and Coagulation Factor II (Prothrombin) complex at 3.7A resolution  EMDB-46660: Human Adenovirus 657 (Ad657) and Coagulation Factor X (FX)complex at 4.0A resolution  EMDB-46770: Human Adenovirus 657 (Ad657) and Coagulation factor II (Prothrombin) at 4.17A resolution.  EMDB-51372: Human Adenovirus 5 (Ad5) and Coagulation Factor X (FX) complex at 3.6A resolution. |
| Chemicals |  ChemComp-CA: |
| Source |
|
 Keywords Keywords | VIRUS / Adenovirus / Hexon / Coagulation factor X / Coagulation factor II / Prothrombin / Complex / Interactions / VIRAL PROTEIN |
 Movie
Movie Controller
Controller Structure viewers
Structure viewers About Yorodumi Papers
About Yorodumi Papers



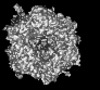

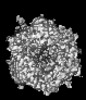

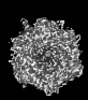

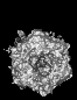




 human adenovirus 5
human adenovirus 5 homo sapiens (human)
homo sapiens (human)