+検索条件
-Structure paper
| タイトル | Fast native-SAD phasing for routine macromolecular structure determination. |
|---|---|
| ジャーナル・号・ページ | Nat. Methods, Vol. 12, Page 131-133, Year 2015 |
| 掲載日 | 2014年5月2日 (構造データの登録日) |
 著者 著者 | Weinert, T. / Olieric, V. / Waltersperger, S. / Panepucci, E. / Chen, L. / Zhang, H. / Zhou, D. / Rose, J. / Ebihara, A. / Kuramitsu, S. ...Weinert, T. / Olieric, V. / Waltersperger, S. / Panepucci, E. / Chen, L. / Zhang, H. / Zhou, D. / Rose, J. / Ebihara, A. / Kuramitsu, S. / Li, D. / Howe, N. / Schnapp, G. / Pautsch, A. / Bargsten, K. / Prota, A.E. / Surana, P. / Kottur, J. / Nair, D.T. / Basilico, F. / Cecatiello, V. / Pasqualato, S. / Boland, A. / Weichenrieder, O. / Wang, B.C. / Steinmetz, M.O. / Caffrey, M. / Wang, M. |
 リンク リンク |  Nat. Methods / Nat. Methods /  PubMed:25506719 PubMed:25506719 |
| 手法 | X線回折 |
| 解像度 | 2.133 - 2.704 Å |
| 構造データ |  PDB-4pgo:  PDB-4pii:  PDB-4r8t:  PDB-4r8u:  PDB-4tn8: 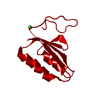 PDB-4tno:  PDB-4wab:  PDB-4wau:  PDB-4wbq:  PDB-4wbx: |
| 化合物 |  ChemComp-CL:  ChemComp-HOH:  ChemComp-IMD:  ChemComp-MG:  ChemComp-1FZ:  ChemComp-GSH: 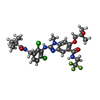 ChemComp-LVJ:  ChemComp-CA: |
| 由来 |
|
 キーワード キーワード | UNKNOWN FUNCTION / Sulfur SAD / SOUTHEAST COLLABORATORY FOR STRUCTURAL GENOMICS / SECSG / STRUCTURAL GENOMICS / HYDROLASE / Serine protease / Protease / NS2b / TRANSFERASE/DNA / nucleotidyl transferase / DNA Polymerase / TRANSFERASE-DNA complex / ELECTRON TRANSPORT / ISOMERASE / native-SAD / CANCER / DRUG TARGET / IN MESO CRYSTALLIZATION / INFLAMMATION / INHIBITOR / LEUKOTRIENE C4 SYNTHASE / LIPID METABOLISM / MEMBRANE-ASSOCIATED PROTEINS IN EICOSANOID AND GLUTATHIONE METABOLISM / MAPAG / MEMBRANE PROTEIN / MPGES1 / PAIN / MICROCRYSTAL / ANOMALOUS DISPERSION / SULFUR-SAD / S-SAD / CELL CYCLE / Mitosis / Kinetochore / CCAN / G-protein / RNA BINDING PROTEIN / OXIDOREDUCTASE / case studies / Swiss Light Source |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




 pyrococcus furiosus (古細菌)
pyrococcus furiosus (古細菌)
 japanese encephalitis virus (日本脳炎ウイルス)
japanese encephalitis virus (日本脳炎ウイルス)
 homo sapiens (ヒト)
homo sapiens (ヒト) neurospora crassa (菌類)
neurospora crassa (菌類)