-検索条件
-検索結果
検索 (著者・登録者: victor & t)の結果1,048件中、1から50件目までを表示しています

EMDB-18644: 
Cryo-EM structure of Streptococcus pneumoniae NADPH oxidase

EMDB-18645: 
Cryo-EM structure of Streptococcus pneumoniae NADPH oxidase in complex with NADPH

EMDB-18646: 
Cryo-EM structure of stably reduced Streptococcus pneumoniae NADPH oxidase in complex with NADH

EMDB-18647: 
Cryo-EM structure of Streptococcus pneumoniae NADPH oxidase F397A mutant in complex with NADPH

PDB-8qt6: 
Cryo-EM structure of Streptococcus pneumoniae NADPH oxidase

PDB-8qt7: 
Cryo-EM structure of Streptococcus pneumoniae NADPH oxidase in complex with NADPH

PDB-8qt9: 
Cryo-EM structure of stably reduced Streptococcus pneumoniae NADPH oxidase in complex with NADH

PDB-8qta: 
Cryo-EM structure of Streptococcus pneumoniae NADPH oxidase F397A mutant in complex with NADPH
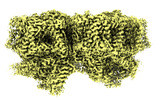
EMDB-41460: 
Structure of a mutated photosystem II complex reveals perturbation of the oxygen-evolving complex

PDB-8tow: 
Structure of a mutated photosystem II complex reveals perturbation of the oxygen-evolving complex

EMDB-36730: 
SARS-CoV-2 Spike RBD (dimer) in complex with two 2S-1244 nanobodies

EMDB-36735: 
Dimer of SARS-CoV-2 BA.2 spike and IBT-CoV144(C3 symmetry)

EMDB-36740: 
Dimer of SARS-CoV-2 BA.2 spike and IBT-CoV144(C1 symmetry)

PDB-8jys: 
SARS-CoV-2 Spike RBD (dimer) in complex with two 2S-1244 nanobodies

EMDB-44148: 
The cryo-EM structure of the H2A.Z-H3.3 double-variant nucleosome

PDB-9b3p: 
The cryo-EM structure of the H2A.Z-H3.3 double-variant nucleosome
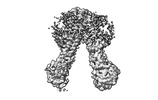
EMDB-41021: 
Transporter associated with antigen processing (TAP) in the apo state

PDB-8t46: 
Transporter associated with antigen processing (TAP) in the apo state

EMDB-18826: 
In situ sub-tomogram average of the E. coli 70S ribosome obtained using honeycomb gold supports

EMDB-41605: 
Protonated state of NorA at pH 5.0

EMDB-41606: 
NorA double mutant - E222QD307N at pH 7.5

EMDB-41607: 
NorA single mutant - E222Q at pH 7.5

EMDB-41608: 
NorA single mutant - D307N at pH 7.5

PDB-8tte: 
Protonated state of NorA at pH 5.0

PDB-8ttf: 
NorA double mutant - E222QD307N at pH 7.5

PDB-8ttg: 
NorA single mutant - E222Q at pH 7.5

PDB-8tth: 
NorA single mutant - D307N at pH 7.5

EMDB-42528: 
CryoEM structure of A/Perth/16/2009 H3 in complex with flu HA central stem VH1-18 antibody UCA6

EMDB-42529: 
CryoEM structure of A/Michigan/45/2015 H1 in complex with flu HA central stem VH1-18 antibody 09-1B12

EMDB-42530: 
CryoEM structure of A/Michigan/45/2015 H1 in complex with flu HA central stem VH1-18 antibody UCA6_N55T

EMDB-42531: 
CryoEM structure of A/Perth/16/2009 H3 in complex with polyclonal Fab from mice immunized with H3 stem nanoparticles-15 days post immunization

EMDB-42532: 
CryoEM structure of A/Perth/16/2009 H3 in complex with polyclonal Fab from mice immunized with H3 stem nanoparticles-28 days post immunization

EMDB-42533: 
CryoEM structure of A/Shanghai/1/2013 H7 in complex with polyclonal Fab from mice immunized with H7 stem nanoparticles-15 days post-immunization

EMDB-42534: 
CryoEM structure of A/Shanghai/1/2013 H7 in complex with polyclonal Fab from mice immunized with H7 stem nanoparticles-28 days post immunization

EMDB-42535: 
CryoEM structure of A/Perth/16/2009 H3

EMDB-42536: 
CryoEM map of A/Shanghai/1/2013 H7 HA

PDB-8ut3: 
CryoEM structure of A/Perth/16/2009 H3 in complex with flu HA central stem VH1-18 antibody UCA6

PDB-8ut4: 
CryoEM structure of A/Michigan/45/2015 H1 in complex with flu HA central stem VH1-18 antibody 09-1B12

PDB-8ut5: 
CryoEM structure of A/Michigan/45/2015 H1 in complex with flu HA central stem VH1-18 antibody UCA6_N55T

PDB-8ut6: 
CryoEM structure of A/Perth/16/2009 H3 in complex with polyclonal Fab from mice immunized with H3 stem nanoparticles-15 days post immunization

PDB-8ut7: 
CryoEM structure of A/Perth/16/2009 H3 in complex with polyclonal Fab from mice immunized with H3 stem nanoparticles-28 days post immunization

PDB-8ut8: 
CryoEM structure of A/Shanghai/1/2013 H7 in complex with polyclonal Fab from mice immunized with H7 stem nanoparticles-15 days post-immunization

PDB-8ut9: 
CryoEM structure of A/Shanghai/1/2013 H7 in complex with polyclonal Fab from mice immunized with H7 stem nanoparticles-28 days post immunization

EMDB-43017: 
60S ribosome biogenesis intermediate (Dbp10 pre-catalytic structure - Overall map)

EMDB-43018: 
60S ribosome biogenesis intermediate (Dbp10 pre-catalytic structure - PTC Local map)

EMDB-43019: 
60S ribosome biogenesis intermediate (Dbp10 pre-catalytic structure - Local map L1 region)

EMDB-43020: 
60S ribosome biogenesis intermediate (Dbp10 pre-catalytic structure - Local map Rrp14/Rrp15/Ssf1 region)

EMDB-43021: 
60S ribosome biogenesis intermediate (Dbp10 catalytic structure - Overall map)

EMDB-43022: 
60S ribosome biogenesis intermediate (Dbp10 catalytic structure - Dbp10 Local map)

EMDB-43023: 
60S ribosome biogenesis intermediate (Dbp10 catalytic structure - Low-pass filtered locally refined map)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
