-検索条件
-検索結果
検索 (著者・登録者: tajkhorshid & e)の結果108件中、1から50件目までを表示しています

EMDB-42480: 
Cryo-EM reconstruction of Staphylococcus aureus Oleate hydratase (OhyA) dimer with an ordered C-terminal membrane-association domain

EMDB-42484: 
Cryo-EM reconstruction of Staphylococcus aureus oleate hydratase (OhyA) dimer with a disordered C-terminal membrane-association domain

PDB-8ur3: 
Cryo-EM reconstruction of Staphylococcus aureus Oleate hydratase (OhyA) dimer with an ordered C-terminal membrane-association domain

PDB-8ur6: 
Cryo-EM reconstruction of Staphylococcus aureus oleate hydratase (OhyA) dimer with a disordered C-terminal membrane-association domain
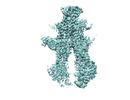
EMDB-41004: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to ATP: BmrCD_IF-ATP2

PDB-8t3k: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to ATP: BmrCD_IF-ATP2
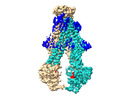
EMDB-29362: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to ATP: BmrCD_IF-ATP

PDB-8fpf: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to ATP: BmrCD_IF-ATP

EMDB-29087: 
Heterodimeric ABC transporter BmrCD in the occluded conformation bound to ATP: BmrCD_OC-ATP

EMDB-29297: 
Structure0915

EMDB-40908: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to substrate and ATP: BmrCD_IF-1HT/ATP

EMDB-40974: 
BmrCD_OC-ADPVi

EMDB-41058: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to substrate and ADPVi: BmrCD_IF-HT/ADPVi

PDB-8fhk: 
Heterodimeric ABC transporter BmrCD in the occluded conformation bound to ATP: BmrCD_OC-ATP

PDB-8fmv: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to substrate and ATP: BmrCD_IF-2HT/ATP

PDB-8szc: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to substrate and ATP: BmrCD_IF-1HT/ATP

PDB-8t1p: 
Heterodimeric ABC transporter BmrCD in the occluded conformation bound to ADPVi: BmrCD_OC-ADPVi

EMDB-16069: 
ABCG2 turnover-1 state with tariquidar bound

EMDB-16075: 
ABCG2 turnover-2 state with tariquidar bound

PDB-8bht: 
ABCG2 turnover-1 state with tariquidar bound

PDB-8bi0: 
ABCG2 turnover-2 state with tariquidar bound

EMDB-28041: 
Cryo-EM Structure of human ABCA7 in BPL/Ch Nanodiscs

EMDB-28044: 
Human ABCA7 in BPL/Ch nanodiscs (Map 2)

EMDB-28047: 
Cryo-EM Structure of human ABCA7 in PE/Ch nanodiscs

EMDB-28050: 
Cryo-EM structure of human ABCA7 in Digitonin

EMDB-28451: 
Cryo-EM Structure of Nanodisc reconstituted human ABCA7 EQ mutant in ATP bound closed state

PDB-8edw: 
Cryo-EM Structure of human ABCA7 in BPL/Ch Nanodiscs

PDB-8ee6: 
Cryo-EM Structure of human ABCA7 in PE/Ch nanodiscs

PDB-8eeb: 
Cryo-EM structure of human ABCA7 in Digitonin

PDB-8eop: 
Cryo-EM Structure of Nanodisc reconstituted human ABCA7 EQ mutant in ATP bound closed state

EMDB-26741: 
Structure of Expanded C. elegans TMC-1 complex

EMDB-26742: 
Structure of Contracted C. elegans TMC-1 complex

EMDB-26743: 
Structure of C. elegans TMC-1 complex with ARRD-6

PDB-7usw: 
Structure of Expanded C. elegans TMC-1 complex

PDB-7usx: 
Structure of Contracted C. elegans TMC-1 complex

PDB-7usy: 
Structure of C. elegans TMC-1 complex with ARRD-6

EMDB-30817: 
High Resolution Cryo-EM Structure of Cytochrome bo3 from E. Coli Reveals High Affinity Quinol Binding Site and Interactions of Protein with Lipids

EMDB-30818: 
High Resolution Cryo-EM Structure of Cytochrome bo3 from E. Coli Reveals High Affinity Quinol Binding Site and Interactions of Protein with Lipids

EMDB-30819: 
High Resolution Cryo-EM Structure of Cytochrome bo3 from E. Coli Reveals High Affinity Quinol Binding Site and Interactions of Protein with Lipids

EMDB-24265: 
E. coli cytochrome bo3 in MSP nanodisc

PDB-7n9z: 
E. coli cytochrome bo3 in MSP nanodisc

EMDB-30471: 
2.55-Angstrom Cryo-EM structure of Cytochrome bo3 from Escherichia coli in Native Membrane

EMDB-30474: 
2.55-Angstrom Cryo-EM structure of Cytochrome bo3 from Escherichia coli in Native Membrane

EMDB-30475: 
Ubiquinol Binding Site of Cytochrome bo3 from Escherichia coli

PDB-7cub: 
2.55-Angstrom Cryo-EM structure of Cytochrome bo3 from Escherichia coli in Native Membrane

PDB-7cuq: 
2.55-Angstrom Cryo-EM structure of Cytochrome bo3 from Escherichia coli in Native Membrane

PDB-7cuw: 
Ubiquinol Binding Site of Cytochrome bo3 from Escherichia coli

EMDB-23329: 
Structure of human prestin in the presence of NaCl

EMDB-23331: 
Structure of human Prestin in nanodisc in the presence of NaCl

EMDB-23334: 
Structure of human prestin in the presence of sodium salicylate and sodium sulfate
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
