-検索条件
-検索結果
検索 (著者・登録者: nayak & ar)の結果61件中、1から50件目までを表示しています

EMDB-40799: 
Cryo-EM consensus map of a double loaded human UBA7-UBE2L6-ISG15 thioester mimetic complex

EMDB-27712: 
Recombinant mouse RyR2 triple phosphonull mutant S2807A/S2813A/S2030A in complex with FKBP12.6 and nanodisc under closed-state conditions

EMDB-27711: 
Recombinant mouse RyR2 triple phosphomimetic mutant S2807D/S2813D/S2030D in complex with FKBP12.6 and nanodisc under closed-state conditions

EMDB-27746: 
Recombinant mouse RyR2 triple phosphomimetic mutant S2807D/S2813D/S2030D in complex with FKBP12.6 and nanodisc under open-state conditions

EMDB-29745: 
Cryo-EM structure of the Mismatch Sensing Complex (I) of Human Mitochondrial DNA Polymerase Gamma

EMDB-29746: 
Cryo-EM structure of the Mismatch Uncoupling Complex (II) of Human Mitochondrial DNA Polymerase Gamma
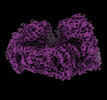
EMDB-29747: 
Cryo-EM structure of the Wedge Alignment Complex (VIII) of Human Mitochondrial DNA Polymerase Gamma
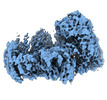
EMDB-29748: 
Cryo-EM structure of the Primer Separation Complex (IX) of Human Mitochondrial DNA Polymerase Gamma

EMDB-29749: 
Cryo-EM structure of the Mismatch Locking Complex (III) of Human Mitochondrial DNA Polymerase Gamma

EMDB-29750: 
Cryo-EM structure of the Guide loop Engagement Complex (VI) of Human Mitochondrial DNA Polymerase Gamma

EMDB-29751: 
Cryo-EM structure of the Guide loop Engagement Complex (IV) of Human Mitochondrial DNA Polymerase Gamma

EMDB-29752: 
Cryo-EM structure of the Guide loop Engagement Complex (V) of Human Mitochondrial DNA Polymerase Gamma
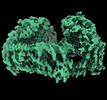
EMDB-41091: 
Cryo-EM structure of the Backtracking Initiation Complex (VII) of Human Mitochondrial DNA Polymerase Gamma

PDB-8g5i: 
Cryo-EM structure of the Mismatch Sensing Complex (I) of Human Mitochondrial DNA Polymerase Gamma

PDB-8g5j: 
Cryo-EM structure of the Mismatch Uncoupling Complex (II) of Human Mitochondrial DNA Polymerase Gamma

PDB-8g5k: 
Cryo-EM structure of the Wedge Alignment Complex (VIII) of Human Mitochondrial DNA Polymerase Gamma

PDB-8g5l: 
Cryo-EM structure of the Primer Separation Complex (IX) of Human Mitochondrial DNA Polymerase Gamma

PDB-8g5m: 
Cryo-EM structure of the Mismatch Locking Complex (III) of Human Mitochondrial DNA Polymerase Gamma

PDB-8g5n: 
Cryo-EM structure of the Guide loop Engagement Complex (VI) of Human Mitochondrial DNA Polymerase Gamma

PDB-8g5o: 
Cryo-EM structure of the Guide loop Engagement Complex (IV) of Human Mitochondrial DNA Polymerase Gamma

PDB-8g5p: 
Cryo-EM structure of the Guide loop Engagement Complex (V) of Human Mitochondrial DNA Polymerase Gamma

PDB-8t7e: 
Cryo-EM structure of the Backtracking Initiation Complex (VII) of Human Mitochondrial DNA Polymerase Gamma

EMDB-40407: 
Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thioester mimetic complex (Form 2)

EMDB-40408: 
Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thioester mimetic complex (Form 1)

EMDB-40409: 
Cryo-EM structure of a single loaded human UBA7-UBE2L6-ISG15 adenylate complex

EMDB-40782: 
Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thioester mimetic complex from a composite map

PDB-8se9: 
Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thioester mimetic complex (Form 2)

PDB-8sea: 
Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thioester mimetic complex (Form 1)

PDB-8seb: 
Cryo-EM structure of a single loaded human UBA7-UBE2L6-ISG15 adenylate complex

PDB-8sv8: 
Cryo-EM structure of a double loaded human UBA7-UBE2L6-ISG15 thioester mimetic complex from a composite map
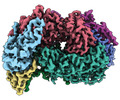
EMDB-29978: 
McrD binds asymmetrically to methyl-coenzyme M reductase improving active site accessibility during assembly
EMDB-29979: 
Apo-apo MCR assembly intermediate

PDB-8gf5: 
McrD binds asymmetrically to methyl-coenzyme M reductase improving active site accessibility during assembly

PDB-8gf6: 
Apo-apo MCR assembly intermediate

EMDB-34167: 
CryoEM structure of cytosol-facing, substrate-bound ratGAT1

PDB-8gnk: 
CryoEM structure of cytosol-facing, substrate-bound ratGAT1

EMDB-26610: 
Cryo-EM structure of rabbit RyR1 in the presence of high Mg2+ and AMP-PCP in nanodisc

EMDB-27795: 
Cryo-EM structure of bundle-forming pilus extension ATPase from E. coli in the presence of AMP-PNP (class-1)

EMDB-27796: 
Cryo-EM structure of bundle-forming pilus extension ATPase from E.coli in the presence of AMP-PNP (class-2)

EMDB-27797: 
Cryo-EM structure of bundle-forming pilus extension ATPase from E.coli in the presence of ADP

EMDB-26262: 
SARS-Cov2 S protein structure in complex with neutralizing monoclonal antibody 002-S21F2

EMDB-26669: 
SARS-Cov2 Omicron varient S protein structure in complex with neutralizing monoclonal antibody 002-S21F2

PDB-7u0p: 
SARS-Cov2 S protein structure in complex with neutralizing monoclonal antibody 002-S21F2

PDB-7upl: 
SARS-Cov2 Omicron varient S protein structure in complex with neutralizing monoclonal antibody 002-S21F2

EMDB-25828: 
Rabbit RyR1 with AMP-PCP and high Ca2+ embedded in nanodisc in inactivated conformation (Dataset-A)

EMDB-25829: 
Rabbit RyR1 with AMP-PCP and high Ca2+ embedded in nanodisc in open conformation

EMDB-25830: 
Rabbit RyR1 with AMP-PCP and high Ca2+ embedded in nanodisc in closed-inactivated conformation class 2 (Dataset-A)

EMDB-25831: 
Rabbit RyR1 with AMP-PCP and high Ca2+ embedded in nanodisc in closed-inactivated conformation class 1(Dataset-A)

EMDB-25832: 
Rabbit RyR1 with AMP-PCP and high Ca2+ embedded in nanodisc in closed-inactivated conformation class 3 (Dataset-A)

EMDB-25833: 
Rabbit RyR1 with AMP-PCP and high Ca2+ embedded in nanodisc in inactivated conformation (Dataset-B)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
