-検索条件
-検索結果
検索 (著者・登録者: mueller & j)の結果263件中、1から50件目までを表示しています

EMDB-18418: 
Human DNA methyltransferase 1

EMDB-50795: 
Human DNA methyltransferase 1 in non-productive complex with DNA
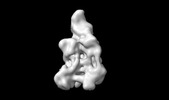
EMDB-50801: 
Human DNA methyltransferase 1 bound to H3Ub2-peptide
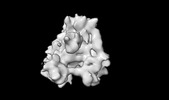
EMDB-50802: 
Human DNA methyltransferase 1 productive DNA complex in presence of H3Ub2-peptide

EMDB-41433: 
Escherichia coli RNA polymerase unwinding intermediate (I1a) at the lambda PR promoter

EMDB-41437: 
Escherichia coli RNA polymerase unwinding intermediate (I1d) at the lambda PR promoter

EMDB-41439: 
Escherichia coli RNA polymerase unwinding intermediate (I1b) at the lambda PR promoter

EMDB-41448: 
Escherichia coli RNA polymerase unwinding intermediate (I1c) at the lambda PR promoter

EMDB-41456: 
Escherichia coli RNA polymerase closed complex intermediate at the lambda PR promoter

PDB-8to1: 
Escherichia coli RNA polymerase unwinding intermediate (I1a) at the lambda PR promoter

PDB-8to6: 
Escherichia coli RNA polymerase unwinding intermediate (I1d) at the lambda PR promoter

PDB-8to8: 
Escherichia coli RNA polymerase unwinding intermediate (I1b) at the lambda PR promoter

PDB-8toe: 
Escherichia coli RNA polymerase unwinding intermediate (I1c) at the lambda PR promoter

PDB-8tom: 
Escherichia coli RNA polymerase closed complex intermediate at the lambda PR promoter

EMDB-19163: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

EMDB-19164: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

EMDB-19165: 
Trimeric HSV-2F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

EMDB-19166: 
Trimeric HSV-2G gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

PDB-8rgz: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

PDB-8rh0: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

PDB-8rh1: 
Trimeric HSV-2F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

PDB-8rh2: 
Trimeric HSV-2G gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

EMDB-17796: 
human RYBP-PRC1 bound to H2AK118ub1 nucleosome

PDB-8pp6: 
human RYBP-PRC1 bound to H2AK118ub1 nucleosome

EMDB-17797: 
human RYBP-PRC1 bound to mononucleosome

PDB-8pp7: 
human RYBP-PRC1 bound to mononucleosome
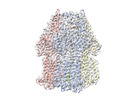
EMDB-17350: 
Single particle cryo-EM co-structure of Klebsiella pneumoniae AcrB with the BDM91288 efflux pump inhibitor at 2.97 Angstrom resolution

PDB-8p1i: 
Single particle cryo-EM co-structure of Klebsiella pneumoniae AcrB with the BDM91288 efflux pump inhibitor at 2.97 Angstrom resolution

EMDB-16242: 
Cryo-EM structure of RANBP10-CTLH SR4 complex

EMDB-16243: 
Cryo-EM map of ARMC8-specific nanobody bound to CTLH-SR4

EMDB-17804: 
Bat-Hp-CoV Nsp1 and eIF1 bound to the human 40S small ribosomal subunit

EMDB-17805: 
MERS-CoV Nsp1 bound to the human 43S pre-initiation complex

PDB-8ppk: 
Bat-Hp-CoV Nsp1 and eIF1 bound to the human 40S small ribosomal subunit

PDB-8ppl: 
MERS-CoV Nsp1 bound to the human 43S pre-initiation complex
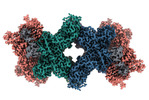
EMDB-16878: 
Cryo-EM structure of the electron bifurcating transhydrogenase StnABC complex from Sporomusa Ovata (state 2)
EMDB-16879: 
Cryo-EM structure of the electron bifurcating transhydrogenase StnABC complex from Sporomusa Ovata (state 1)

PDB-8oh5: 
Cryo-EM structure of the electron bifurcating transhydrogenase StnABC complex from Sporomusa Ovata (state 2)

PDB-8oh9: 
Cryo-EM structure of the electron bifurcating transhydrogenase StnABC complex from Sporomusa Ovata (state 1)

EMDB-18194: 
Cryo-electron tomogram of GEM2-labelled Mito-EGFP in HeLa cells

EMDB-16303: 
In situ subtomogram average of GEM2 particles in human cells

EMDB-33887: 
Rubisco from Phaeodactylum tricornutum bound to PYCO1(452-592)

EMDB-35158: 
Rubisco from Phaeodactylum tricornutum

EMDB-35159: 
PYCO1(452-592) from Phaeodactylum tricornutum

EMDB-35166: 
Rubisco from Phaeodactylum tricornutum bound to PYCO1(452-592)

PDB-7yk5: 
Rubisco from Phaeodactylum tricornutum bound to PYCO1(452-592)

EMDB-28783: 
EsN-dhsU36mm2 full map (map 1)

EMDB-28784: 
EsN-dhsU36mm2 local map (map 2)

EMDB-28785: 
EsN-dhsU36mm2 composite map
EMDB-28786: 
EsN-dhsU36duplex full map (map 1)

EMDB-28790: 
EsN-dhsU36duplex local map (map 2)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
