-検索条件
-検索結果
検索 (著者・登録者: maria & ts)の結果299件中、1から50件目までを表示しています

EMDB-50358: 
In vitro-induced genome-releasing intermediate of Rhodobacter microvirus Ebor computed with C5 symmetry

EMDB-18592: 
E.coli DNA gyrase in complex with 217 bp substrate DNA and LEI-800

PDB-8qqi: 
E.coli DNA gyrase in complex with 217 bp substrate DNA and LEI-800

EMDB-50356: 
Empty capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-50357: 
Native capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-50359: 
Rhodobacter microvirus Ebor attached to B10 host cell reconstructed by single particle analysis with applied C5 symmetry

EMDB-50360: 
Rhodobacter microvirus Ebor attached to the outer membrane vesicle

EMDB-50361: 
Rhodobacter microvirus Ebor attached to the host cell reconstructed by subtomogram averaging

PDB-9ffg: 
Empty capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

PDB-9ffh: 
Native capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-38372: 
SARS-CoV-2 Omicron BQ.1.1 Variant Spike Protein Complexed with MO11 Fab

PDB-8xi6: 
SARS-CoV-2 Omicron BQ.1.1 Variant Spike Protein Complexed with MO11 Fab

EMDB-19132: 
Structure of dynein-2 intermediate chain DYNC2I2 (WDR34) in complex with dynein-2 heavy chain DYNC2H1.

EMDB-19133: 
Structure of dynein-2 intermediate chain DYNC2I1 (WDR60) in complex with the dynein-2 heavy chain DYNC2H1.

PDB-8rgg: 
Structure of dynein-2 intermediate chain DYNC2I2 (WDR34) in complex with dynein-2 heavy chain DYNC2H1.

PDB-8rgh: 
Structure of dynein-2 intermediate chain DYNC2I1 (WDR60) in complex with the dynein-2 heavy chain DYNC2H1.

EMDB-42601: 
CryoEM structure of Kappa Opioid Receptor bound to a semi-peptide and Gi1
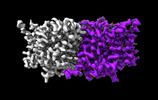
EMDB-29883: 
CLC-ec1 E202Y at pH 4.5 100mM Cl Turn
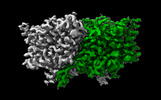
EMDB-29884: 
CLC-ec1 R230C/L249C/C85A at pH 4.5 100mM Cl Swap
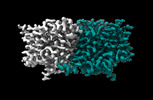
EMDB-29885: 
CLC-ec1 R230C/L249C/C85A at pH 4.5 100mM Cl Turn
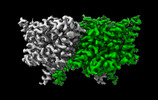
EMDB-29890: 
CLC-ec1 L25C/A450C/C85A at pH 4.5 100mM Cl Intermediate
EMDB-29899: 
CLC-ec1 L25C/A450C/C85A at pH 4.5 100mM Cl Twist

PDB-8ga0: 
CLC-ec1 E202Y at pH 4.5 100mM Cl Turn

PDB-8ga1: 
CLC-ec1 R230C/L249C/C85A at pH 4.5 100mM Cl Swap

PDB-8ga3: 
CLC-ec1 R230C/L249C/C85A at pH 4.5 100mM Cl Turn

PDB-8ga5: 
CLC-ec1 L25C/A450C/C85A at pH 4.5 100mM Cl Intermediate

PDB-8gah: 
CLC-ec1 L25C/A450C/C85A at pH 4.5 100mM Cl Twist

EMDB-17659: 
ACAD9-WT in complex with ECSIT-CTER

EMDB-17660: 
Cryo-EM structure of human ACAD9-S191A

EMDB-17661: 
ACAD9 homodimer WT

PDB-8phe: 
ACAD9-WT in complex with ECSIT-CTER

PDB-8phf: 
Cryo-EM structure of human ACAD9-S191A

EMDB-29026: 
CryoEM structure of Kappa Opioid Receptor bound to a semi-peptide and Gi1

PDB-8feg: 
CryoEM structure of Kappa Opioid Receptor bound to a semi-peptide and Gi1

EMDB-41265: 
Cryo-EM structure of the Tripartite ATP-independent Periplasmic (TRAP) transporter SiaQM from Haemophilus influenzae (parallel dimer)

EMDB-41266: 
Cryo-EM structure of the Tripartite ATP-independent Periplasmic (TRAP) transporter SiaQM from Haemophilus influenzae (antiparallel dimer)

PDB-8thi: 
Cryo-EM structure of the Tripartite ATP-independent Periplasmic (TRAP) transporter SiaQM from Haemophilus influenzae (parallel dimer)

PDB-8thj: 
Cryo-EM structure of the Tripartite ATP-independent Periplasmic (TRAP) transporter SiaQM from Haemophilus influenzae (antiparallel dimer)

EMDB-16805: 
Cryo-EM structure of PcrV/Fab(30-B8)

EMDB-16807: 
Cryo-EM structure of PcrV/Fab(11-E5)

PDB-8cr9: 
Cryo-EM structure of PcrV/Fab(30-B8)

PDB-8crb: 
Cryo-EM structure of PcrV/Fab(11-E5)

EMDB-15985: 
Small molecule positive allosteric modulation of homomeric kainate receptors GluK1-3: Development of screening assays and insight into GluK3 structure

EMDB-15986: 
Ionotropic glutamate receptor K3 with glutamate and BPAM

EMDB-17030: 
Helical nucleocapsid of the Respiratory Syncytial Virus

EMDB-17031: 
Double-ring nucleocapsid of the Respiratory Syncytial Virus

EMDB-17034: 
Helical nucleocapsid of the N1-370 mutant of the human Respiratory Syncytial Virus

EMDB-17035: 
Subsection of a helical nucleocapsid of the Respiratory Syncytial Virus

EMDB-17036: 
Double-headed nucleocapsid of the human Respiratory Syncytial Virus

EMDB-17037: 
Ring-capped nucleocapsid of the Respiratory Syncytial Virus
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
