-検索条件
-検索結果
検索 (著者・登録者: ling & sl)の結果250件中、1から50件目までを表示しています
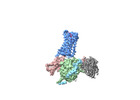
EMDB-39901: 
cryo-EM structure of the octreotide-bound SSTR5-Gi complex
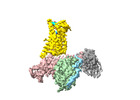
EMDB-39931: 
Cryo-EM structure of the pasireotide-bound SSTR5-Gi complex

EMDB-18592: 
E.coli DNA gyrase in complex with 217 bp substrate DNA and LEI-800

PDB-8qqi: 
E.coli DNA gyrase in complex with 217 bp substrate DNA and LEI-800

EMDB-40825: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

PDB-8sx3: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

EMDB-41277: 
Cryo-EM structure of a SUR1/Kir6.2-Q52R ATP-sensitive potassium channel in the presence of PIP2 in the open conformation

EMDB-41278: 
Cryo-EM structure of a SUR1/Kir6.2-Q52R ATP-sensitive potassium channel in the presence of PIP2 in the open conformation

EMDB-43766: 
Kir6.2-Q52R/SUR1 apo closed channel

EMDB-19024: 
Structure of the PNMA2 capsid

EMDB-19025: 
Structure of the five-fold capsomer of the PNMA2 capsid

EMDB-19026: 
Structure of the three-fold capsomer of the PNMA2 capsid

EMDB-19027: 
Structure of the two-fold capsomer of the PNMA2 capsid

EMDB-41374: 
Antibody N3-1 bound to RBDs in the up and down conformations
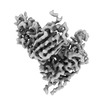
EMDB-41382: 
Antibody N3-1 bound to RBD in the up conformation
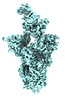
EMDB-41399: 
Antibody N3-1 bound to SARS-CoV-2 spike

PDB-8tm1: 
Antibody N3-1 bound to RBDs in the up and down conformations

PDB-8tma: 
Antibody N3-1 bound to RBD in the up conformation

EMDB-37716: 
Human calcium-sensing receptor bound with cinacalcet in detergent

EMDB-37724: 
Human calcium-sensing receptor(CaSR) bound to cinacalcet in complex with Gq protein

EMDB-16328: 
Outer membrane attachment porin OmpM1 from Veillonella parvula

EMDB-16332: 
Outer membrane attachment porin OmpM1 from Veillonella parvula, native

EMDB-16333: 
Outer membrane attachment porin OmpM1 from Veillonella parvula, C3 symmetry

PDB-8bym: 
Outer membrane attachment porin OmpM1 from Veillonella parvula

PDB-8bys: 
Outer membrane attachment porin OmpM1 from Veillonella parvula, native

PDB-8byt: 
Outer membrane attachment porin OmpM1 from Veillonella parvula, C3 symmetry

EMDB-28036: 
Cryo-EM structure of the full-length human NF1 dimer

PDB-8edm: 
Cryo-EM structure of the full-length human NF1 dimer

EMDB-17383: 
Tau filaments extracted from human brain with the DeltaK281 mutation in MAPT

EMDB-17539: 
Cryo-EM structure of dimeric UBR5

EMDB-17540: 
Cryo-EM structure of full-length human UBR5 (homotetramer)

EMDB-17542: 
Negative stain map of UBR5 (dimer) in complex with RARA/RXRA

EMDB-15288: 
Substrate-free levan utilisation machinery (utilisome)

EMDB-15289: 
Levan utilisation machinery (utilisome) with levan fructo-oligosaccharides DP 8-12

EMDB-15290: 
Core SusCD transporter units from the levan utilisome with levan fructo-oligosaccharides DP 8-12

EMDB-15291: 
Inactive levan utilisation machinery (utilisome) in the presence of levan fructo-oligosaccharides DP 15-25

EMDB-15292: 
Core SusCD transporter units from the inactive levan utilisome in the presence of levan fructo-oligosaccharides DP 15-25

EMDB-15293: 
Consensus reconstruction of the dextran utilisation system

PDB-8a9y: 
Substrate-free levan utilisation machinery (utilisome)

PDB-8aa0: 
Levan utilisation machinery (utilisome) with levan fructo-oligosaccharides DP 8-12

PDB-8aa1: 
Core SusCD transporter units from the levan utilisome with levan fructo-oligosaccharides DP 8-12

PDB-8aa2: 
Inactive levan utilisation machinery (utilisome) in the presence of levan fructo-oligosaccharides DP 15-25

PDB-8aa3: 
Core SusCD transporter units from the inactive levan utilisome in the presence of levan fructo-oligosaccharides DP 15-25

PDB-8aa4: 
SusC components of the dextran utilisation system (utilisome)

EMDB-33497: 
Neurokinin A bound to active human neurokinin 2 receptor in complex with G324
EMDB-29502: 
Fast and versatile sequence- independent protein docking for nanomaterials design using RPXDock

PDB-8fwd: 
Fast and versatile sequence- independent protein docking for nanomaterials design using RPXDock

EMDB-27826: 
Cryo-EM structure of the full-length human NF1 dimer

PDB-8e20: 
Cryo-EM structure of the full-length human NF1 dimer

EMDB-29396: 
Antibody vFP53.02 in complex with HIV-1 envelope trimer BG505 DS-SOSIP
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
