-検索条件
-検索結果
検索 (著者・登録者: li & ht)の結果1,208件中、1から50件目までを表示しています

EMDB-44965: 
Sub-tomogram average of the RSV M lattice from native virions released from RSV-infected BEAS-2B cells cultured on EM grids

EMDB-44966: 
Sub-tomogram average of a pair of RSV F trimers from native virions released from RSV-infected BEAS-2B cells cultured on EM grids

EMDB-44968: 
Sub-tomogram average of two pairs of RSV F trimers from the surface of native virions released from RSV-infected BEAS-2B cells cultured on EM grids

EMDB-44969: 
Sub-tomogram average of two pairs of RSV F trimers from the surface of native virions released from RSV-infected BEAS-2B cells cultured on EM grids

EMDB-44971: 
Sub-tomogram average of two pairs of RSV F trimers from the surface of native virions released from RSV-infected BEAS-2B cells cultured on EM grids

EMDB-45639: 
Cryo-EM structure of in-vitro alpha-synuclein fibril

PDB-9ck3: 
Cryo-EM structure of in-vitro alpha-synuclein fibril

EMDB-44599: 
Cryo-EM structure of the mammalian peptide transporter PepT2 bound to cefadroxil

EMDB-44600: 
Cryo-EM structure of the mammalian peptide transporter PepT2 bound to amoxicillin

EMDB-44601: 
Cryo-EM structure of the mammalian peptide transporter PepT2 bound to cloxacillin, pose 1

EMDB-44602: 
Cryo-EM structure of the mammalian peptide transporter PepT2 bound to cloxacillin, pose 2

EMDB-39025: 
Structure of HCoV-HKU1A spike in the functionally anchored-3up conformation with 3TMPRSS2

EMDB-39026: 
Local structure of HCoV-HKU1A spike in complex with TMPRSS2 and glycan

EMDB-39036: 
Structure of HCoV-HKU1C spike in the functionally anchored-1up conformation with 1TMPRSS2

EMDB-39037: 
Structure of HCoV-HKU1C spike in the functionally anchored-2up conformation with 2TMPRSS2

EMDB-39038: 
Structure of HCoV-HKU1C spike in the functionally anchored-3up conformation with 2TMPRSS2

EMDB-39039: 
Structure of HCoV-HKU1C spike in the functionally anchored-3up conformation with 3TMPRSS2

EMDB-39040: 
Local structure of HCoV-HKU1C spike in complex with TMPRSS2 and glycan

EMDB-39041: 
Structure of HCoV-HKU1C spike in the inactive-closed conformation

EMDB-39042: 
Structure of HCoV-HKU1C spike in the inactive-1up conformation

EMDB-39043: 
Structure of HCoV-HKU1C spike in the inactive-2up conformation

EMDB-39044: 
Structure of HCoV-HKU1C spike in the glycan-activated-closed conformation

EMDB-39045: 
Structure of HCoV-HKU1C spike in the glycan-activated-1up conformation

EMDB-39046: 
Structure of HCoV-HKU1C spike in the glycan-activated-2up conformation

EMDB-39047: 
Structure of HCoV-HKU1C spike in the glycan-activated-3up conformation

EMDB-39048: 
Local structure of HCoV-HKU1C spike in complex with glycan

EMDB-16917: 
Human apo pseudouridine synthase 3 (PUS3)

EMDB-16926: 
Human pseudouridine synthase 3 and tRNA-Gln
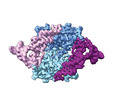
EMDB-19830: 
Human pseudouridine synthase 3 (PUS3 R116A mutant) and two tRNA-Gln

EMDB-19831: 
Human pseudouridine synthase 3 (PUS3 R116A mutant) and one tRNA-Gln
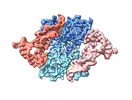
EMDB-19832: 
Human pseudouridine synthase 3 (PUS3 D118A mutant) and two tRNA-Arg
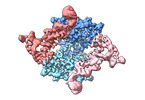
EMDB-19833: 
Human pseudouridine synthase 3 (PUS3 D118A mutant) and two pre-tRNA-Arg

EMDB-19834: 
Human pseudouridine synthase 3 (PUS3 R116A mutant)

EMDB-19835: 
Human pseudouridine synthase 3 (PUS3 K119A mutant)

EMDB-19836: 
Human pseudouridine synthase 3 (wild type)

PDB-8okd: 
Human pseudouridine synthase 3 and tRNA-Gln

PDB-9enb: 
Human pseudouridine synthase 3 (PUS3 R116A mutant) and two tRNA-Gln

PDB-9enc: 
Human pseudouridine synthase 3 (PUS3 R116A mutant) and one tRNA-Gln

PDB-9ene: 
Human pseudouridine synthase 3 (PUS3 D118A mutant) and two tRNA-Arg

PDB-9enf: 
Human pseudouridine synthase 3 (PUS3 D118A mutant) and two pre-tRNA-Arg

PDB-9f9q: 
Human apo pseudouridine synthase 3 (PUS3 D118A mutant)

EMDB-50580: 
SOLIST cryo-tomogram of native left ventricle mouse heart muscle #1

EMDB-50582: 
SOLIST native mouse heart muscle tomogram #2

EMDB-50358: 
In vitro-induced genome-releasing intermediate of Rhodobacter microvirus Ebor computed with C5 symmetry

EMDB-43706: 
Human DNA polymerase theta helicase domain in complex with ssDNA, dimer form

EMDB-43816: 
Human DNA polymerase theta helicase domain in complex with AMP-PNP, dimer form

EMDB-43817: 
Human DNA polymerase theta helicase domain dimer, apo-form

EMDB-43818: 
Human DNA polymerase theta helicase domain tetramer, apo-form

EMDB-45217: 
Human DNA polymerase theta helicase domain in microhomology annealed state 1, dimer form

EMDB-45218: 
Human DNA polymerase theta helicase domain in microhomology annealed state 2, dimer form
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
