-検索条件
-検索結果
検索 (著者・登録者: koh & fa)の結果93件中、1から50件目までを表示しています

EMDB-16032: 
Structure of mouse heavy chain apoferritin solved by subtomogram averaging of 100 tilt series in Warp/M

EMDB-15854: 
Structure of mouse heavy chain apoferritin solved by subtomogram averaging of 10 tilt series in Warp/M

EMDB-16799: 
Cryo-EM structure of the NINJ1 filament

PDB-8cqr: 
Cryo-EM structure of the NINJ1 filament

EMDB-13346: 
Single-particle cryo-EM reconstruction of the tetrahedral 24mer of Hsp17 from Caenorhabditis elegans

EMDB-15870: 
Subtomogram average of the human Sec61-TRAP-OSTA-translocon

EMDB-15871: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Decoding-Sampling State

EMDB-15872: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Classical Pre+ State

EMDB-15873: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Classical Pre State

EMDB-15874: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Rotated-1 Pre+ State
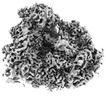
EMDB-15875: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Rotated-1 Pre State
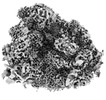
EMDB-15876: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Rotated-2 Pre State

EMDB-15877: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Translocation State

EMDB-15878: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Post State

EMDB-15879: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Non-Rotated Hibernating State

EMDB-15880: 
Subtomogram Average of Soluble and ER Membrane-Associated Ribosomes in the Rotated Hibernating State

EMDB-15884: 
Subtomogram Average of the Ribosome-Sec61-TRAP-OSTA-Translocon Complex

EMDB-15885: 
Subtomogram Average of the Ribosome-Sec61-TRAP-Translocon Complex
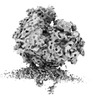
EMDB-15886: 
Subtomogram Average of the Ribosome-Sec61-TRAP-Multipass-Translocon Complex

EMDB-15887: 
Subtomogram Average of the Ribosome-Sec61-Multipass-Translocon Complex

EMDB-15888: 
Subtomogram Average of the Ribosome-EBP1 Complex

EMDB-15889: 
Subtomogram Average of the Idle Ribosome-Sec61-TRAP-OSTA-Translocon Complex

EMDB-15890: 
Subtomogram Average of the Sec61-TRAP-OSTA Translocon

EMDB-15891: 
Subtomogram Average of the Sec61-TRAP-OSTA-L1 Translocon
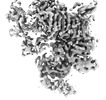
EMDB-15892: 
Subtomogram Average of the Sec61-TRAP-OSTA-L2 Translocon

EMDB-15893: 
CryoEM Structure of Extended eEF1A bound to the Ribosome in the Classical Pre State

EMDB-14025: 
In vitro assembled 297-394 tau filaments, 700 rpm (34b)

EMDB-14044: 
In vitro assembled 266/297 - 391 tau filaments with MgCl2 and NaCl (14b)

EMDB-14467: 
2.1A T20S Proteosome from 200kV Glacios with Selectris and Falcon 4

EMDB-14042: 
In vitro assembled 258-391 tau filaments with sodium azide, (41a)

EMDB-14316: 
In vitro assembled 266/297 - 391 tau filaments with NaHCO3 and NaCl (16a)

EMDB-14320: 
In vitro assembled 266/297 - 391 tau filaments with KCl (10b)

EMDB-14023: 
In vitro assembled tau filaments into Quadruple Helical Filaments type 1 (2c)

EMDB-14026: 
In vitro assembled 266/297 - 391 tau filaments with LiCl (9a)

EMDB-14027: 
In vitro assembled 266/297 - 391 tau filaments with LiCl (9b)

EMDB-14028: 
In vitro assembled 297-394 tau filaments in PBS (35d)

EMDB-14029: 
In vitro assembled 300-391 tau filaments in PBS (36a)

EMDB-14030: 
In vitro assembled 258-391 tau filaments, 700 rpm (38a)

EMDB-14038: 
In vitro assembled 266/297 - 391 tau filaments with KCl (10a)

EMDB-14039: 
In vitro assembled 258-391 tau filaments with heparan sulfate, 700 rpm (40a)

EMDB-14040: 
In vitro assembled 266/297 - 391 tau filaments with CuCl2 (12a)

EMDB-14041: 
In vitro assembled 258-391 tau filaments with phosphoglycerate, 700 rpm (39a)

EMDB-14043: 
In vitro assembled 297-408 S396D S400D T403D S404D tau filaments (42a)

EMDB-14045: 
In vitro assembled 258-391 tau filaments with phosphoglycerate, 700 rpm (39a)

EMDB-14046: 
In vitro assembled 266/297 - 391 tau filaments with ZnCl2 (11a)

EMDB-14047: 
In vitro assembled 266-391 S356D tau filaments with NaCl (45a)

EMDB-14053: 
in vitro assembled 266/297 - 391 tau filaments with MgCl2 and NaCl (14a)

EMDB-14054: 
In vitro assembled tau filaments with MgSO4 and NaCl (15a)

EMDB-14055: 
In vitro assembled 266-391 S356D tau filaments with KCl (44a)

EMDB-14056: 
In vitro assembled 266/297 - 391 tau filaments with MgSO4 and NaCl (15b)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
