-検索条件
-検索結果
検索 (著者・登録者: hsu & al)の結果257件中、1から50件目までを表示しています

EMDB-38216: 
Cryo-EM structure of SARS-CoV-2 S-BQ.1 in complex with antibody O5C2

PDB-8xbf: 
Cryo-EM structure of SARS-CoV-2 S-BQ.1 in complex with antibody O5C2

EMDB-38691: 
Thiamine-bound human SLC19A3

EMDB-38692: 
Pyridoxamine-bound human SLC19A3

EMDB-38693: 
Fedratinib-bound human SLC19A3

PDB-8xv2: 
Thiamine-bound human SLC19A3

PDB-8xv5: 
Pyridoxamine-bound human SLC19A3

PDB-8xv9: 
Fedratinib-bound human SLC19A3
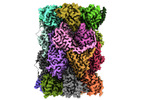
EMDB-29764: 
Structure of the Plasmodium falciparum 20S proteasome complexed with inhibitor TDI-8304
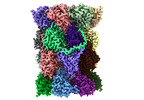
EMDB-29765: 
Structure of the Plasmodium falciparum 20S proteasome beta-6 A117D mutant complexed with inhibitor WLW-vs

EMDB-42148: 
Structure of human constitutive 20S proteasome complexed with the inhibitor TDI-8304

PDB-8g6e: 
Structure of the Plasmodium falciparum 20S proteasome complexed with inhibitor TDI-8304

PDB-8g6f: 
Structure of the Plasmodium falciparum 20S proteasome beta-6 A117D mutant complexed with inhibitor WLW-vs

PDB-8ud9: 
Structure of human constitutive 20S proteasome complexed with the inhibitor TDI-8304

EMDB-29172: 
Cryo-EM structure of Cryptococcus neoformans trehalose-6-phosphate synthase homotetramer in complex with uridine diphosphate and glucose-6-phosphate

PDB-8fhw: 
Cryo-EM structure of Cryptococcus neoformans trehalose-6-phosphate synthase homotetramer in complex with uridine diphosphate and glucose-6-phosphate

EMDB-41066: 
Human VMAT2 in complex with tetrabenazine

EMDB-41067: 
Human VMAT2 in complex with reserpine

EMDB-41068: 
Human VMAT2 in complex with serotonin

PDB-8t69: 
Human VMAT2 in complex with tetrabenazine

PDB-8t6a: 
Human VMAT2 in complex with reserpine

PDB-8t6b: 
Human VMAT2 in complex with serotonin
EMDB-41205: 
Cryo-electron tomography reconstruction of mammalian 80S ribosome in the non-rotated state with P, A/T-site tRNA, and eEF1A cofactor from EMPIAR-10064 dataset by constrained classification

EMDB-41207: 
Cryo-electron tomography reconstruction of mammalian 80S ribosome from EMPIAR-10064 in the translocation intermediates (TI) state with eEF2 cofactor and closed L1 by constrained classification.

EMDB-41210: 
Cryo-electron tomography reconstruction of mammalian 80S ribosome in the translocation intermediates (TI) state with eEF2 cofactor and open L1 from EMPIAR-10064 dataset by constrained classification.

EMDB-41211: 
Cryo-electron tomography reconstruction of mammalian 80S ribosome in the non-rotated state with P, A/T-site tRNA from EMPIAR-10064 dataset by constrained classification.

EMDB-41212: 
Cryo-electron tomography reconstruction of mammalian 80S ribosome in the non-rotated state with E, P, A/T-site tRNA and eEF1A cofactor from EMPIAR-10064 dataset by constrained classification.

EMDB-41220: 
Cryo-electron tomography reconstruction of M. pneumoniae 70S ribosome in decoding state with EF-Tu-tRNA and P-site tRNA from EMPIAR-10499 dataset by constrained classification.

EMDB-41221: 
Cryo-electron tomography reconstruction of M. pneumoniae 70S ribosome in classical non-rotated state with L1 open, obtained from EMPIAR-10499 dataset by constrained classification

EMDB-41222: 
Cryo-electron tomography reconstruction of M. pneumoniae 70S ribosome in the classical non-rotated state with half-closed L1 stalk, obtained from EMPIAR-10499 dataset by constrained classification

EMDB-41223: 
Cryo-electron tomography reconstruction of 80S ribosome in non-rotated state class 1, obtained from the EMPIAR-10987 dataset by constrained 3D classification.

EMDB-41224: 
Cryo-electron tomography reconstruction of 80S ribosome in non-rotated state class 2, obtained from the EMPIAR-10987 dataset by constrained 3D classification.

EMDB-41225: 
Cryo-electron tomography reconstruction of 80S ribosome in non-rotated state class 3, obtained from the EMPIAR-10987 dataset by constrained 3D classification.
EMDB-41226: 
Cryo-electron tomography reconstruction of 80S ribosome in non-rotated decoding state class 4, obtained from the EMPIAR-10987 dataset by constrained 3D classification.

EMDB-41227: 
Cryo-electron tomography reconstruction of 80S ribosome in Translocation-intermediate-POST-3 state class 5, obtained from the EMPIAR-10987 dataset by constrained 3D classification.

EMDB-41196: 
A 3.2 Angstrom structure of HIV-1 CA-SP1 from 5 tomograms in EMPIAR-10164 dataset by single-particle cryo-electron tomography

EMDB-41197: 
A 3 Angstrom structure of HIV-1 CA-SP1 from EMPIAR-10164 dataset by single-particle cryo-electron tomography

EMDB-41199: 
1.8 Angstrom mouse heavy chain apoferritin from EMPIAR-11273 dataset by single-particle cryo-electron tomography

EMDB-28650: 
Human S1P transporter Spns2 in an inward-facing open conformation (state 1)

EMDB-28651: 
Human S1P transporter Spns2 in an outward-facing open conformation (state 4)

EMDB-28652: 
Human S1P transporter Spns2 in an inward-facing open conformation (state 1*)

EMDB-28653: 
Human S1P transporter Spns2 in an outward-facing partially occluded conformation (state 3)

EMDB-28654: 
Human S1P transporter Spns2 in an outward-facing partially occluded conformation (state 2)

PDB-8ex4: 
Human S1P transporter Spns2 in an inward-facing open conformation (state 1)

PDB-8ex5: 
Human S1P transporter Spns2 in an outward-facing open conformation (state 4)

PDB-8ex6: 
Human S1P transporter Spns2 in an inward-facing open conformation (state 1*)

PDB-8ex7: 
Human S1P transporter Spns2 in an outward-facing partially occluded conformation (state 3)

PDB-8ex8: 
Human S1P transporter Spns2 in an outward-facing partially occluded conformation (state 2)

EMDB-29860: 
Structure of inhibitor 16d-bound SPNS2

PDB-8g92: 
Structure of inhibitor 16d-bound SPNS2
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
