-検索条件
-検索結果
検索 (著者・登録者: hanna & m)の結果1,102件中、1から50件目までを表示しています

EMDB-50358: 
In vitro-induced genome-releasing intermediate of Rhodobacter microvirus Ebor computed with C5 symmetry
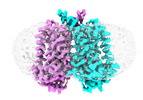
EMDB-43712: 
Human EBP complexed with compound 1
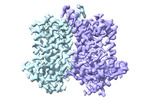
EMDB-43713: 
Human EBP complexed with compound 3a

PDB-8w0r: 
Human EBP complexed with compound 1

PDB-8w0s: 
Human EBP complexed with compound 3a

EMDB-50356: 
Empty capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-50357: 
Native capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-50359: 
Rhodobacter microvirus Ebor attached to B10 host cell reconstructed by single particle analysis with applied C5 symmetry

EMDB-50360: 
Rhodobacter microvirus Ebor attached to the outer membrane vesicle

EMDB-50361: 
Rhodobacter microvirus Ebor attached to the host cell reconstructed by subtomogram averaging

PDB-9ffg: 
Empty capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

PDB-9ffh: 
Native capsid of Rhodobacter microvirus Ebor computed with I4 symmetry

EMDB-41271: 
Consensus map of 96nm repeat of human respiratory doublet microtubule, RS1-2 region

EMDB-40815: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies C3V5, V1V3, N611 and base from participant 017

EMDB-40816: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp41-N611/FP and base from participant 03

EMDB-40817: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp41-N611/FP and base from participant 07

EMDB-40818: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp120-GH and base from participant 09

EMDB-40819: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies C3V5, V1V3, gp41-GH/FP and base from participant 11
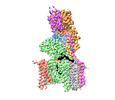
EMDB-17779: 
Structure of human oligosaccharyltransferase OST-A complex bound to NGI-1

PDB-8pn9: 
Structure of human oligosaccharyltransferase OST-A complex bound to NGI-1

EMDB-44416: 
Binned tomogram of stationary phase Burkholderia thailandensis

EMDB-19767: 
Structure of a 2873 Scaffold Base DNA Origami V1

EMDB-19769: 
Structure of a 2873 Scaffold Base DNA Origami V2

EMDB-19770: 
Structure of a 2873 Scaffold Base DNA Origami V3

EMDB-19775: 
Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 with Desalted Purified Staples

EMDB-19776: 
Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 with HPLC Purified Staples

EMDB-19867: 
Cryo-EM Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 and TBA

EMDB-19874: 
Refinement Focused on the 1st Body of a 1033 Scaffold-Based DNA Origami Nanostructure V4 with TBA

EMDB-19875: 
Refinement Focused on the 2nd Body of a 1033 Scaffold-Based DNA Origami Nanostructure V4 with TBA

EMDB-19876: 
Refinement Focused on the 3rd Body of a 1033 Scaffold-Based DNA Origami Nanostructure V4 with TBA

PDB-9eoq: 
Cryo-EM Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 and TBA

EMDB-41963: 
Preholo-Proteasome from Beta 3 D205 deletion

EMDB-41993: 
Proteasome 20S Core Particle from Beta 3 D205 deletion

PDB-8u6y: 
Preholo-Proteasome from Beta 3 D205 deletion

PDB-8u7u: 
Proteasome 20S Core Particle from Beta 3 D205 deletion

EMDB-19496: 
Structure of the formin Cdc12 bound to the barbed end of phalloidin-stabilized F-actin.

EMDB-19497: 
Cryo-EM reconstruction of the formin Cdc12 bound to the barbed end of F-actin (without phalloidin)

EMDB-19499: 
Structure of the F-actin barbed end bound by Cdc12 and profilin (ring complex) at a resolution of 6.3 Angstrom

EMDB-19501: 
Structure of the undecorated barbed end of F-actin.

EMDB-19503: 
Structure of the F-actin barbed end bound by formin mDia1

EMDB-19522: 
Structure of the formin INF2 bound to the barbed end of F-actin.

PDB-8rtt: 
Structure of the formin Cdc12 bound to the barbed end of phalloidin-stabilized F-actin.

PDB-8rty: 
Structure of the F-actin barbed end bound by Cdc12 and profilin (ring complex) at a resolution of 6.3 Angstrom

PDB-8ru0: 
Structure of the undecorated barbed end of F-actin.

PDB-8ru2: 
Structure of the F-actin barbed end bound by formin mDia1

PDB-8rv2: 
Structure of the formin INF2 bound to the barbed end of F-actin.
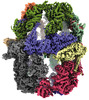
EMDB-19477: 
Saccharomyces cerevisiae FAS type I

EMDB-19489: 
Tobacco mosaic virus from scanning transmission electron microscopy at CSA=2.0 mrad

EMDB-41907: 
Computationally Designed, Expandable O4 Octahedral Handshake Nanocage
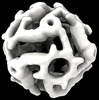
EMDB-42031: 
Computational Designed Nanocage O43_129_+8
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
