-検索条件
-検索結果
検索 (著者・登録者: frost & a)の結果163件中、1から50件目までを表示しています

EMDB-40940: 
TRPV1 in Nanodisc bound with lysophosphatidic acid in all four monomers

EMDB-40941: 
TRPV1 in Nanodisc not bound with lysophosphatidic acid (apo)

EMDB-40949: 
TRPV1 in nanodisc bound with one LPA in one monomer

EMDB-40951: 
TRPV1 in nanodisc bound with two LPA molecules in opposite monomers

EMDB-41005: 
TRPV1 in nanodisc bound with 2 LPA molecules in neighboring monomers

EMDB-41006: 
TRPV1 in nanodisc bound with 3 LPA molecules
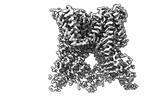
EMDB-41847: 
TRPV1 in nanodisc bound with diC8-PIP2 in the dilated state
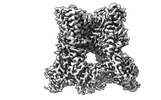
EMDB-41848: 
TRPV1 in nanodisc bound with diC8-PIP2 in the closed state

EMDB-41855: 
TRPV1 in nanodisc bound with PI-Br4 bound in Conformation 1 (monomer)
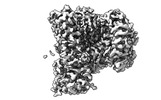
EMDB-41857: 
TRPV1 in nanodisc bound with PI-Br4 bound in Conformation 2 (monomer)

EMDB-41864: 
TRPV1 in nanodisc bound with empty vanilloid binding pocket at 4C

EMDB-41866: 
TRPV1 in nanodisc bound with empty vanilloid binding pocket at 25C

EMDB-41873: 
TRPV1 in nanodisc bound with PIP2-Br4

EMDB-41879: 
TRPV1 in nanodisc bound with PI-Br4, consensus structure

PDB-8t0c: 
TRPV1 in Nanodisc bound with lysophosphatidic acid in all four monomers

PDB-8t0e: 
TRPV1 in Nanodisc not bound with lysophosphatidic acid (apo)

PDB-8t0y: 
TRPV1 in nanodisc bound with one LPA in one monomer

PDB-8t10: 
TRPV1 in nanodisc bound with two LPA molecules in opposite monomers

PDB-8t3l: 
TRPV1 in nanodisc bound with 2 LPA molecules in neighboring monomers

PDB-8t3m: 
TRPV1 in nanodisc bound with 3 LPA molecules

PDB-8u2z: 
TRPV1 in nanodisc bound with diC8-PIP2 in the dilated state

PDB-8u30: 
TRPV1 in nanodisc bound with diC8-PIP2 in the closed state

PDB-8u3a: 
TRPV1 in nanodisc bound with PI-Br4 bound in Conformation 1 (monomer)

PDB-8u3c: 
TRPV1 in nanodisc bound with PI-Br4 bound in Conformation 2 (monomer)

PDB-8u3j: 
TRPV1 in nanodisc bound with empty vanilloid binding pocket at 4C

PDB-8u3l: 
TRPV1 in nanodisc bound with empty vanilloid binding pocket at 25C

PDB-8u43: 
TRPV1 in nanodisc bound with PIP2-Br4

PDB-8u4d: 
TRPV1 in nanodisc bound with PI-Br4, consensus structure

EMDB-18657: 
PROTAC-mediated complex of KRAS with VHL/Elongin-B/Elongin-C/Cullin-2/Rbx1
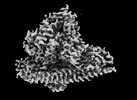
EMDB-41617: 
CryoEM structure of PI3Kalpha

PDB-8tu6: 
CryoEM structure of PI3Kalpha

EMDB-26977: 
CryoEM structure of human S-OPA1 assembled on lipid membrane in membrane-adjacent state

EMDB-26984: 
CryoEM structure of human S-OPA1 assembled on lipid membrane in membrane-distal state

PDB-8ct1: 
CryoEM structure of human S-OPA1 assembled on lipid membrane in membrane-adjacent state

PDB-8ct9: 
CryoEM structure of human S-OPA1 assembled on lipid membrane in membrane-distal state

EMDB-28540: 
BG505 UFO-E2p-L4P nanoparticle reconstructed by focused refinement with a mask around the nanoparticle core
EMDB-28541: 
BG505 UFO trimer map reconstructed from BG505 UFO-E2p-L4P nanoparticle by localized reconstruction

EMDB-28542: 
BG505 UFO-10GS-I3-01v9-L7P nanoparticle reconstructed by focused refinement with a mask around the nanoparticle core

EMDB-28543: 
BG505 UFO trimer map reconstructed from BG505 UFO-10GS-I3-01v9-L7P nanoparticle by localized reconstruction

PDB-8eqn: 
BG505 UFO-E2p-L4P nanoparticle reconstructed by focused refinement with a mask around the nanoparticle core

EMDB-25765: 
Cryo-EM structure of Human Enterovirus D68 US/MO/14-18947 strain native virion

EMDB-25772: 
Cryo-EM structure of Human Enterovirus D68 US/MO/14-18947 strain virion in complex with inhibitor 11526092

EMDB-25773: 
Cryo-EM structure of Human Enterovirus D68 US/MO/14-18947 strain virion in complex with pleconaril

EMDB-25774: 
Cryo-EM structure of Human Enterovirus D68 US/MO/14-18947 strain in complex with inhibitor 11526091 (no/low occupancy-no inhibitor modeled)

EMDB-25776: 
Cryo-EM structure of Human Enterovirus D68 US/MO/14-18947 strain in complex with inhibitor 11526093 (no/low occupancy-no inhibitor modeled)

EMDB-27991: 
CryoEM reconstruction of membrane-bound, left-handed CHMP1B+IST1 filament with brominated SDPC probe

EMDB-28694: 
CryoEM reconstruction of membrane-bound, right-handed CHMP1B+IST1 with brominated SDPC probe

EMDB-28695: 
CryoEM reconstruction of membrane-bound, left-handed CHMP1B+IST1 filament with brominated POPS probe

EMDB-28696: 
CryoEM reconstruction of membrane-bound, right-handed CHMP1B+IST1 filament with brominated POPS probe

EMDB-28697: 
CryoEM reconstruction of membrane-bound, left-handed CHMP1B+IST1 filament with brominated PIP2 probe
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
