-検索条件
-検索結果
検索 (著者・登録者: finn & r)の結果77件中、1から50件目までを表示しています

EMDB-44737: 
WT Pseudomonas phage PP7 capped tube VLP

EMDB-44743: 
WT Pseudomonas phage PP7 open tube VLP

EMDB-44767: 
C2 cage of PP7-AY-PP7: Pseudomonas phage PP7 coat protein dimer

EMDB-44768: 
C3 cage of PP7-AY-PP7: Pseudomonas phage PP7 coat protein dimer

EMDB-44773: 
D5 cage of PP7-AY-PP7: Pseudomonas phage PP7 coat protein dimer

EMDB-44782: 
D5E cage of PP7-AY-PP7: Pseudomonas phage PP7 coat protein dimer

EMDB-44783: 
Icosahedral T=3 cage of PP7-AY-PP7: Pseudomonas phage PP7 coat protein dimer

EMDB-44788: 
Icosahedral T=4 cage of PP7-AY-PP7: Pseudomonas phage PP7 coat protein dimer

EMDB-41816: 
Cryo-EM structure of the RAF1-HSP90-CDC37 complex in the closed state

EMDB-41817: 
Cryo-EM structure of the HSP90 dimer (NTD-MD) in the semi-open state

EMDB-41818: 
Cryo-EM structure of the cross-linked HSP90 dimer (NTD-MD) in the semi-open state

PDB-8u1l: 
Cryo-EM structure of the RAF1-HSP90-CDC37 complex in the closed state

PDB-8u1m: 
Cryo-EM structure of the HSP90 dimer (NTD-MD) in the semi-open state

PDB-8u1n: 
Cryo-EM structure of the cross-linked HSP90 dimer (NTD-MD) in the semi-open state

EMDB-18120: 
The closed state of the ASFV apo-RNA polymerase

EMDB-18129: 
The open state of the ASFV apo-RNA polymerase

EMDB-18163: 
consensus map of the ASFV RNA polymerase in open conformation

EMDB-18164: 
The stalk domain of the ASFV RNA polymerase obtained from Multibody refinement

PDB-8q3b: 
The closed state of the ASFV apo-RNA polymerase

PDB-8q3k: 
The open state of the ASFV apo-RNA polymerase

EMDB-43013: 
Myxococcus xanthus HEnc-K417N(A) protein shell with icosahedral T=1 symmetry

EMDB-43016: 
Myxococcus xanthus HEnc-K417N(A) protein shell with tetrahedral symmetry (12 pentamers, 4 hexamers)

EMDB-43037: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D3 symmetry (12 pentamers, 3 hexamers)

EMDB-43038: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D6 symmetry (12 pentamers, 8 hexamers)

EMDB-43039: 
Myxococcus xanthus HEnc-K417N(A) protein shell with C2 symmetry (12 pentamers, 9 hexamers)

EMDB-43040: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D3 symmetry (12 pentamers, 11 hexamers)

EMDB-43041: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D2 symmetry (12 pentamers, 12 hexamers)

EMDB-43042: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D2 symmetry (12 pentamers, 14 hexamers)

EMDB-43043: 
Myxococcus xanthus HEnc-K417N(A) protein shell with D5 symmetry (12 pentamers, 15 hexamers)

EMDB-43113: 
Myxococcus xanthus EncA WT protein shell with icosahedral symmetry T=3

EMDB-43036: 
Myxococcus xanthus HEnc-K417N(A) protein shell with icosahedral T=3 symmetry

EMDB-16512: 
MiniCoV-ADDomer, a SARS-CoV-2 epitope presenting viral like particle

EMDB-16522: 
Structure of ADDoCoV-ADAH11

PDB-8c9n: 
MiniCoV-ADDomer, a SARS-CoV-2 epitope presenting viral like particle

EMDB-42149: 
S1V2-72 Fab bound to EHA2 from influenza B/Malaysia/2506/2004

PDB-8udg: 
S1V2-72 Fab bound to EHA2 from influenza B/Malaysia/2506/2004

EMDB-27203: 
IMM20190 Fab complex with SARS-CoV-2 Spike Trimer

EMDB-27192: 
IMM20253 Fab complex with Trimer Spike protein of SARS-CoV-2 virus
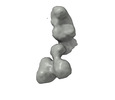
EMDB-27193: 
IMM20253 Fab complex with Spike monomer
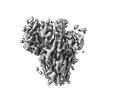
EMDB-27204: 
IMM20184 Fab complex with SARS-CoV-2 Spike Trimer

EMDB-27081: 
IMM20184 and IMM20253 Fabs in ternary complex with SARS-CoV-2 receptor binding domain

EMDB-25366: 
Chlorella virus hyaluronan synthase

EMDB-25367: 
Chlorella virus hyaluronan synthase inhibited by UDP

EMDB-25368: 
Chlorella virus Hyaluronan Synthase bound to UDP-GlcNAc

EMDB-25369: 
Chlorella virus Hyaluronan Synthase in the GlcNAc-primed channel-closed state

EMDB-25370: 
Chlorella virus Hyaluronan Synthase in the GlcNAc-primed, channel-open state

PDB-7sp6: 
Chlorella virus hyaluronan synthase

PDB-7sp7: 
Chlorella virus hyaluronan synthase inhibited by UDP

PDB-7sp8: 
Chlorella virus Hyaluronan Synthase bound to UDP-GlcNAc

PDB-7sp9: 
Chlorella virus Hyaluronan Synthase in the GlcNAc-primed channel-closed state
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
