-検索条件
-検索結果
検索 (著者・登録者: chand & d)の結果434件中、1から50件目までを表示しています

EMDB-41040: 
Human HCN1 F186C S264C C309A bound to cAMP, reconstituted in LMNG + SPL

PDB-8t4y: 
Human HCN1 F186C S264C C309A bound to cAMP, reconstituted in LMNG + SPL

EMDB-41036: 
Closed human HCN1 F186C S264C bound to cAMP, reconstituted in LMNG + SPL

PDB-8t4m: 
Closed human HCN1 F186C S264C bound to cAMP, reconstituted in LMNG + SPL

EMDB-29950: 
SARS-Cov2 S protein structure in complex with neutralizing monoclonal antibody 002-S21B10

EMDB-29975: 
Overall map of SARS-Cov2 S protein structure in complex with neutralizing monoclonal antibody 002-S21B10

EMDB-40007: 
Local map of SARS-Cov2 S protein structure in complex with neutralizing monoclonal antibody 002-S21B10

EMDB-41711: 
Disulfide-stabilized HIV-1 CA hexamer in complex with PQBP1 Nt

PDB-8ty6: 
Disulfide-stabilized HIV-1 CA hexamer in complex with PQBP1 Nt
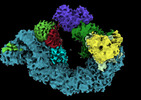
EMDB-19567: 
Closed crosslinked structure of (NEDD8)-CRL2VHL-MZ1-Brd4BD2-Ub(G76S, K48C)-UBE2R1(C93K, S138C, C191S, C223S)-Ub

EMDB-40967: 
Cryo-EM structure of a full-length, native Drp1 dimer

PDB-8t1h: 
Cryo-EM structure of a full-length, native Drp1 dimer

EMDB-19569: 
Open non-crosslinked structure Brd4BD2-MZ1-(NEDD8)-CRL2VHL

EMDB-40699: 
SARS-CoV-2 replication-transcription complex bound to nsp9 and UMPCPP, as a pre-catalytic NMPylation intermediate

EMDB-40707: 
SARS-CoV-2 replication-transcription complex bound to RNA-nsp9, as a noncatalytic RNA-nsp9 binding mode

EMDB-40708: 
SARS-CoV-2 replication-transcription complex bound to RNA-nsp9 and GDP-betaS, as a pre-catalytic deRNAylation/mRNA capping intermediate

PDB-8sq9: 
SARS-CoV-2 replication-transcription complex bound to nsp9 and UMPCPP, as a pre-catalytic NMPylation intermediate

PDB-8sqj: 
SARS-CoV-2 replication-transcription complex bound to RNA-nsp9, as a noncatalytic RNA-nsp9 binding mode

PDB-8sqk: 
SARS-CoV-2 replication-transcription complex bound to RNA-nsp9 and GDP-betaS, as a pre-catalytic deRNAylation/mRNA capping intermediate

EMDB-41075: 
SARS-CoV-2 spike in complex with Fab 71281-33

EMDB-41076: 
SARS-CoV-2 spike in complex with Fab 71281-33 (2)

EMDB-41153: 
Integrin alpha-v beta-8 in complex with minibinder B8_BP_dsulf

EMDB-41154: 
Integrin alpha-v beta-6 in complex with minibinder B6_BP_dslf

PDB-8tcf: 
Integrin alpha-v beta-8 in complex with minibinder B8_BP_dsulf

PDB-8tcg: 
Integrin alpha-v beta-6 in complex with minibinder B6_BP_dslf

EMDB-16052: 
Cryo-EM structure of stalled rabbit 80S ribosomes in complex with human CCR4-NOT and CNOT4

PDB-8bhf: 
Cryo-EM structure of stalled rabbit 80S ribosomes in complex with human CCR4-NOT and CNOT4

EMDB-29077: 
Cryo-EM structure of the STAR-0215 Fab in complex with active human plasma kallikrein

PDB-8fgx: 
Cryo-EM structure of the STAR-0215 Fab in complex with active human plasma kallikrein

EMDB-27808: 
RSV F trimer bound to RSV-199 Fab

EMDB-27846: 
HMPV F monomer bound to RSV-199 Fab

EMDB-27990: 
HMPV F complex with 4I3 Fab

EMDB-27995: 
HMPV F dimer bound to RSV-199 Fab

PDB-8dzw: 
RSV F trimer bound to RSV-199 Fab

PDB-8e2u: 
HMPV F monomer bound to RSV-199 Fab

PDB-8eay: 
HMPV F complex with 4I3 Fab

PDB-8ebp: 
HMPV F dimer bound to RSV-199 Fab

EMDB-34551: 
Cryo-EM structure of HACE1 monomer

EMDB-34586: 
Cryo-EM structure of HACE1 dimer

PDB-8h8x: 
Cryo-EM structure of HACE1 monomer

PDB-8hae: 
Cryo-EM structure of HACE1 dimer

EMDB-29857: 
Molecular mechanism of nucleotide inhibition of human uncoupling protein 1

PDB-8g8w: 
Molecular mechanism of nucleotide inhibition of human uncoupling protein 1

EMDB-16799: 
Cryo-EM structure of the NINJ1 filament

PDB-8cqr: 
Cryo-EM structure of the NINJ1 filament
EMDB-16914: 
Cryo-EM structure of the DnaD-NTD tetramer

PDB-8ojj: 
Cryo-EM structure of the DnaD-NTD tetramer

PDB-8sah: 
Huntingtin C-HEAT domain in complex with HAP40

EMDB-29790: 
30S focus refined map of WT E.coli ribosome complexed with A-site ortho-aminobenzoic acid charged tRNA-Phe

EMDB-26656: 
SARS-Cov2 S protein structure in complex with neutralizing monoclonal antibody 034_32
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
