-検索条件
-検索結果
検索 (著者・登録者: shi & j)の結果6,392件中、1から50件目までを表示しています

EMDB-19576: 
CRYO-EM STRUCTURE OF LEISHMANIA MAJOR 80S RIBOSOME WITH A/P/E-site tRNA AND mRNA : PARENTAL STRAIN

EMDB-19582: 
CRYO-EM STRUCTURE OF LEISHMANIA MAJOR 80S RIBOSOME WITH A/P/E-site tRNA AND mRNA : LM32Cs3H1 sKO STRAIN

PDB-8rxh: 
CRYO-EM STRUCTURE OF LEISHMANIA MAJOR 80S RIBOSOME WITH A/P/E-site tRNA AND mRNA : PARENTAL STRAIN

PDB-8rxx: 
CRYO-EM STRUCTURE OF LEISHMANIA MAJOR 80S RIBOSOME WITH A/P/E-site tRNA AND mRNA : LM32Cs3H1 sKO STRAIN

EMDB-43753: 
Yeast U1 snRNP with humanized U1C Zinc-Finger domain

PDB-8w2o: 
Yeast U1 snRNP with humanized U1C Zinc-Finger domain

EMDB-38215: 
Human GPR34 -Gi complex bound to S3E-LysoPS

EMDB-38217: 
Human GPR34 -Gi complex bound to S3E-LysoPS, receptor focused

PDB-8xbe: 
Human GPR34 -Gi complex bound to S3E-LysoPS

PDB-8xbg: 
Human GPR34 -Gi complex bound to S3E-LysoPS, receptor focused

EMDB-17216: 
CRYO-EM STRUCTURE OF LEISHMANIA MAJOR 80S RIBOSOME : PARENTAL STRAIN

PDB-8ovj: 
CRYO-EM STRUCTURE OF LEISHMANIA MAJOR 80S RIBOSOME : PARENTAL STRAIN
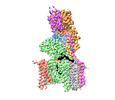
EMDB-17779: 
Structure of human oligosaccharyltransferase OST-A complex bound to NGI-1

PDB-8pn9: 
Structure of human oligosaccharyltransferase OST-A complex bound to NGI-1

EMDB-41639: 
Langya henipavirus fusion protein in postfusion state

EMDB-41640: 
Langya henipavirus fusion protein in prefusion state

EMDB-41641: 
Langya henipavirus postfusion F protein in complex with the 4G5 Fab, local refinement of the viral membrane distal region

EMDB-41642: 
Langya henipavirus postfusion F protein in complex with 4G5 Fab, local refinement of the viral membrane proximal region

EMDB-41644: 
Langya henipavirus postfusion fusion protein in complex with 4G5 Fab (global refinement)

PDB-8tve: 
Langya henipavirus fusion protein in postfusion state

PDB-8tvf: 
Langya henipavirus fusion protein in prefusion state

PDB-8tvg: 
Langya henipavirus postfusion F protein in complex with the 4G5 Fab, local refinement of the viral membrane distal region

PDB-8tvh: 
Langya henipavirus postfusion F protein in complex with 4G5 Fab, local refinement of the viral membrane proximal region

EMDB-34500: 
Cryo-EM structure of human exon-defined spliceosome in the late pre-B state.

EMDB-34505: 
Cryo-EM structure of human exon-defined spliceosome in the mature pre-B state.

EMDB-34507: 
Cryo-EM structure of human exon-defined spliceosome in the mature B state.

EMDB-34508: 
Cryo-EM structure of human exon-defined spliceosome in the early B state.

PDB-8h6e: 
Cryo-EM structure of human exon-defined spliceosome in the late pre-B state.

PDB-8h6j: 
Cryo-EM structure of human exon-defined spliceosome in the mature pre-B state.

PDB-8h6k: 
Cryo-EM structure of human exon-defined spliceosome in the mature B state.

PDB-8h6l: 
Cryo-EM structure of human exon-defined spliceosome in the early B state.

EMDB-37963: 
Cryo-EM structure of the hamster prion 23-144 fibril at pH 3.7

PDB-8wzx: 
Cryo-EM structure of the hamster prion 23-144 fibril at pH 3.7

EMDB-41636: 
Ghanaian virus fusion glycoprotein (GhV F)

EMDB-41643: 
Langya Virus G glycoprotein (LayV G) with stabilizing mutations

EMDB-43593: 
Langya Virus attachment (G) glycoprotein with K85L/L86K mutation

PDB-8tvb: 
Ghanaian virus fusion glycoprotein (GhV F)

PDB-8tvi: 
Langya Virus G glycoprotein (LayV G) with stabilizing mutations

PDB-8vwp: 
Langya Virus attachment (G) glycoprotein with K85L/L86K mutation

EMDB-36594: 
Cryo-EM structure of a designed AAV8-based vector

PDB-8jre: 
Cryo-EM structure of a designed AAV8-based vector

EMDB-37985: 
Cryo-EM structure of adenosine receptor A3AR bound to CF101

EMDB-37986: 
Cryo-EM structure of adenosine receptor A3AR bound to CF102

PDB-8x16: 
Cryo-EM structure of adenosine receptor A3AR bound to CF101

PDB-8x17: 
Cryo-EM structure of adenosine receptor A3AR bound to CF102

EMDB-18214: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex - hexameric assembly
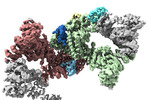
EMDB-18216: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused cullin dimer
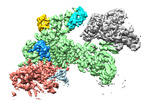
EMDB-18217: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused on E2-like density

EMDB-18218: 
Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused dimeric core
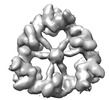
EMDB-18220: 
Structure of the hexameric CUL9-RBX1 complex with deletion of CUL9 CPH domain
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
