-検索条件
-検索結果
検索 (著者・登録者: miyazaki & n)の結果142件中、1から50件目までを表示しています

EMDB-16440: 
C1 reconstruction of Tanay virus particle

EMDB-32974: 
Capsid structure of Staphylococcus jumbo bacteriophage S6

PDB-7x30: 
Capsid structure of Staphylococcus jumbo bacteriophage S6

EMDB-35535: 
Human ornithine transcarbamylase expressed using mRNA lipid nanoparticles in human cells and PEGylated
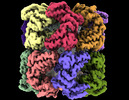
EMDB-33349: 
Cryo-EM structure of GroEL bound to unfolded substrate (UGT1A) at 2.8 Ang. resolution (Consensus Refinement)
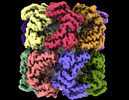
EMDB-33350: 
Cryo-EM structure of double occupied ring (DOR) of GroEL-UGT1A complex at 2.7 Ang. resolution
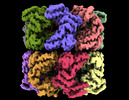
EMDB-33351: 
Cryo-EM structure of single empty ring 2 (SER2) of GroEL-UGT1A complex at 3.2 Ang. resolution
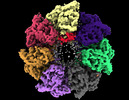
EMDB-33352: 
Cryo-EM structure of occupied ring subunit 4 (OR4) of GroEL complexed with polyalanine model of UGT1A from GroEL-UGT1A double occupied ring complex

EMDB-33353: 
Cryo-EM structure of empty ring subunit 1 (ER1) from single empty ring of GroEL-UGT1A complex

EMDB-33354: 
Cryo-EM structure of empty ring subunit 2 (ER2) from GroEL-UGT1A single empty ring complex
EMDB-33355: 
Cryo-EM structure of occupied ring subunit 1 (OR1) of GroEL from GroEL-UGT1A double occupied ring complex
EMDB-33356: 
Cryo-EM structure of occupied ring subunit 2 (OR2) of GroEL from GroEL-UGT1A double occupied ring complex
EMDB-33357: 
Cryo-EM structure of occupied ring subunit 3 (OR3) of GroEL from GroEL-UGT1A double occupied ring complex
EMDB-33358: 
Cryo-EM structure of occupied ring subunit 4 (OR4) of GroEL from GroEL-UGT1A double occupied ring complex
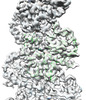
EMDB-33007: 
A CBg-ParM filament with ADP
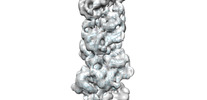
EMDB-33008: 
A CBg-ParM filament with GTP and a short incubation time
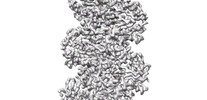
EMDB-33009: 
A CBg-ParM filament with ADP
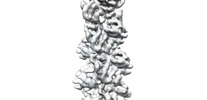
EMDB-33012: 
A CBg-ParM filament with GTP or GDPPi

EMDB-32614: 
Overall structure of Helicobacter pylori bacteriophage KHP30

EMDB-32616: 
Tail structure of Helicobacter pylori bacteriophage KHP30

PDB-7wmp: 
Tail structure of Helicobacter pylori bacteriophage KHP30

EMDB-15855: 
Rosellinia necatrix megabirnavirus 1-W779 full capsid

EMDB-15857: 
Rosellinia necatrix megabirnavirus 1-W779 empty capsid

EMDB-15859: 
Rosellinia necatrix megabirnavirus 1-W779 full capsid with Crown protein

PDB-8b4z: 
Rosellinia necatrix megabirnavirus 1-W779 full capsid

PDB-8b59: 
Rosellinia necatrix megabirnavirus 1-W779 Crown protein
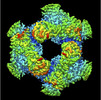
EMDB-32571: 
Cryo-EM structure of GH31 alpha-1,3-glucosidase from Lactococcus lactis subsp. cremoris

PDB-7wlg: 
Cryo-EM structure of GH31 alpha-1,3-glucosidase from Lactococcus lactis subsp. cremoris

EMDB-14174: 
TMEM106B filaments with Fold I from Alzheimer's disease (case 1)

EMDB-14176: 
TMEM106B filaments with Fold I-d from Multiple system atrophy (case 18)

EMDB-14187: 
TMEM106B filaments with Fold IIa from Multiple system atrophy (case 19)

EMDB-14188: 
TMEM106B filaments with Fold IIb from Multiple system atrophy (case 19)

EMDB-14189: 
TMEM106B filaments with Fold III from Multiple system atrophy (case 17)
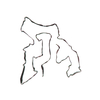
PDB-7qvc: 
TMEM106B filaments with Fold I from Alzheimer's disease (case 1)
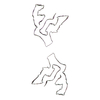
PDB-7qvf: 
TMEM106B filaments with Fold I-d from Multiple system atrophy (case 18)
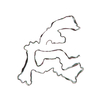
PDB-7qwg: 
TMEM106B filaments with Fold IIa from Multiple system atrophy (case 19)
PDB-7qwl: 
TMEM106B filaments with Fold IIb from Multiple system atrophy (case 19)

PDB-7qwm: 
TMEM106B filaments with Fold III from Multiple system atrophy (case 17)

EMDB-31905: 
Structure of C2S2M2-type PSII-FCPII supercomplex from diatom

EMDB-31906: 
Structure of S1M1-type FCPII complex from diatom

PDB-7vd5: 
Structure of C2S2M2-type PSII-FCPII supercomplex from diatom

PDB-7vd6: 
Structure of S1M1-type FCPII complex from diatom

EMDB-30820: 
Structure of Wild-type PSI monomer1 from Cyanophora paradoxa

EMDB-30821: 
Structure of Wild-type PSI monomer2 from Cyanophora paradoxa

EMDB-30822: 
Structure of Wild-type PSI tetramer from Cyanophora paradoxa

EMDB-30823: 
Structure of GraFix PSI tetramer from Cyanophora paradoxa

PDB-7dr0: 
Structure of Wild-type PSI monomer1 from Cyanophora paradoxa

PDB-7dr1: 
Structure of Wild-type PSI monomer2 from Cyanophora paradoxa

PDB-7dr2: 
Structure of GraFix PSI tetramer from Cyanophora paradoxa

EMDB-30793: 
Capsid structure of human sapovirus
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
