-検索条件
-検索結果
検索 (著者・登録者: mayer & a)の結果136件中、1から50件目までを表示しています

EMDB-43508: 
Structure of a bacterial gasdermin small oval pore assembly

EMDB-43509: 
Structure of a bacterial gasdermin medium oval pore assembly

EMDB-43510: 
Structure of a bacterial gasdermin large oval pore assembly

EMDB-43511: 
Structure of a bacterial gasdermin double pore assembly

EMDB-43513: 
Structure of a bacterial gasdermin slinky-like oligomer from a heterogeneous sample

EMDB-15411: 
Single particle structure of Atg18-WT

PDB-8afx: 
Single particle structure of Atg18-WT

EMDB-18657: 
PROTAC-mediated complex of KRAS with VHL/Elongin-B/Elongin-C/Cullin-2/Rbx1

EMDB-41983: 
Structure of the phage immune evasion protein Gad1 bound to the Gabija GajAB complex

PDB-8u7i: 
Structure of the phage immune evasion protein Gad1 bound to the Gabija GajAB complex

EMDB-15408: 
Tube assembly of Atg18-PR72AA

EMDB-15410: 
Tube assembly of Atg18-WT

EMDB-15412: 
Subtomogram average of membrane-bound Atg18 oligomers

PDB-8afq: 
Tube assembly of Atg18-PR72AA

PDB-8afw: 
Tube assembly of Atg18-WT

PDB-8afy: 
Subtomogram average of membrane-bound Atg18 oligomers

EMDB-17015: 
Cryo-EM map of the focused refinement of the subfamily III haloalkane dehalogenase from Haloferax mediterranei dimer forming hexameric assembly.

PDB-8ooh: 
Cryo-EM map of the focused refinement of the subfamily III haloalkane dehalogenase from Haloferax mediterranei dimer forming hexameric assembly.

EMDB-16998: 
Cryo-EM structure of subfamily III haloalkane dehalogenase DhmeA from Haloferax mediterranei

EMDB-27205: 
Closed state of SARS-CoV-2 BA.2 variant spike protein

EMDB-27206: 
One RBD-up state of SARS-CoV-2 BA.2 variant spike protein

EMDB-27207: 
Middle state of SARS-CoV-2 BA.2 variant spike protein

PDB-8d55: 
Closed state of SARS-CoV-2 BA.2 variant spike protein

PDB-8d56: 
One RBD-up state of SARS-CoV-2 BA.2 variant spike protein

PDB-8d5a: 
Middle state of SARS-CoV-2 BA.2 variant spike protein

EMDB-40570: 
Structure of a bacterial gasdermin slinky-like oligomer

PDB-8sl0: 
Structure of a bacterial gasdermin slinky-like oligomer

EMDB-29016: 
Cryo-EM structure of SARS-CoV-2 postfusion spike in membrane
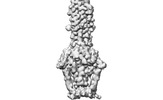
EMDB-29017: 
Cryo-EM structure of SARS-CoV-2 postfusion spike in membrane

EMDB-29018: 
Cryo-EM structure of SARS-CoV-2 postfusion spike in membrane

PDB-8fdw: 
Cryo-EM structure of SARS-CoV-2 postfusion spike in membrane

EMDB-25419: 
Previously uncharacterized rectangular bacteria in the dolphin mouth

EMDB-35208: 
Cryo-EM structure of the polyphosphate polymerase VTC complex(Vtc4/Vtc3/Vtc1)

PDB-8i6v: 
Cryo-EM structure of the polyphosphate polymerase VTC complex(Vtc4/Vtc3/Vtc1)

EMDB-29323: 
Structure of RdrA from Escherichia coli RADAR defense system

EMDB-29324: 
Map of RdrA from Escherichia coli RADAR defense system in single-split conformation

EMDB-29325: 
Map of RdrA from Escherichia coli RADAR defense system in double-split conformation

EMDB-29326: 
Structure of RdrA from Streptococcus suis RADAR defense system

EMDB-29327: 
Structure of RdrB from Escherichia coli RADAR defense system

EMDB-29328: 
Structure of RdrA-RdrB complex from Escherichia coli RADAR defense system

PDB-8fnt: 
Structure of RdrA from Escherichia coli RADAR defense system

PDB-8fnu: 
Structure of RdrA from Streptococcus suis RADAR defense system

PDB-8fnv: 
Structure of RdrB from Escherichia coli RADAR defense system

PDB-8fnw: 
Structure of RdrA-RdrB complex from Escherichia coli RADAR defense system

EMDB-27704: 
Cryo-EM structure of insulin receptor (IR) bound with S597 peptide

EMDB-27705: 
Cryo-EM structure of insulin receptor (IR) bound with S597 component 2

PDB-8dtl: 
Cryo-EM structure of insulin receptor (IR) bound with S597 peptide

PDB-8dtm: 
Cryo-EM structure of insulin receptor (IR) bound with S597 component 2

EMDB-31642: 
Local construction of SARS-CoV-2 S protein RBD in complex with XG014 Fab

EMDB-25188: 
Full-length insulin receptor bound with site 1 binding deficient mutant insulin (A-V3E)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
