-検索条件
-検索結果
検索 (著者・登録者: maeda & k)の結果82件中、1から50件目までを表示しています

EMDB-34741: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-11

EMDB-34742: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-11 focused on RBD and NIV-11 interface

PDB-8hgl: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-11

PDB-8hgm: 
Structure of SARS-CoV-2 spike RBD in complex with neutralizing antibody NIV-11

EMDB-33820: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-8 focused on RBD and NIV-8 interface

EMDB-33821: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-8 (state 1)

EMDB-33822: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-8 (state 2)

EMDB-33823: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-10 focused on RBD and NIV-10 interface

EMDB-33824: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-10 (state 1)

EMDB-33825: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-10 (state 2)

EMDB-33826: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-10 (state 3)

EMDB-33827: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-13 focused on RBD and NIV-13 interface

EMDB-33828: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-13 (state 1)

EMDB-33829: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-13 (state 2)

EMDB-33830: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-13 (state 3)

PDB-7yh6: 
Structure of SARS-CoV-2 spike RBD in complex with neutralizing antibody NIV-8

PDB-7yh7: 
SARS-CoV-2 spike in complex with neutralizing antibody NIV-8 (state 2)
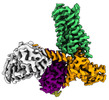
EMDB-25748: 
Cryo-EM structure of ACh-bound M2R-Go signaling complex in S1 state
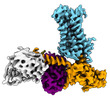
EMDB-25749: 
Cryo-EM structure of ACh-bound M2R-Go signaling complex in S2 state
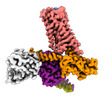
EMDB-25751: 
Cryo-EM structure of S1 state ACh-bound M2R-Go signaling complex with a PAM
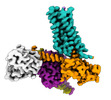
EMDB-25752: 
Cryo-EM structure of S2 state ACh-bound M2R-Go signaling complex with a PAM

PDB-7t8x: 
Cryo-EM structure of ACh-bound M2R-Go signaling complex in S1 state

PDB-7t90: 
Cryo-EM structure of ACh-bound M2R-Go signaling complex in S2 state

PDB-7t94: 
Cryo-EM structure of S1 state ACh-bound M2R-Go signaling complex with a PAM

PDB-7t96: 
Cryo-EM structure of S2 state ACh-bound M2R-Go signaling complex with a PAM

EMDB-25706: 
Cryo-EM structure of human SIMC1-SLF2 complex

PDB-7t5p: 
Cryo-EM structure of human SIMC1-SLF2 complex

EMDB-25446: 
Cryo-EM structure of Arabidopsis Ago10-guide RNA complex

EMDB-25482: 
Cryo-EM structure of Arabidopsis Ago10-guide-target RNA complex in a bent duplex conformation

PDB-7sva: 
Cryo-EM structure of Arabidopsis Ago10-guide RNA complex

PDB-7swq: 
Cryo-EM structure of Arabidopsis Ago10-guide-target RNA complex in a bent duplex conformation
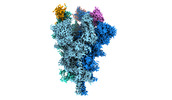
EMDB-32078: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobodies P86
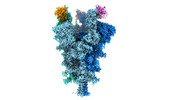
EMDB-32079: 
Cryo-EM structure of the SARS-CoV-2 spike protein (3-up RBD) bound to neutralizing nanobodies P86
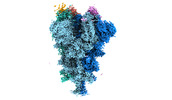
EMDB-32080: 
Cryo-EM structure of the SARS-CoV-2 spike protein (1-up RBD) bound to neutralizing nanobodies P17
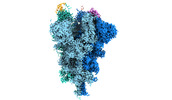
EMDB-32081: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobodies P17

PDB-7vq0: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobodies P86

EMDB-24496: 
Structure of CX3CL1-US28-G11iN18-scFv16 in TL-state

EMDB-24500: 
Structure of CX3CL1-US28-Gi-scFv16 in C-state

EMDB-24501: 
Structure of CX3CL1-US28-Gi-scFv16 in OC-state

EMDB-24506: 
Structure of US27-Gi-scFv16 in CL-state

EMDB-24507: 
Binding mode of US27-Gi-scFv16 in OCL-state

PDB-7rkf: 
Structure of CX3CL1-US28-G11iN18-scFv16 in TL-state

EMDB-22338: 
Structure of the Epstein-Barr virus GPCR BILF1 in complex with human Gi

PDB-7jhj: 
Structure of the Epstein-Barr virus GPCR BILF1 in complex with human Gi

EMDB-30420: 
Structure of the far-red light utilizing photosystem I of Acaryochloris marina

PDB-7coy: 
Structure of the far-red light utilizing photosystem I of Acaryochloris marina

EMDB-22375: 
Cryo-EM structure of human ATG9A in amphipols

EMDB-22376: 
cryo-EM structure of human ATG9A in nanodiscs

EMDB-22377: 
cryo-EM structure of human ATG9A in LMNG micelles

PDB-7jlo: 
Cryo-EM structure of human ATG9A in amphipols
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
