-検索条件
-検索結果
検索 (著者・登録者: gabriel & c & lander)の結果186件中、1から50件目までを表示しています

EMDB-41788: 
S. cerevisiae Pex1/Pex6 with 1 mM ATP

PDB-8u0v: 
S. cerevisiae Pex1/Pex6 with 1 mM ATP

EMDB-29280: 
Cas1-Cas2/3 integrase and IHF bound to CRISPR leader, repeat and foreign DNA

PDB-8flj: 
Cas1-Cas2/3 integrase and IHF bound to CRISPR leader, repeat and foreign DNA

EMDB-29070: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA(UGA-TL)

PDB-8ffy: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA(UGA-TL)

EMDB-27269: 
Cryo-EM structure of human DELE1 in oligomeric form

PDB-8d9x: 
Cryo-EM structure of human DELE1 in oligomeric form

EMDB-25730: 
Cryo-EM structure of full-length hepatitis C virus E1E2 glycoprotein in complex with AR4A, AT12009, and IGH505 Fabs

EMDB-27578: 
nsEM map of E1E2 AMS0232 glycoprotein in complex with monoclonal antibody AR4A

PDB-7t6x: 
Cryo-EM structure of full-length hepatitis C virus E1E2 glycoprotein in complex with AR4A, AT12009, and IGH505 Fabs
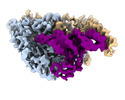
EMDB-26310: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA (GCU)
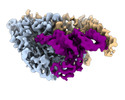
EMDB-26311: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA(GCU-TL)

PDB-7u2a: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA (GCU)

PDB-7u2b: 
Cryo-electron microscopy structure of human mt-SerRS in complex with mt-tRNA(GCU-TL)

EMDB-27012: 
Cereblon-DDB1 in the Apo form

EMDB-27234: 
Cereblon~DDB1 bound to CC-92480 with DDB1 in the linear conformation

EMDB-27235: 
Cereblon~DDB1 bound to CC-92480 with DDB1 in the twisted conformation

EMDB-27236: 
Cereblon~DDB1 bound to CC-92480 with DDB1 in the hinged conformation

EMDB-27237: 
Cereblon~DDB1 in the Apo form with DDB1 in the hinged conformation

EMDB-27238: 
Cereblon-DDB1 in the Apo form with DDB1 in the twisted conformation
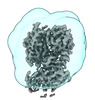
EMDB-27239: 
Cereblon bound to CC-92480, Focused Refinement

EMDB-27240: 
Cereblon-DDB1 bound to CC-92480 and Ikaros ZF1-2-3

EMDB-27241: 
Cereblon~DDB1 bound to Iberdomide and Ikaros ZF1-2-3

EMDB-27242: 
Cereblon~DDB1 bound to Pomalidomide

PDB-8cvp: 
Cereblon-DDB1 in the Apo form

PDB-8d7u: 
Cereblon~DDB1 bound to CC-92480 with DDB1 in the linear conformation

PDB-8d7v: 
Cereblon~DDB1 bound to CC-92480 with DDB1 in the twisted conformation

PDB-8d7w: 
Cereblon~DDB1 bound to CC-92480 with DDB1 in the hinged conformation

PDB-8d7x: 
Cereblon~DDB1 in the Apo form with DDB1 in the hinged conformation

PDB-8d7y: 
Cereblon-DDB1 in the Apo form with DDB1 in the twisted conformation

PDB-8d7z: 
Cereblon-DDB1 bound to CC-92480 and Ikaros ZF1-2-3

PDB-8d80: 
Cereblon~DDB1 bound to Iberdomide and Ikaros ZF1-2-3

PDB-8d81: 
Cereblon~DDB1 bound to Pomalidomide

EMDB-25502: 
Yeast Lon (PIM1) with endogenous substrate

PDB-7sxo: 
Yeast Lon (PIM1) with endogenous substrate

EMDB-24795: 
Mouse heavy chain apoferritin determined in presence of 1.66% glycerol.

EMDB-24796: 
Mouse heavy chain apoferritin determined in presence of 20% glycerol (200keV)

EMDB-24797: 
Mouse heavy chain apoferritin determined in presence of 20% glycerol (300keV)

EMDB-24798: 
Rabbit muscle aldolase determined in presence of 20% v/v glycerol

EMDB-24799: 
Rabbit muscle aldolase determined in the presence of 20% v/v glycerol.

EMDB-24850: 
Compressed conformation of nighttime state KaiC

EMDB-24851: 
Extended conformation of nighttime state KaiC

EMDB-24852: 
Extended conformation of daytime state KaiC

PDB-7s65: 
Compressed conformation of nighttime state KaiC

PDB-7s66: 
Extended conformation of nighttime state KaiC

PDB-7s67: 
Extended conformation of daytime state KaiC

EMDB-23061: 
Cryo-EM structure of Ephydatia fluviatilis PiwiA-piRNA complex

EMDB-23062: 
Cryo-EM structure of EfPiwiA(MID/PIWI domains)-piRNA-long-target complex

EMDB-23063: 
Cryo-EM structure of Ephydatia fluviatilis PiwiA-piRNA-target complex
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
