| 登録情報 | データベース: PDB / ID: 6in7
|
|---|
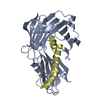
| タイトル | Crystal structure of AlgU in complex with MucA(cyto) |
|---|
 要素 要素 | - RNA polymerase sigma-H factor
- Sigma factor AlgU negative regulatory protein
|
|---|
 キーワード キーワード |  TRANSCRIPTION (転写 (生物学)) / TRANSCRIPTION (転写 (生物学)) /  Sigma factor (シグマ因子) Sigma factor (シグマ因子) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of cellular response to heat / regulation of polysaccharide biosynthetic process / positive regulation of response to oxidative stress / positive regulation of cell adhesion involved in single-species biofilm formation / positive regulation of single-species biofilm formation on inanimate substrate / negative regulation of bacterial-type flagellum-dependent cell motility / alginic acid biosynthetic process / sigma factor antagonist activity / cellular response to antibiotic / DNA-binding transcription activator activity ...positive regulation of cellular response to heat / regulation of polysaccharide biosynthetic process / positive regulation of response to oxidative stress / positive regulation of cell adhesion involved in single-species biofilm formation / positive regulation of single-species biofilm formation on inanimate substrate / negative regulation of bacterial-type flagellum-dependent cell motility / alginic acid biosynthetic process / sigma factor antagonist activity / cellular response to antibiotic / DNA-binding transcription activator activity / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex /  sigma factor activity / sigma factor activity /  デオキシリボ核酸 / DNA-templated transcription initiation / transcription cis-regulatory region binding / positive regulation of DNA-templated transcription / デオキシリボ核酸 / DNA-templated transcription initiation / transcription cis-regulatory region binding / positive regulation of DNA-templated transcription /  細胞膜類似検索 - 分子機能 細胞膜類似検索 - 分子機能 Anti sigma-E protein RseA, N-terminal / Anti sigma-E protein RseA, C-terminal / Anti sigma-E protein RseA, N-terminal domain superfamily / Anti sigma-E protein RseA, N-terminal domain / Anti sigma-E protein RseA, C-terminal domain / RNA polymerase sigma-70 RpoE type / RNA polymerase sigma factor 70, region 4 type 2 /  Sigma-70, region 4 / RNA polymerase sigma factor 70, ECF, conserved site / Sigma-70 factors ECF subfamily signature. ...Anti sigma-E protein RseA, N-terminal / Anti sigma-E protein RseA, C-terminal / Anti sigma-E protein RseA, N-terminal domain superfamily / Anti sigma-E protein RseA, N-terminal domain / Anti sigma-E protein RseA, C-terminal domain / RNA polymerase sigma-70 RpoE type / RNA polymerase sigma factor 70, region 4 type 2 / Sigma-70, region 4 / RNA polymerase sigma factor 70, ECF, conserved site / Sigma-70 factors ECF subfamily signature. ...Anti sigma-E protein RseA, N-terminal / Anti sigma-E protein RseA, C-terminal / Anti sigma-E protein RseA, N-terminal domain superfamily / Anti sigma-E protein RseA, N-terminal domain / Anti sigma-E protein RseA, C-terminal domain / RNA polymerase sigma-70 RpoE type / RNA polymerase sigma factor 70, region 4 type 2 /  Sigma-70, region 4 / RNA polymerase sigma factor 70, ECF, conserved site / Sigma-70 factors ECF subfamily signature. / RNA polymerase sigma-70 like / RNA polymerase sigma factor, region 2, helix turn helix motif / Rna Polymerase Sigma Factor; Chain: A / RNA polymerase sigma-70 region 2 / RNA polymerase sigma-70 like domain / Sigma-70 region 2 / RNA polymerase sigma factor, region 2 / RNA polymerase sigma factor, region 3/4-like / Winged helix-like DNA-binding domain superfamily/Winged helix DNA-binding domain / Arc Repressor Mutant, subunit A / Winged helix-like DNA-binding domain superfamily / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 Sigma-70, region 4 / RNA polymerase sigma factor 70, ECF, conserved site / Sigma-70 factors ECF subfamily signature. / RNA polymerase sigma-70 like / RNA polymerase sigma factor, region 2, helix turn helix motif / Rna Polymerase Sigma Factor; Chain: A / RNA polymerase sigma-70 region 2 / RNA polymerase sigma-70 like domain / Sigma-70 region 2 / RNA polymerase sigma factor, region 2 / RNA polymerase sigma factor, region 3/4-like / Winged helix-like DNA-binding domain superfamily/Winged helix DNA-binding domain / Arc Repressor Mutant, subunit A / Winged helix-like DNA-binding domain superfamily / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性  ニコチンアミド / Sigma factor AlgU negative regulatory protein / RNA polymerase sigma-H factor類似検索 - 構成要素 ニコチンアミド / Sigma factor AlgU negative regulatory protein / RNA polymerase sigma-H factor類似検索 - 構成要素 |
|---|
| 生物種 |    Pseudomonas aeruginosa (緑膿菌) Pseudomonas aeruginosa (緑膿菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.96 Å 分子置換 / 解像度: 1.96 Å |
|---|
 データ登録者 データ登録者 | Li, S. / Zhang, Q. / Bartlam, M. |
|---|
| 資金援助 |  中国, 1件 中国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Natural Science Foundation of China | 31570128 |  中国 中国 |
|
|---|
 引用 引用 |  ジャーナル: Febs J. / 年: 2019 ジャーナル: Febs J. / 年: 2019
タイトル: Structural basis for the recognition of MucA by MucB and AlgU in Pseudomonas aeruginosa.
著者: Li, S. / Lou, X. / Xu, Y. / Teng, X. / Liu, R. / Zhang, Q. / Wu, W. / Wang, Y. / Bartlam, M. |
|---|
| 履歴 | | 登録 | 2018年10月24日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2019年7月24日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年1月8日 | Group: Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.2 | 2023年11月22日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRANSCRIPTION (転写 (生物学)) /
TRANSCRIPTION (転写 (生物学)) /  Sigma factor (シグマ因子)
Sigma factor (シグマ因子) 機能・相同性情報
機能・相同性情報 sigma factor activity /
sigma factor activity /  デオキシリボ核酸 / DNA-templated transcription initiation / transcription cis-regulatory region binding / positive regulation of DNA-templated transcription /
デオキシリボ核酸 / DNA-templated transcription initiation / transcription cis-regulatory region binding / positive regulation of DNA-templated transcription /  細胞膜
細胞膜
 Pseudomonas aeruginosa (緑膿菌)
Pseudomonas aeruginosa (緑膿菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.96 Å
分子置換 / 解像度: 1.96 Å  データ登録者
データ登録者 中国, 1件
中国, 1件  引用
引用 ジャーナル: Febs J. / 年: 2019
ジャーナル: Febs J. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6in7.cif.gz
6in7.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6in7.ent.gz
pdb6in7.ent.gz PDB形式
PDB形式 6in7.json.gz
6in7.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/in/6in7
https://data.pdbj.org/pub/pdb/validation_reports/in/6in7 ftp://data.pdbj.org/pub/pdb/validation_reports/in/6in7
ftp://data.pdbj.org/pub/pdb/validation_reports/in/6in7 リンク
リンク 集合体
集合体
 要素
要素
 Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)
Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)
 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: P38107
Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: P38107
 Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)
Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)
 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q06198
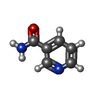
Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q06198 ニコチンアミド
ニコチンアミド 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL18U1 / 波長: 0.977853 Å
/ ビームライン: BL18U1 / 波長: 0.977853 Å : 0.977853 Å / 相対比: 1
: 0.977853 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj