+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6a93 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of 5-HT2AR in complex with risperidone | ||||||
 要素 要素 | 5-hydroxytryptamine receptor 2A,Soluble cytochrome b562 | ||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein localization to cytoskeleton / 1-(4-iodo-2,5-dimethoxyphenyl)propan-2-amine binding / Gq/11-coupled serotonin receptor activity / phospholipase C-activating serotonin receptor signaling pathway / positive regulation of phosphatidylinositol biosynthetic process / G protein-coupled serotonin receptor signaling pathway / G protein-coupled serotonin receptor complex / serotonin receptor signaling pathway /  セロトニン受容体 / artery smooth muscle contraction ...protein localization to cytoskeleton / 1-(4-iodo-2,5-dimethoxyphenyl)propan-2-amine binding / Gq/11-coupled serotonin receptor activity / phospholipase C-activating serotonin receptor signaling pathway / positive regulation of phosphatidylinositol biosynthetic process / G protein-coupled serotonin receptor signaling pathway / G protein-coupled serotonin receptor complex / serotonin receptor signaling pathway / セロトニン受容体 / artery smooth muscle contraction ...protein localization to cytoskeleton / 1-(4-iodo-2,5-dimethoxyphenyl)propan-2-amine binding / Gq/11-coupled serotonin receptor activity / phospholipase C-activating serotonin receptor signaling pathway / positive regulation of phosphatidylinositol biosynthetic process / G protein-coupled serotonin receptor signaling pathway / G protein-coupled serotonin receptor complex / serotonin receptor signaling pathway /  セロトニン受容体 / artery smooth muscle contraction / cell body fiber / urinary bladder smooth muscle contraction / セロトニン受容体 / artery smooth muscle contraction / cell body fiber / urinary bladder smooth muscle contraction /  serotonin binding / negative regulation of synaptic transmission, glutamatergic / G protein-coupled serotonin receptor activity / serotonin binding / negative regulation of synaptic transmission, glutamatergic / G protein-coupled serotonin receptor activity /  neurotransmitter receptor activity / neurotransmitter receptor activity /  temperature homeostasis / regulation of dopamine secretion / protein tyrosine kinase activator activity / behavioral response to cocaine / detection of temperature stimulus involved in sensory perception of pain / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / negative regulation of potassium ion transport / activation of phospholipase C activity / positive regulation of fat cell differentiation / detection of mechanical stimulus involved in sensory perception of pain / release of sequestered calcium ion into cytosol / positive regulation of vasoconstriction / presynaptic modulation of chemical synaptic transmission / positive regulation of glycolytic process / dendritic shaft / phosphatidylinositol 3-kinase/protein kinase B signal transduction / temperature homeostasis / regulation of dopamine secretion / protein tyrosine kinase activator activity / behavioral response to cocaine / detection of temperature stimulus involved in sensory perception of pain / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / negative regulation of potassium ion transport / activation of phospholipase C activity / positive regulation of fat cell differentiation / detection of mechanical stimulus involved in sensory perception of pain / release of sequestered calcium ion into cytosol / positive regulation of vasoconstriction / presynaptic modulation of chemical synaptic transmission / positive regulation of glycolytic process / dendritic shaft / phosphatidylinositol 3-kinase/protein kinase B signal transduction /  カベオラ / glycolytic process / intracellular calcium ion homeostasis / カベオラ / glycolytic process / intracellular calcium ion homeostasis /  記憶 / positive regulation of neuron apoptotic process / positive regulation of peptidyl-tyrosine phosphorylation / virus receptor activity / 記憶 / positive regulation of neuron apoptotic process / positive regulation of peptidyl-tyrosine phosphorylation / virus receptor activity /  presynaptic membrane / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of cytosolic calcium ion concentration / chemical synaptic transmission / cytoplasmic vesicle / presynaptic membrane / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of cytosolic calcium ion concentration / chemical synaptic transmission / cytoplasmic vesicle /  postsynaptic membrane / G alpha (q) signalling events / postsynaptic membrane / G alpha (q) signalling events /  electron transfer activity / electron transfer activity /  ペリプラズム / positive regulation of ERK1 and ERK2 cascade / response to xenobiotic stimulus / iron ion binding / ペリプラズム / positive regulation of ERK1 and ERK2 cascade / response to xenobiotic stimulus / iron ion binding /  神経繊維 / 神経繊維 /  樹状突起 / neuronal cell body / glutamatergic synapse / positive regulation of cell population proliferation / 樹状突起 / neuronal cell body / glutamatergic synapse / positive regulation of cell population proliferation /  heme binding / protein-containing complex binding / identical protein binding / heme binding / protein-containing complex binding / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)  Escherichia coli (大腸菌) Escherichia coli (大腸菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3 Å 分子置換 / 解像度: 3 Å | ||||||
 データ登録者 データ登録者 | Kimura, T.K. / Asada, H. / Inoue, A. / Kadji, F.M.N. / Im, D. / Mori, C. / Arakawa, T. / Hirata, K. / Nomura, Y. / Nomura, N. ...Kimura, T.K. / Asada, H. / Inoue, A. / Kadji, F.M.N. / Im, D. / Mori, C. / Arakawa, T. / Hirata, K. / Nomura, Y. / Nomura, N. / Aoki, J. / Iwata, S. / Shimamura, T. | ||||||
| 資金援助 |  日本, 1件 日本, 1件
| ||||||
 引用 引用 |  ジャーナル: Nat.Struct.Mol.Biol. / 年: 2019 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2019タイトル: Structures of the 5-HT2Areceptor in complex with the antipsychotics risperidone and zotepine. 著者: Kimura, K.T. / Asada, H. / Inoue, A. / Kadji, F.M.N. / Im, D. / Mori, C. / Arakawa, T. / Hirata, K. / Nomura, Y. / Nomura, N. / Aoki, J. / Iwata, S. / Shimamura, T. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6a93.cif.gz 6a93.cif.gz | 306 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6a93.ent.gz pdb6a93.ent.gz | 246.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6a93.json.gz 6a93.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/a9/6a93 https://data.pdbj.org/pub/pdb/validation_reports/a9/6a93 ftp://data.pdbj.org/pub/pdb/validation_reports/a9/6a93 ftp://data.pdbj.org/pub/pdb/validation_reports/a9/6a93 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 41931.051 Da / 分子数: 2 / 変異: S162K, M164W, R120I, H124I, R128G, M29W / 由来タイプ: 組換発現 由来: (組換発現)   Homo sapiens (ヒト), (組換発現) Homo sapiens (ヒト), (組換発現)   Escherichia coli (大腸菌) Escherichia coli (大腸菌)遺伝子: HTR2A, HTR2, cybC 発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: P28223, UniProt: P0ABE7 |
|---|
-非ポリマー , 5種, 9分子 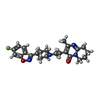








| #2: 化合物 |  リスペリドン リスペリドン#3: 化合物 |  コレステロール コレステロール#4: 化合物 | ChemComp-ZN / | #5: 化合物 |  ポリエチレングリコール ポリエチレングリコール#6: 化合物 | ChemComp-PLM / |  パルミチン酸 パルミチン酸 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.21 Å3/Da / 溶媒含有率: 61.62 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 脂質キュービック相法 詳細: 100 mM MES, pH 6.0-6.5, 28-31% (v/v) PEG 400, 120-200 mM Am-formate, 1-2% (v/v) DMSO PH範囲: 6.0-6.5 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SPring-8 SPring-8  / ビームライン: BL32XU / 波長: 1 Å / ビームライン: BL32XU / 波長: 1 Å |
| 検出器 | タイプ: RAYONIX MX225-HS / 検出器: CCD / 日付: 2016年12月15日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.707→46.241 Å / Num. obs: 29971 / % possible obs: 83.96 % / 冗長度: 22.4 % / CC1/2: 0.993 / Net I/σ(I): 6.63 |
| 反射 シェル | 解像度: 2.707→2.8 Å / CC1/2: 0.677 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 2RH1 解像度: 3→46.241 Å / SU ML: 0.39 / 交差検証法: FREE R-VALUE / σ(F): 2.03 / 位相誤差: 26.96
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3→46.241 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj


















