+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6wh4 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of HTR2A with inverse agonist | |||||||||
 Components Components | 5-hydroxytryptamine receptor 2A,Soluble cytochrome b562 fusion | |||||||||
 Keywords Keywords | MEMBRANE PROTEIN / HTR2A | |||||||||
| Function / homology |  Function and homology information Function and homology informationprotein localization to cytoskeleton / positive regulation of heat generation / 1-(4-iodo-2,5-dimethoxyphenyl)propan-2-amine binding / Gq/11-coupled serotonin receptor activity / positive regulation of phosphatidylinositol biosynthetic process / G protein-coupled serotonin receptor signaling pathway / G protein-coupled serotonin receptor complex / neurofilament / cell body fiber / phospholipase C-activating serotonin receptor signaling pathway ...protein localization to cytoskeleton / positive regulation of heat generation / 1-(4-iodo-2,5-dimethoxyphenyl)propan-2-amine binding / Gq/11-coupled serotonin receptor activity / positive regulation of phosphatidylinositol biosynthetic process / G protein-coupled serotonin receptor signaling pathway / G protein-coupled serotonin receptor complex / neurofilament / cell body fiber / phospholipase C-activating serotonin receptor signaling pathway / artery smooth muscle contraction / positive regulation of cytokine production involved in immune response / Serotonin receptors / serotonin receptor activity / G protein-coupled serotonin receptor activity / sensitization / serotonin receptor signaling pathway / urinary bladder smooth muscle contraction / neurotransmitter receptor activity / serotonin binding / positive regulation of platelet aggregation / negative regulation of synaptic transmission, glutamatergic / positive regulation of DNA biosynthetic process / temperature homeostasis / positive regulation of vasoconstriction / detection of temperature stimulus involved in sensory perception of pain / regulation of dopamine secretion / negative regulation of potassium ion transport / protein tyrosine kinase activator activity / positive regulation of execution phase of apoptosis / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / detection of mechanical stimulus involved in sensory perception of pain / positive regulation of fat cell differentiation / behavioral response to cocaine / release of sequestered calcium ion into cytosol / presynaptic modulation of chemical synaptic transmission / positive regulation of glycolytic process / dendritic shaft / glycolytic process / electron transport chain / caveola / memory / intracellular calcium ion homeostasis / positive regulation of inflammatory response / positive regulation of neuron apoptotic process / positive regulation of cytosolic calcium ion concentration / presynaptic membrane / virus receptor activity / cytoplasmic vesicle / G alpha (q) signalling events / chemical synaptic transmission / postsynaptic membrane / electron transfer activity / periplasmic space / positive regulation of ERK1 and ERK2 cascade / iron ion binding / response to xenobiotic stimulus / axon / neuronal cell body / positive regulation of cell population proliferation / heme binding / dendrite / protein-containing complex binding / glutamatergic synapse / identical protein binding / plasma membrane Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 3.4 Å MOLECULAR REPLACEMENT / Resolution: 3.4 Å | |||||||||
 Authors Authors | Kim, K.L. / Che, T. / Krumm, B.E. / Roth, B.L. | |||||||||
| Funding support |  United States, 2items United States, 2items
| |||||||||
 Citation Citation |  Journal: Cell(Cambridge,Mass.) / Year: 2020 Journal: Cell(Cambridge,Mass.) / Year: 2020Title: Structure of a Hallucinogen-Activated Gq-Coupled 5-HT 2A Serotonin Receptor Authors: Kim, K.L. / Che, T. / Panova, O. / DiBerto, J.F. / Lyu, J. / Krumm, B.E. / Wacker, D. / Robertson, M.J. / Seven, A.B. / Nichols, D.E. / Shoichet, B.K. / Skiniotis, G. / Roth, B.L. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6wh4.cif.gz 6wh4.cif.gz | 272 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6wh4.ent.gz pdb6wh4.ent.gz | 172.3 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6wh4.json.gz 6wh4.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/wh/6wh4 https://data.pdbj.org/pub/pdb/validation_reports/wh/6wh4 ftp://data.pdbj.org/pub/pdb/validation_reports/wh/6wh4 ftp://data.pdbj.org/pub/pdb/validation_reports/wh/6wh4 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6wgtC  5tvnS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 
| ||||||||||||
| 3 | 
| ||||||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 3 molecules ABC
| #1: Protein | Mass: 50534.918 Da / Num. of mol.: 3 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human), (gene. exp.) Homo sapiens (human), (gene. exp.)  Gene: HTR2A, HTR2, cybC / Production host:  |
|---|
-Non-polymers , 6 types, 15 molecules 



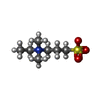






| #2: Chemical | | #3: Chemical | ChemComp-OLA / #4: Chemical | #5: Chemical | ChemComp-PEG / #6: Chemical | ChemComp-NDS / | #7: Chemical | ChemComp-1PE / | |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.79 Å3/Da / Density % sol: 67.55 % |
|---|---|
| Crystal grow | Temperature: 293.15 K / Method: lipidic cubic phase / pH: 7 Details: 100 mM Tris (pH7.0) 380 mM potassium phosphate-monobasic 33% PEG 400 100 mM Guanidine HCL 300 mM NDSB-195 |
-Data collection
| Diffraction | Mean temperature: 80 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 23-ID-B / Wavelength: 1.03317 Å / Beamline: 23-ID-B / Wavelength: 1.03317 Å |
| Detector | Type: DECTRIS EIGER X 16M / Detector: PIXEL / Date: Mar 10, 2019 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.03317 Å / Relative weight: 1 |
| Reflection | Resolution: 3→34.78 Å / Num. obs: 41269 / % possible obs: 99.2 % / Redundancy: 5.2 % / Biso Wilson estimate: 65.18 Å2 / CC1/2: 0.994 / Net I/σ(I): 4.2 |
| Reflection shell | Resolution: 3→3.12 Å / Num. unique obs: 4468 / CC1/2: 0.306 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 5TVN Resolution: 3.4→33.5 Å / SU ML: 0.4708 / Cross valid method: FREE R-VALUE / σ(F): 1.37 / Phase error: 30.5739 Stereochemistry target values: GeoStd + Monomer Library + CDL v1.2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 48.64 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 3.4→33.5 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
|
 Movie
Movie Controller
Controller













 PDBj
PDBj




















