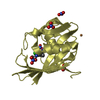
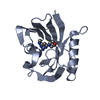
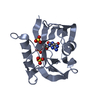
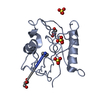
登録情報 データベース : PDB / ID : 5hzxタイトル Crystal structure of zebrafish MTH1 in complex with TH588 Nudix (Nucleoside diphosphate linked moiety X)-type motif 1 キーワード / / / / 機能・相同性 生物種 Danio rerio (ゼブラフィッシュ)手法 / / / 解像度 : 1.9 Å データ登録者 Narwal, M. / Gustafsson, R. / Brautigam, L. / Pudelko, L. / Jemth, A.-S. / Gad, H. / Karsten, S. / Carreras-Puigvert, J. / Homan, E. / Berndt, C. ...Narwal, M. / Gustafsson, R. / Brautigam, L. / Pudelko, L. / Jemth, A.-S. / Gad, H. / Karsten, S. / Carreras-Puigvert, J. / Homan, E. / Berndt, C. / Berglund, U.W. / Helleday, T. / Stenmark, P. 資金援助 組織 認可番号 国 Swedish Society for Medical research Karolinska Institute KID funding German Research Foundation Knut and Alice Wallenberg Foundation Swedish Foundation for Strategic Research Swedish Cancer Society Swedish Research Council Wenner-Gren Foundation Goran Gustafsson Foundation Swedish Children's Cancer Foundation Swedish Pain Relief Foundation Torsten and Ragnar Soderberg Foundation
ジャーナル : Cancer Res. / 年 : 2016タイトル : Hypoxic Signaling and the Cellular Redox Tumor Environment Determine Sensitivity to MTH1 Inhibition.著者 : Brautigam, L. / Pudelko, L. / Jemth, A.S. / Gad, H. / Narwal, M. / Gustafsson, R. / Karsten, S. / Carreras Puigvert, J. / Homan, E. / Berndt, C. / Berglund, U.W. / Stenmark, P. / Helleday, T. 履歴 登録 2016年2月3日 登録サイト / 処理サイト 改定 1.0 2016年2月10日 Provider / タイプ 改定 1.1 2016年2月24日 Group 改定 1.2 2016年6月1日 Group 改定 1.3 2017年9月6日 Group / カテゴリ / Item 改定 1.4 2024年1月10日 Group / Database references / Refinement descriptionカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_ncs_dom_lim Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ncs_dom_lim.beg_auth_comp_id / _struct_ncs_dom_lim.beg_label_asym_id / _struct_ncs_dom_lim.beg_label_comp_id / _struct_ncs_dom_lim.beg_label_seq_id / _struct_ncs_dom_lim.end_auth_comp_id / _struct_ncs_dom_lim.end_label_asym_id / _struct_ncs_dom_lim.end_label_comp_id / _struct_ncs_dom_lim.end_label_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード HYDROLASE (加水分解酵素) / Inhibitor /
HYDROLASE (加水分解酵素) / Inhibitor /  Complex /
Complex /  MTH1 / TH588
MTH1 / TH588 機能・相同性情報
機能・相同性情報 2-hydroxy-dATP diphosphatase / 8-oxo-7,8-dihydroguanosine triphosphate pyrophosphatase activity / hydrolase activity, acting on acid anhydrides, in phosphorus-containing anhydrides / DNA protection /
2-hydroxy-dATP diphosphatase / 8-oxo-7,8-dihydroguanosine triphosphate pyrophosphatase activity / hydrolase activity, acting on acid anhydrides, in phosphorus-containing anhydrides / DNA protection /  phosphatase activity /
phosphatase activity /  加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 /
加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 /  ミトコンドリアマトリックス ...purine deoxyribonucleotide metabolic process / Phosphate bond hydrolysis by NUDT proteins / nucleoside triphosphate diphosphatase activity /
ミトコンドリアマトリックス ...purine deoxyribonucleotide metabolic process / Phosphate bond hydrolysis by NUDT proteins / nucleoside triphosphate diphosphatase activity /  2-hydroxy-dATP diphosphatase / 8-oxo-7,8-dihydroguanosine triphosphate pyrophosphatase activity / hydrolase activity, acting on acid anhydrides, in phosphorus-containing anhydrides / DNA protection /
2-hydroxy-dATP diphosphatase / 8-oxo-7,8-dihydroguanosine triphosphate pyrophosphatase activity / hydrolase activity, acting on acid anhydrides, in phosphorus-containing anhydrides / DNA protection /  phosphatase activity /
phosphatase activity /  加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 /
加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 /  ミトコンドリアマトリックス /
ミトコンドリアマトリックス /  RNA binding /
RNA binding /  metal ion binding /
metal ion binding /  細胞核 /
細胞核 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Danio rerio (ゼブラフィッシュ)
Danio rerio (ゼブラフィッシュ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 スウェーデン,
スウェーデン,  ドイツ, 12件
ドイツ, 12件  引用
引用 ジャーナル: Cancer Res. / 年: 2016
ジャーナル: Cancer Res. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5hzx.cif.gz
5hzx.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5hzx.ent.gz
pdb5hzx.ent.gz PDB形式
PDB形式 5hzx.json.gz
5hzx.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/hz/5hzx
https://data.pdbj.org/pub/pdb/validation_reports/hz/5hzx ftp://data.pdbj.org/pub/pdb/validation_reports/hz/5hzx
ftp://data.pdbj.org/pub/pdb/validation_reports/hz/5hzx
 リンク
リンク 集合体
集合体


 要素
要素
 Danio rerio (ゼブラフィッシュ)
Danio rerio (ゼブラフィッシュ)
 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q7ZWC3
Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q7ZWC3










 硫酸塩
硫酸塩 グリセリン
グリセリン ジエチレングリコール
ジエチレングリコール 酢酸塩
酢酸塩 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  BESSY
BESSY  / ビームライン: 14.1 / 波長: 0.91991 Å
/ ビームライン: 14.1 / 波長: 0.91991 Å : 0.91991 Å / 相対比: 1
: 0.91991 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj





